Ribonuclease R (RNase R)是一种3’-5’方向的RNA外切酶,主要应用于circRNA的鉴定和富集,今天湖人再和大家分享一下具体使用及常见问题。
1 RNase R反应体系
相关文章方法描述里RNase R的使用量和反应体系颇有不同,从1U-4U RNase R/μg RNA不等,下图是一个推荐的反应体系。
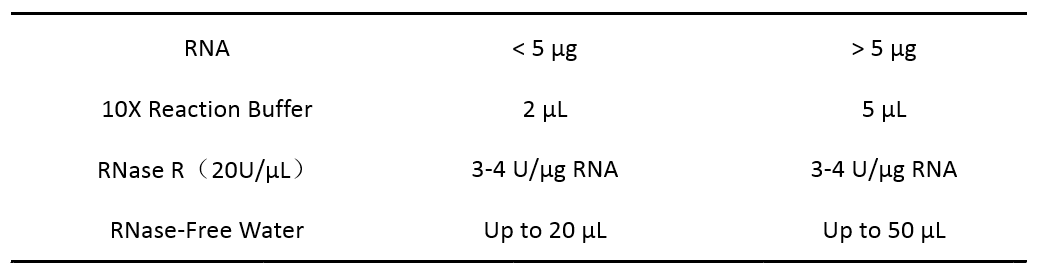
图1 RNase R消化反应体系(吉赛生物)
2 circRNA的鉴定
circRNA的鉴定或验证常使用RT-PCR和Northern blot实验,根据RNase R(+)和RNase R(-)组中是否检测到条带来证明检测的分子是circRNA。
RT-PCR中若要证明检测的基因是circRNA,需要体现在RNase R(+)和RNase R(-)组中条带有无或丰度高低有差异,也可以和分别使用Divergent Primer和Convergent Primer检测cDNA和gDNA样品的结果结合起来。
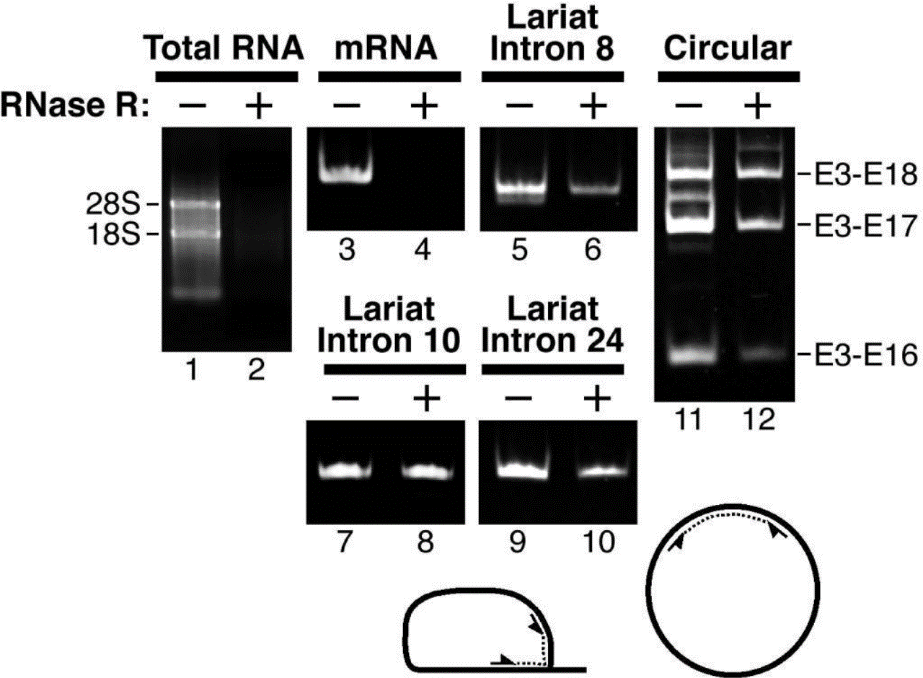
图2 使用RT-PCR检测mRNA,在RNase R(-)中有条带,在RNase R(+)中无条带;检测Lariat/Circular RNA,在RNase R(-)和RNase R(+)样品中都有条带,表明mRNA被消化,而Lariat/Circular RNA耐受消化(Suzuki H et al., 2006)
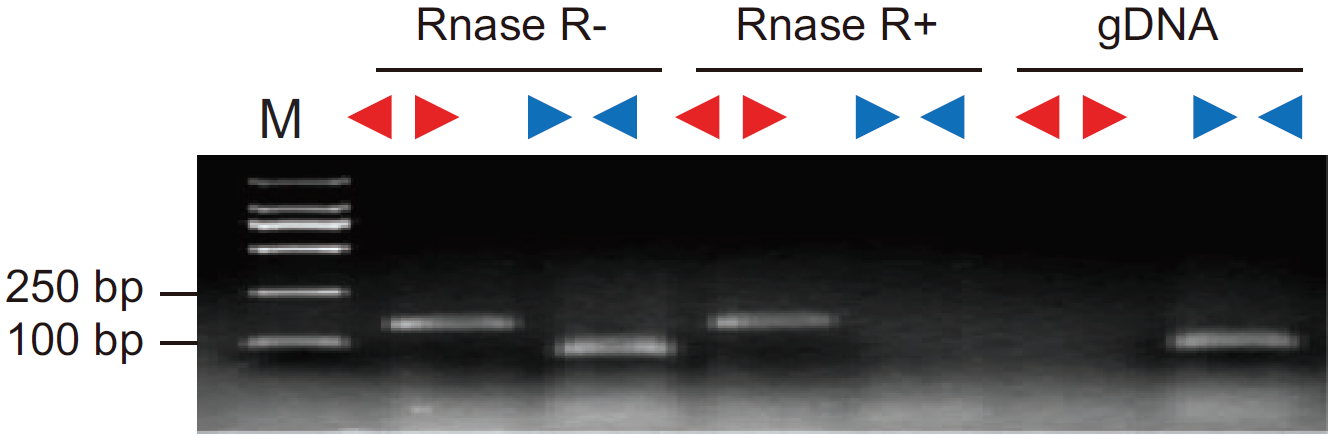
图3 RNase R消化后分别以Divergent Primer和Convergent Primer检测cDNA样品。RNase R(+)中Divergent Primer有条带,Convergent Primer无条带;RNase R(-)中Divergent Primer和Convergent Primer都有条带,表明检测的基因为circRNA,并且耐受RNase R消化(Yibing Y et al., 2018)
Northern blot实验使用探针特异性检测内源的circRNA和线性RNA,过程中无PCR扩增,能够真实地反映circRNA和线性RNA的定性定量分析结果,是circRNA鉴定中必不可少的一个实验。
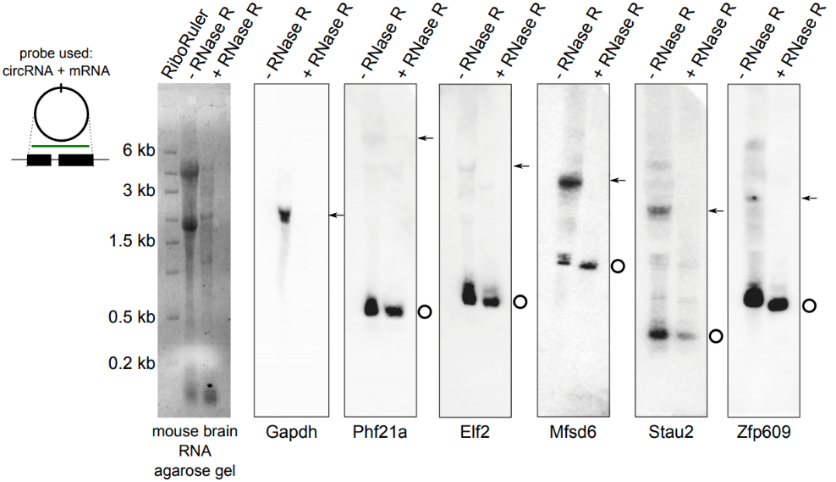
图4 以3U RNase R/µg RNA 进行RNase R消化15分钟,电泳显示RNase R(+)组中28S/18S条带变淡。使用同时检测circRNA和mRNA的探针进行Northern blot实验,结果显示RNase R(+)组检测不到线性的Gapdh、Phf21a、Elf2、Mfsd6、Stau2或Zfp609,可以检测到对应的circRNA,表明mRNA被消化,而circRNA耐受消化(Rybak-Wolf, A. et al., 2015)
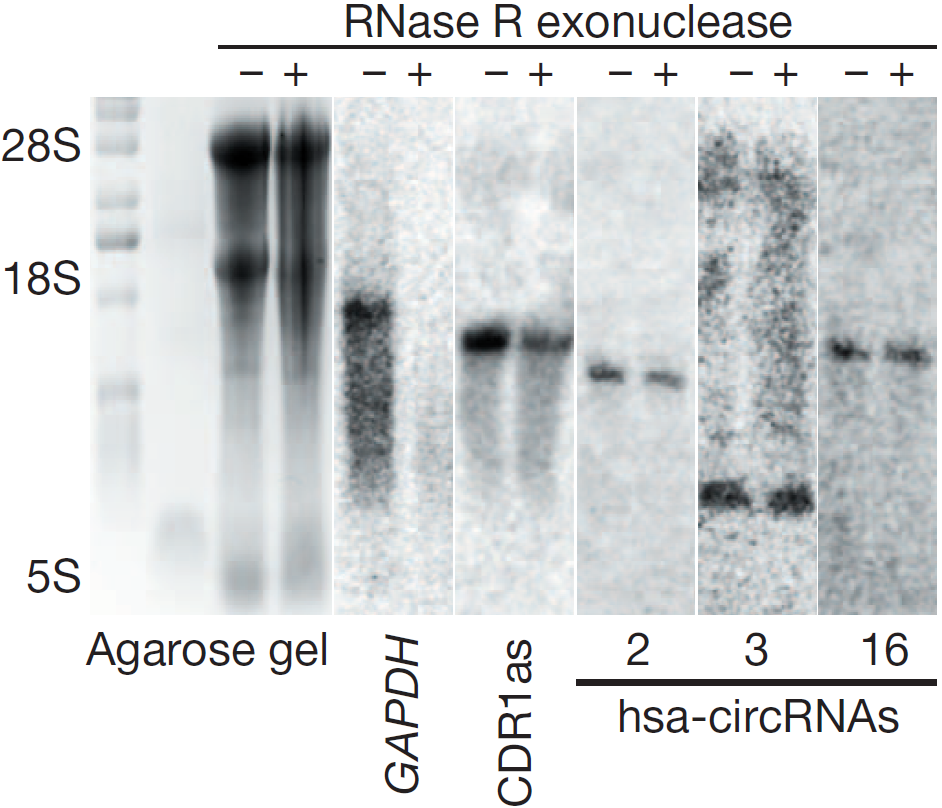
图5 RNase R消化产物电泳显示RNase R(+)组中28S/18S/5S条带变淡。Northern blot检测显示RNase R(+)组中无线性GAPDH的条带,CDR1as和hsa-circRNA 2/3/16能检测到条带,表明线性RNA被消化,而circRNA耐受消化(Memczak S et al., 2013)
3 circRNA的富集
高通量测序时常需要使用RNase R对circRNA进行富集,为探究RNase R消化后circRNA的富集程度和相关基因的变化,可以同时做RNase R(+)和RNase R(-)组样品的测序。测序显示RNase R(+)组中junction reads相对于RNase R(-)样本一般有5-10倍的富集,可鉴定出几千到上万个circRNAs。
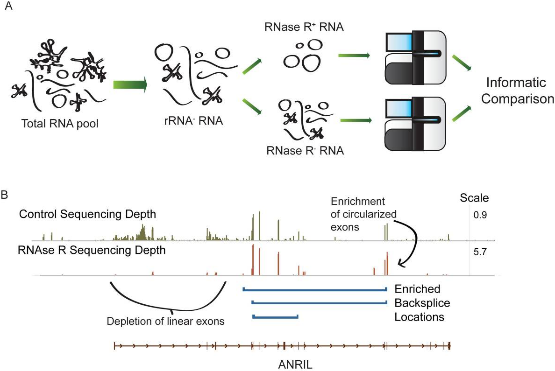
图6 RNase R(+)和RNase R(-) RNA测序示意图(Jeck WR et al., 2014)
4 RNase R消化实验结果
Total RNA经消化后直接进行电泳检测,RNase R(-)组中28S/18S/5S三条带单一明亮,而RNase R(+)组中28S/18S/5S条带会变淡或不可见(Rybak-Wolf, A. et al., 2015; Suzuki H et al., 2006)。但如果RNase R+组中5S条带加亮,且28S/18S条带处有拖尾,则可能是RNA被外源RNA酶降解,而不是RNase R消化产生的。
a 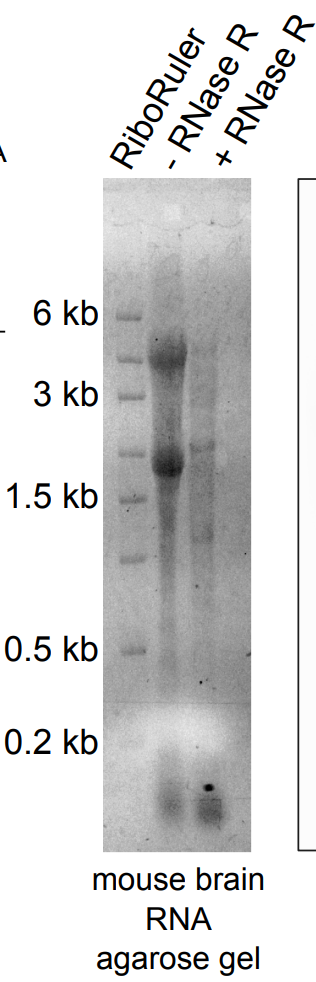 b
b 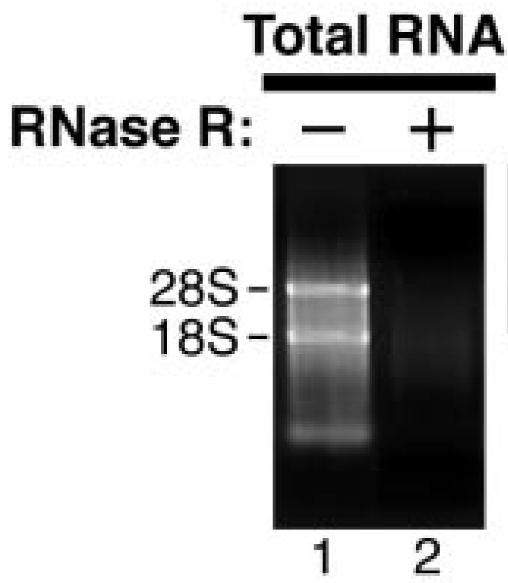
图7 a, RNase R消化后条带变淡;b, RNase R消化后条带不可见(Rybak-Wolf, A. et al., 2015; Suzuki H et al., 2006)
5 Q&As
5.1 测量浓度
实验中要先对total RNA测量浓度,以计算相应的比例和使用量来进行RNase R消化。消化后的反应液无需测量浓度,实际上因为反应体系内的蛋白质、盐离子等成分影响,测量到的浓度也不会准确。
5.2 纯化回收
经RNase R消化后富集的circRNA可使用苯酚:氯仿:异戊醇溶液、RNA纯化柱或磁珠进行纯化回收,没条件的也可以直接使用TRIzol LS Reagent。经RNase R消化后直接进行RT-PCR的,一般可以不做纯化,酶失活后直接进行逆转录反应即可(Legnini I et al., 2017; Guarnerio J et al., 2016; Cheng ZF et al., 2002)。
5.3 RNase R消化反应温度和时间
RNase R消化一般于37℃进行反应,时间10-30 min为宜(Memczak S et al., 2013; Legnini I et al., 2017),不推荐过长时间的消化。另外检测不同的基因可能需要摸索反应时间,湖人实验室直接用于RT-PCR检测时常用5-15 min消化就能得到很好的结果,线性基因丰度可以降低几十到几百倍,circRNA丰度则基本不变。
5.4 qPCR定量计算
RNase R消化后不能再使用ACTB或GAPDH作为内参,此时可以将原始的RNA等分为两份,一份进行RNase R处理(RNase R(+)),另一份不处理(RNase R(-)),统一以RNase R(-)组中的内参为计算标准(Zhang Y et al., 2016; Panda AC et al., 2018)。
另外在RNase R消化后如果进行纯化回收,RNA的浓度可能会有变化,不适宜再以RNase R(-)组中的内参为计算标准。此时可以在纯化回收前加入少量其他物种的RNA作为外参,统一以外参标准化样品后进行计算(Rybak-Wolf, A. et al., 2015; Pamudurti NR et al., 2017)。
5.5 circRNA也被消化?
实验中有时会发现经RNase R消化后部分circRNA丰度也有明显降低,此时要检查消化反应体系有无问题,考虑是否被外源RNA酶降解。另外有些circRNA经长时间RNase R消化后丰度也会降低,可能是因为其耐受RNase R消化力弱(Zhang Y et al., 2016)。
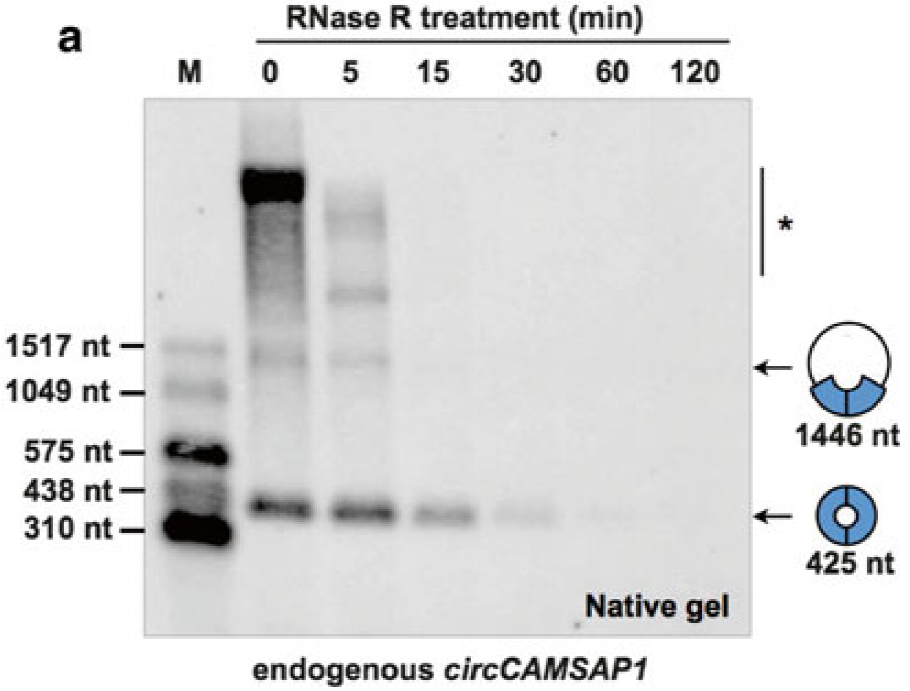
图8 RNase R消化0-120 min,Northern blot检测显示circCAMSAP1在15 min时就明显被消化(Zhang Y et al., 2016)
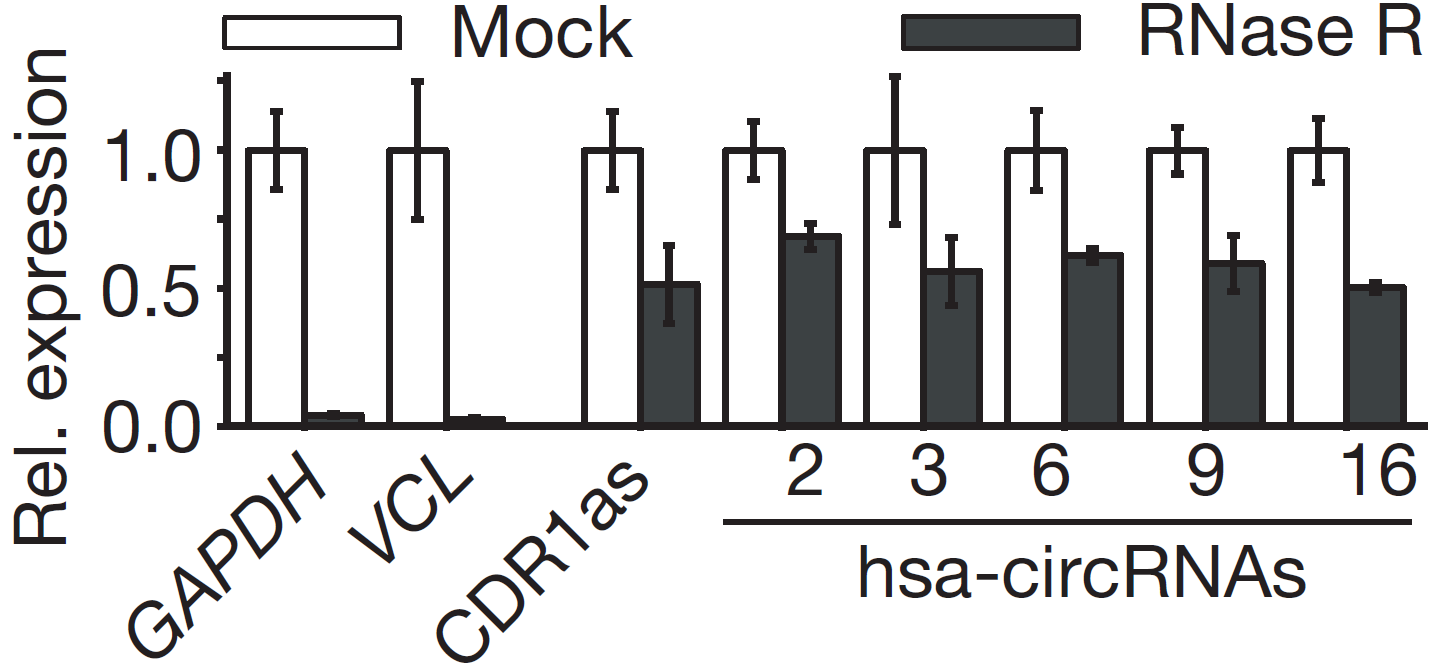
图9 RNase R消化后进行qPCR检测,RNase R组中has-circRNA 2/3/6/9/16和CDR1as丰度有轻微降低,但和GAPDH相比,circRNAs显示有超过10倍的耐受RNase R消化力(Memczak S et al., 2013)
湖人实验室也经常会检测到RNase R消化后circRNA丰度会降低的情况,一般缩短消化时间可以减少circRNA被消化的可能,另外对比线性RNA和circRNA消化后丰度降低的倍数,是明显能看到circRNA比线性RNA耐受消化的。
5.6 线性RNA消化不完全?
实验中经常发现经RNase R消化后做RT-PCR依然能检测到线性RNA的条带,总要怀疑是消化不够彻底或消化体系不合适。但其实这种情况是正常的,因为在限定RNase R用量和反应时间的条件下,底物中线性RNA很难被彻底消化,而增加RNase R用量或反应时间又可能导致部分circRNA被消化,因此在固定体系中平衡反应时间是很有必要的。另外注意以半定量PCR的思路,统一PCR扩增条件和循环次数,电泳检测也可能得到理想的结果图。如果RNase R消化后circRNA丰度不变或轻微降低(RT-PCR检测条带基本不变),而线性RNA丰度明显降低(RT-PCR检测条带变淡或不可见),这样的结果也是符合预期的。

图10 RNase R消化后进行PCR检测,与RNase R(-)组相比,RNase R(+)组中linear mRNA条带变淡,表明被消化;RNase R(+)组中ci-ankrd52和circCAMSAP1条带变亮,表明被富集(Zhang Y et al., 2016)
参考文献:
- Cheng ZF, Deutscher MP. J. Purification and characterization of the Escherichia coli exoribonuclease RNase R. Comparison with RNase II. Biol. Chem. 2002; 277:21624–21629.
- Rybak-Wolf, A. et al.. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed. Mol. Cell. 58, 870–885 (2015).
- Yibing Yang et al.. Novel Role of FBXW7 Circular RNA in Repressing Glioma Tumorigenesis. J Natl Cancer Inst. 2018 Mar 1;110(3).
- Suzuki H et al.. Characterization of RNase R-digested cellular RNA source that consists of lariat and circular RNAs from pre-mRNA splicing. Nucleic Acids Res. 2006; 34(8), e63.
- Memczak S, Jens M, Elefsinioti A, et al.. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 2013;495(7441):333–338.
- Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs. Nat Biotechnol. 2014; 32:453–61.
- Guarnerio J, Bezzi M, Jeong JC, Paffenholz SV, Berry K, Naldini MM et al.. Oncogenic Role of Fusion-circRNAs Derived from Cancer-Associated Chromosomal Translocations. Cell. 2016 Aug 11;166(4):1055-1056.
- Legnini I, Di Timoteo G, Rossi F, Morlando M, Briganti F, Sthandier O, Fatica A, Santini T, Andronache A, Wade M, et al.. Circ-ZNF609 Is a Circular RNA that Can Be Translated and Functions in Myogenesis. Mol Cell. 2017;66(1):22–37.e29.
- Zhang, Y., Yang, L., and Chen, L.L. (2016c). Characterization of Circular RNAs. Methods Mol. Biol. 1402, 215–227.
- Panda AC, Gorospe M. Detection and Analysis of Circular RNAs by RT-PCR. Bio Protoc. 2018 Mar 20;8(6). pii: e2775.
- Pamudurti NR, Bartok O, Jens M, Ashwal-Fluss R, Stottmeister C, Ruhe L et al.. Translation of CircRNAs. Mol Cell. 2017 Apr 6;66(1):9-21.e7.
.png)