环状RNA (circRNA)具有独特的共价单链闭环结构。高通量测序已经确定许多circRNAs在细胞中具有关键的生理和病理作用。在RCC的发展过程中,某些circRNAs起着不可或缺的作用。例如,cRAPGEF5通过调节miR-27a-3p/TXNIP通路抑制RCC增殖和转移,CircSDHC通过其对miR-127-3p的海绵效应促进RCC进展[1]。然而,RCC的诊断和治疗需要进一步解析circRNA在肾癌中的作用机制。

一、circNTNG1的筛选过程及在RCC中的特性以及低表达
CircNTNG1(circBase ID: hsa_ circ_0002286)来自1号染色体上的NTNG1基因,由外显子1反向剪接而成。Sanger测序证实了RCC细胞中存在CircNTNG1。背靠背引物验证/FISH定位/R酶耐受处理证明CircNTNG1的成环/定位胞质/稳定性。
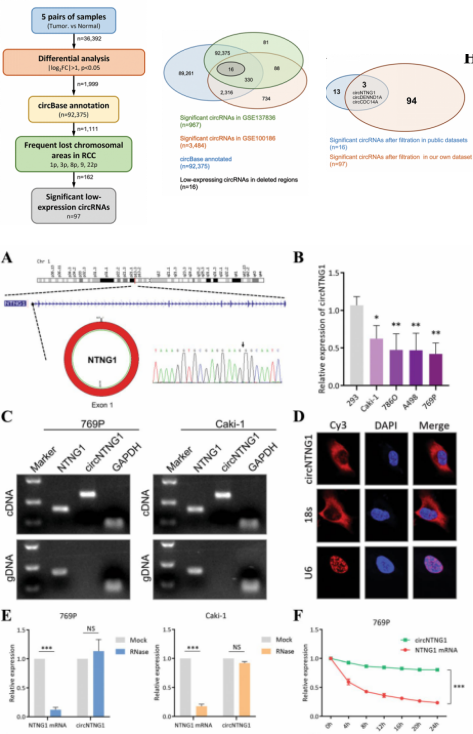
图1. circNTNG1的筛选过程及特性
二、CircNTNG1对miR-19b-3p产生海绵效应
miR-19b-3p在肿瘤组织中高表达,与低OS和RFS显著相关,其主要调控功能是离子结合和核酸结合。作者应用miR-19b-3p模拟物促进RCC细胞迁移和侵袭,而抑制剂则相反,这体现了miR-19b-3p的致癌能力。
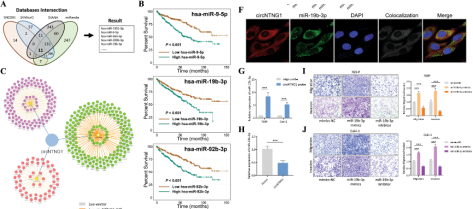
图2. CircNTNG1对miR-19b-3p产生海绵效应
三、miR-19b-3p通过抑制HOXA5的表达调控RCC的进展
在circNTNG1/miRNA/mRNA相互作用网络中,预测出20个下调最显著的mRNA中,只有NR3C2、PPARA、SOX6、SMARCA2、HOXA5和SATB1与核酸结合活性相关,这与miR-19b-3p的预测功能一致。应用miR-19b-3p模拟物的RCC细胞系769P进行体外验证后,HOXA5成为最有希望的下游靶点。双荧光素酶报告实验验证,miR-19b-3p与HOXA5结合。并且,miR-19b-3p mimic/inhibitor亦能抑制/促进HOXA5的表达。HOXA5是HOXA家族中独特的RCC抑癌基因。随后作者发现,过表达HOXA5可降低RCC细胞的迁移和侵袭能力,敲低则相反,但是无论过表达还是敲低,都不改变增殖能力。体外实验结果亦显示,在过表达HOXA5可减少肺定植,肺转移灶明显减少。
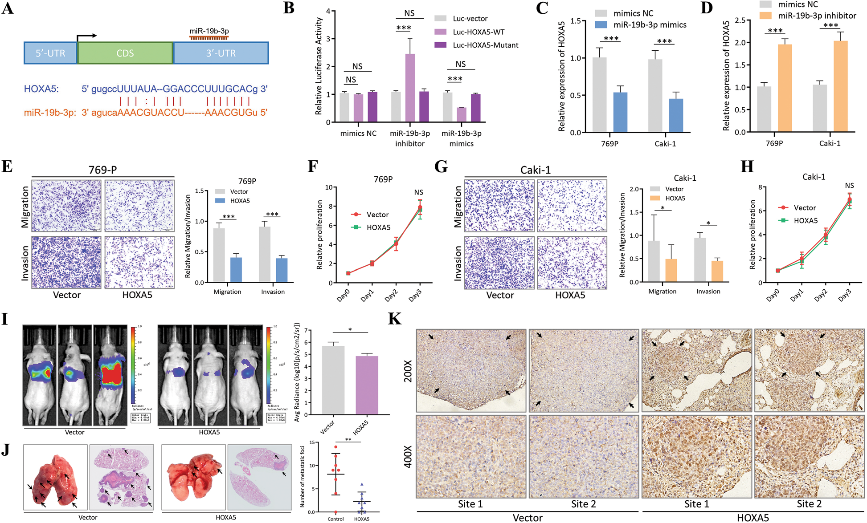
图3. miR-19b-3p通过抑制HOXA5的表达调控RCC的进展
四、HOXA5通过下调SNAI2 (slug)抑制上皮间充质转化(EMT)激活
然而,HOXA5如何抑制Slug?GO分析表示,HOXA5是一种典型的DNA结合蛋白,与转录活性相关。因此,HOXA5可能作为转录抑制因子来实现对Slug的抑制作用。结合JASPAR数据库和序列分析,作者发现Slug的启动子区域包含一个被HOXA5识别的特定序列。此外,参考肝癌HOXA5 ChIP-seq数据集(GSE170384),在Slug启动子区域发现了几个峰值。在此基础上,设计了针对Slug启动子区域的引物,HOXA5 ChIP-qPCR实验中该引物被显著富集。并且,在HOXA5过表达后,随着Slug的重新表达,蛋白质和细胞的迁移/侵袭能力也发生了相应的变化。这些结果证实了HOXA5与Slug的启动子区结合并抑制其表达,从而调节EMT。
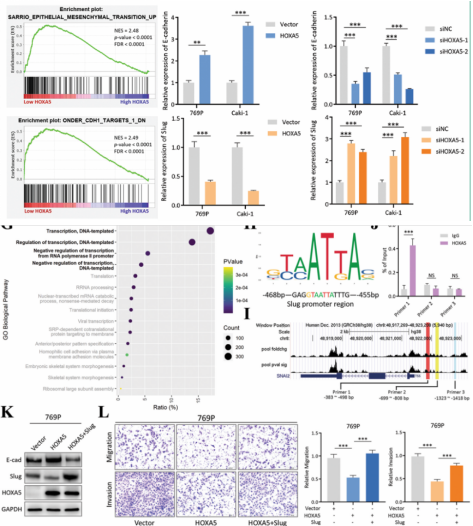
图4. HOXA5通过下调slug抑制EMT激活
五、HOXA5招募DNMT3A,增加Slug启动子区DNA甲基化水平
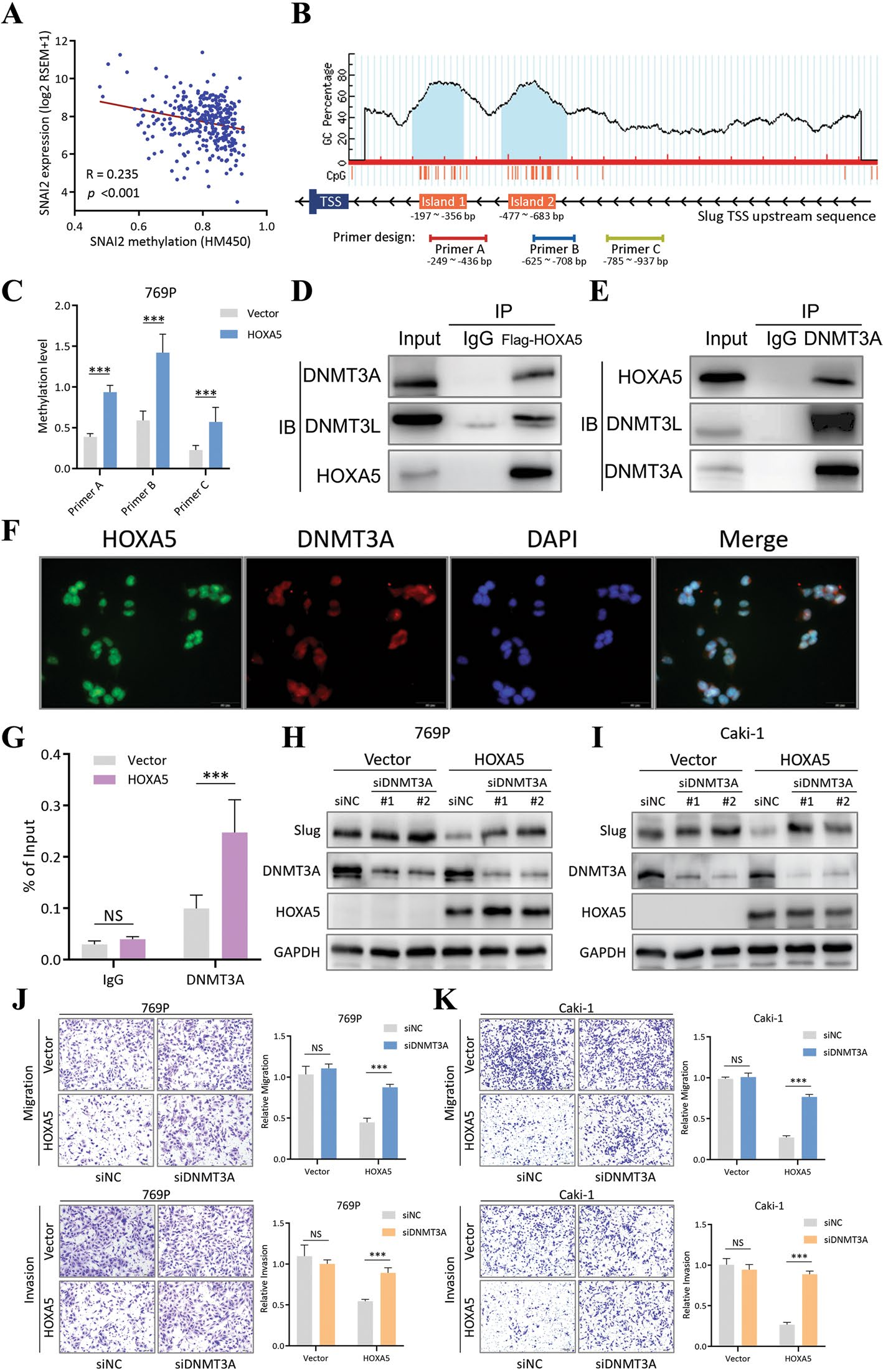
图5. HOXA5招募DNMT3A,增加Slug启动子区DNA甲基化水平
六、circNTNG1/miR-19b-3p/HOXA5调控轴的完整性
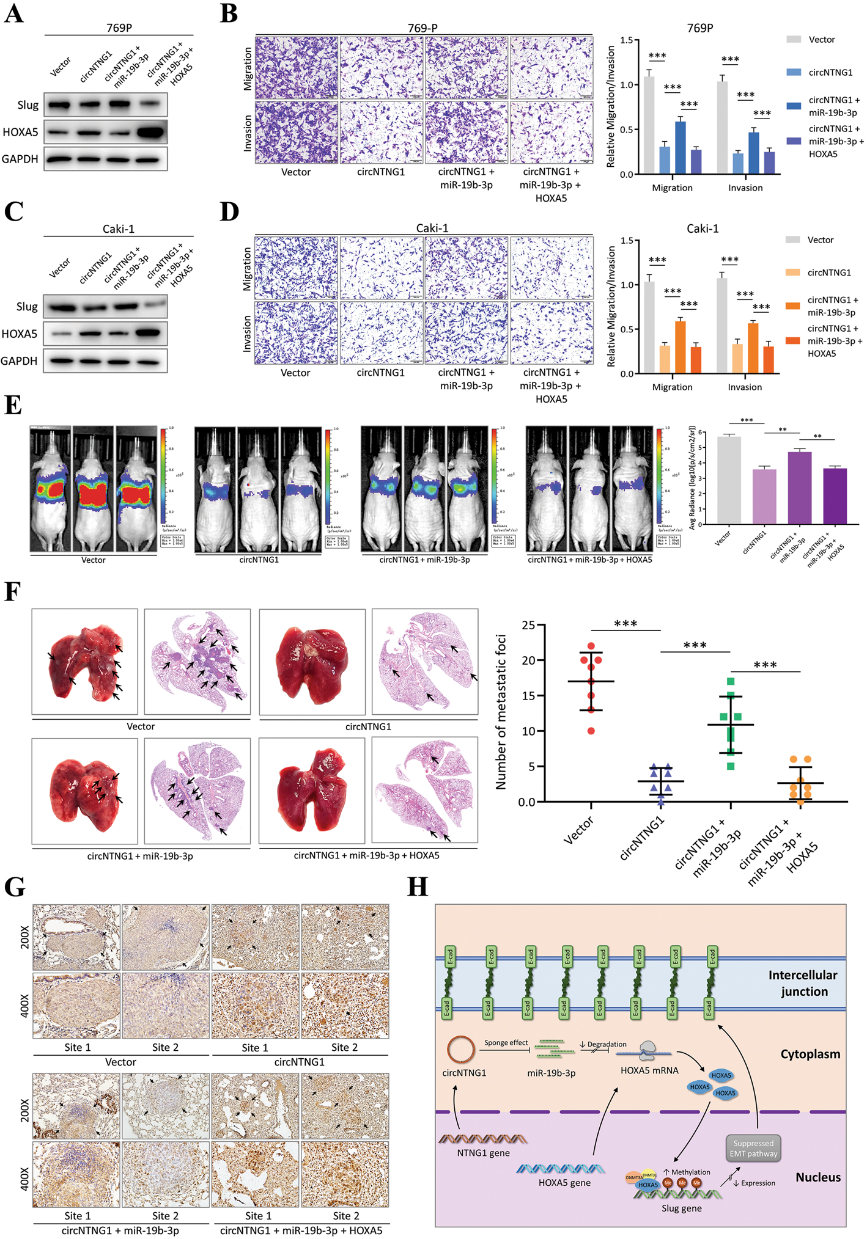
图6.circNTNG1/miR-19b-3p/HOXA5调控轴的完整性
https://doi.org/10.1186/s12943-022-01694-7
参考文献:
[1] Chen Q, et al. CircRNA cRAPGEF5 inhibits the growth and metastasis of renal cell carcinoma via the miR-27a-3p/TXNIP pathway. Cancer Lett. 2020; 469:68–77.
[2] Ribatti D, Tamma R, Annese T. Epithelial-mesenchymal transition in Cancer: a historical overview. Transl Oncol. 2020;13(6):100773.
[3] Jeltsch A. Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. Chembiochem. 2002;3(4):274–93.
.png)