4月29日,Briefings in Bioinformatics在线发表了一个全新的circRNA数据库平台,VirusCircBase,这是首个病毒circRNA的数据库资源([1])。文章的通讯作者是湖南大学彭友松教授。

VirusCircBase数据库首页
VirusCircBase数据库收录了病毒来源的11924种circRNA,涵盖了四大类23种病毒。数据库提供了数据统计,信息浏览,信息检索,miRNA互作分析结果等检索工具,还提供了免费数据下载。
VirusCircBase网址:http://www.computationalbiology.cn/ViruscircBase/home.html
图1 VirusCircBase数据库首页
数据库概况
VirusCircBase数据库的数据主要通过在线数据重分析获得,主要是GEO和SRA的数据,共整合分析了54个dataset的数据。利用三种常用的circRNA分析工具:CIRI2,find_circ,circRNA_finder。
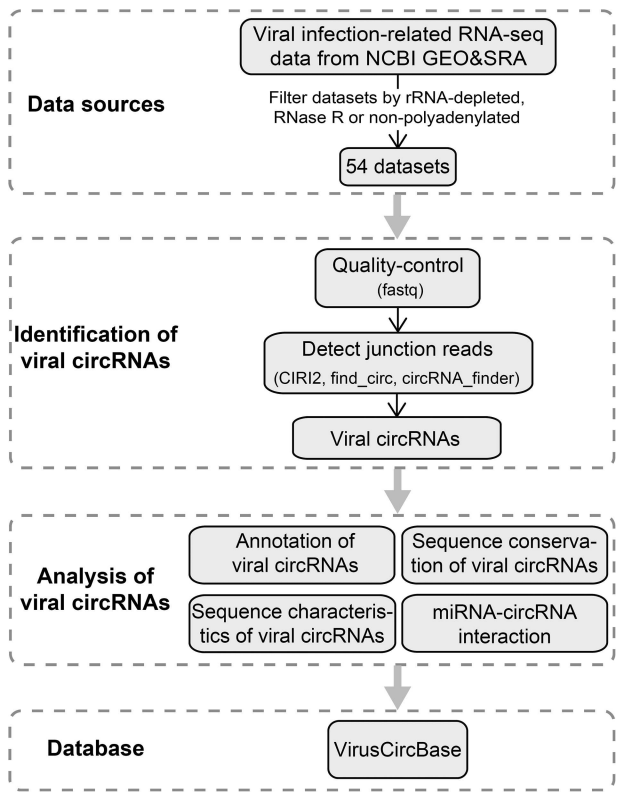
图2 VirusCircBase数据库数据分析流程([1])
三种工具共分析出病毒来源的circRNA 11924种,其中626种是三种工具均可分析到的。从病毒物种来看,双链DNA(dsDNA)病毒来源的circRNA占了约70%,其中人丙型疱疹病毒8 (HHV8)分析到4177种circRNA,人乙型疱疹病毒5 (HHV5)分析到2554种,人γ疱疹病毒4 (EBV)分析到1002种。
单链RNA(ssRNA)病毒分析到了2151种circRNA,其中信德比斯病毒(Sindbis virus)分析到了1771种circRNA,其他如小鼠诺如病毒(193种),寨卡病毒(91种),甲型流感病毒(48种),埃博拉病毒(44种)。
逆转录病毒也有circRNA发现,如HIV1就分析到了227种circRNA分子。
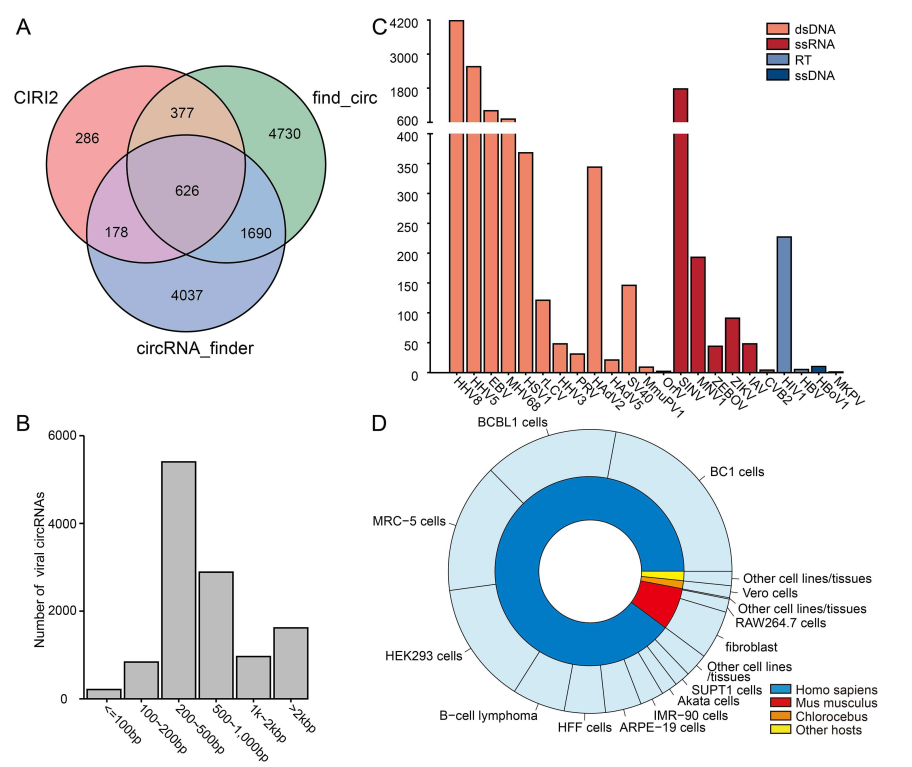
图3 VirusCircBase数据库数据总体情况([1])
病毒circRNA的基本特征
病毒来源circRNA长度总体在200~1000bp范围内,绝大部分的病毒circRNA来自dsDNA病毒和ssRNA病毒,两类病毒来源circRNA的平均长度分别是462 bp和439 bp。线性和环状基因组的病毒来源circRNA数目没有明显的差别。病毒circRNA的反向拼接位点在病毒基因组中的分布没有太明显的倾向性,大部分都是随机分布在病毒基因组的各个位置,但HHV8,HAdV2和MHV68中circRNA的反向拼接位点集中在个别位点。
为分析病毒circRNA可能的生成机制,作者挑选了丰度较高的1592种circRNA进行分析,绝大部分的circRNA的侧翼序列带有重复序列或反向互补序列。
保守性方面,绝大部分病毒circRNA都是物种特异性的序列,极少数circRNA在同种类的病毒中重复出现。
作者还预测分析了病毒circRNA与miRNA的互作情况,互作miRNA对应的通路进行了分析,相应的信息可以从VirusCircBase数据库检索。
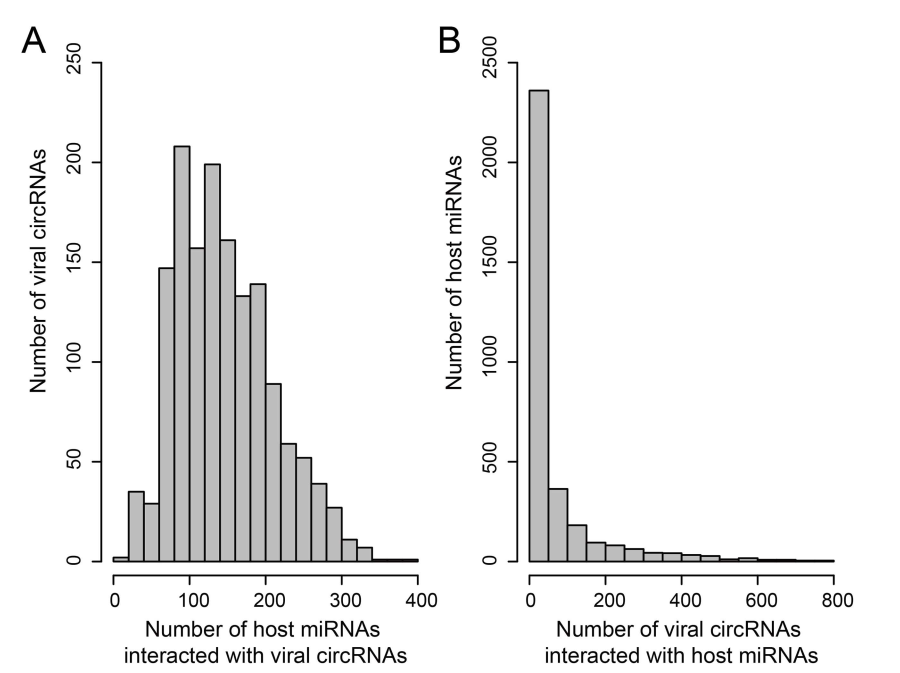
图4 病毒circRNA互作miRNA预测分析 ([1])
参考文献
1. Cai, Z., et al., VirusCircBase: a database of virus circular RNAs. Brief Bioinform, 2020.
.png)