最新的国自然获批项目表明早期 circRNA 粗犷型研究阶段已经过去,而深入单个 circRNA 或 circRNA 特征集的功能与作用机制的研究成为主流。
如何全面而深入地剖析 circRNA 分子?可能许多 circRNA 研究学者都是“朝思暮想”。而 David T. Humphreys 团队可能感应到了大家的号召,开发了一款全面分析 circRNA 分子的软件 Ularcirc,于 22日发表在了 Nucleic Acids Research 上。
Ularcirc 最大的亮点在于能够整合 forward-splicing 与 backward-splicing 事件,我们先盘点一下 Ularcirc 的功能。
Ularcirc 功能一览
1. 可视化界面
不用再为“秃顶”发愁,而且对于 circRNA 功能探索有直观感受。该软件是用 R 语言架构在 shiny 上的,所以个人电脑很适合。下图展示了它的欢迎界面
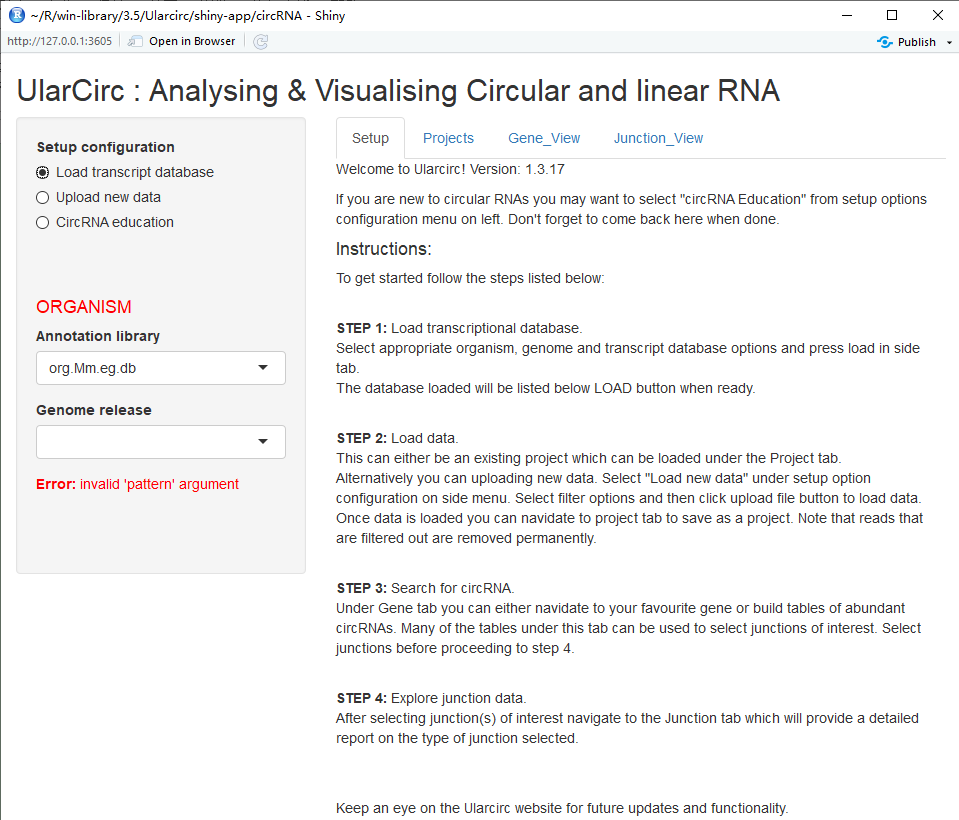
2. 从头检测 circRNA
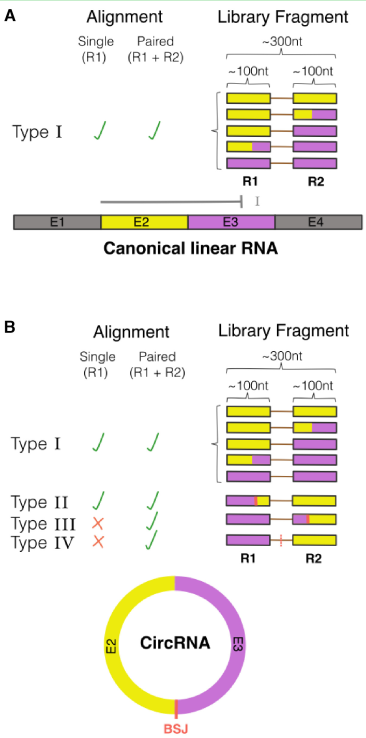
Ularcirc 能够从头(de novo)鉴定 circRNA。
A / B 分别展示了序列比对可能的结果,A 图为线性 RNA 的比对结果可能情况,而 B 图为 circRNA 的。而我们所检测的 BSJ 实际就是图 B 的 Type II / III,作为 circRNA 分子的候选。
另外,Ularcirc 定义了两个参数 RAD 与 FSJ support score 来评估 circRNA 真实性。
3. 环状及线性转录可视化
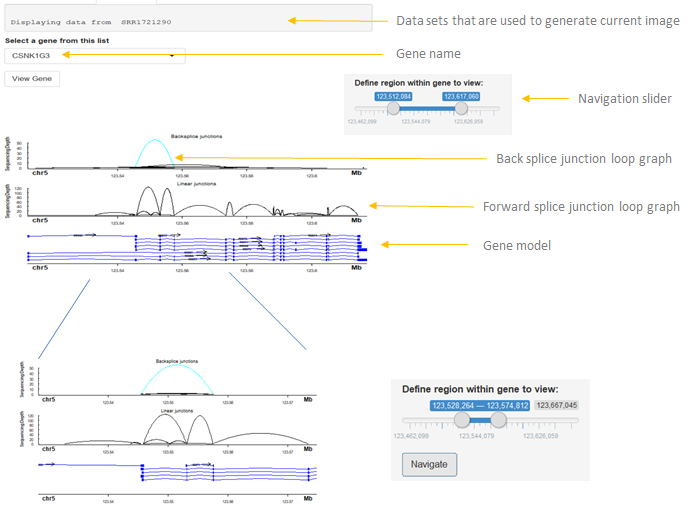
通过 Ularcirc,我们可以了解某个基因的 circRNA 以及线性 RNA 转录情况。
4. ORF 识别
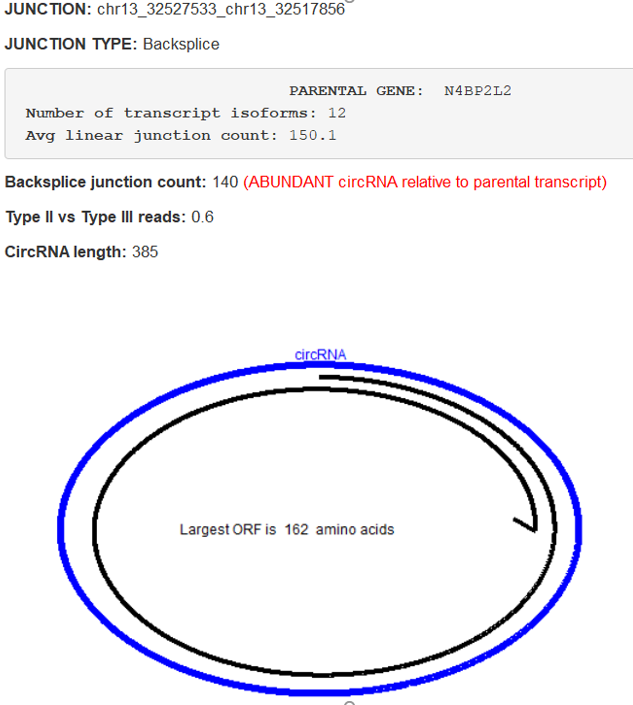
从图中我们可以获得很多 circRNA 的信息,并且能够观察到该 circRNA 的 ORF 的长度是要大于 circRNA 本身的长度。
5. miRNA 结合位点识别
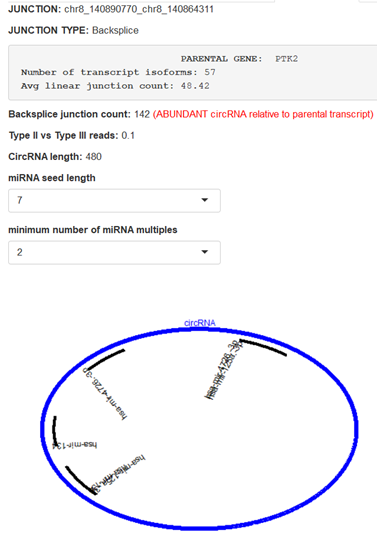
Ularcirc 能够识别 circRNA 序列上的 miRNA 结合位点并可视化,但可视化图并不好看。
6. circRNA 与线性 RNA 表达模式的可视化
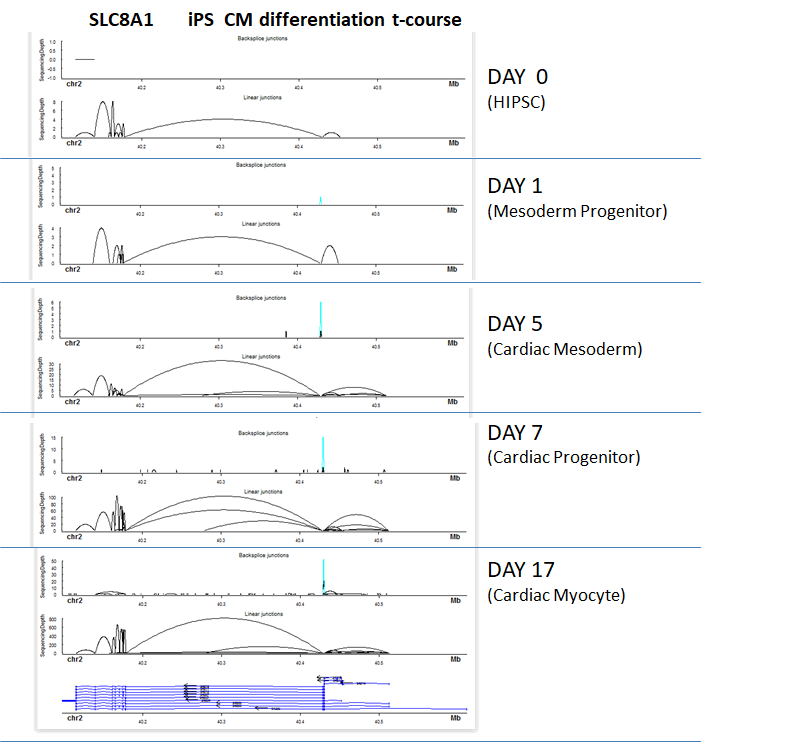
图中展示了 SLC8A1 这个基因在心肌细胞分化过程中 BSJ 以及 FSJ 的表达模式,每幅图上面表示 BSJ,下面表示 FSJ;其中连线表示 splicing junction,而连线的高低表示表达丰度的高低。这里的 BSJ 被用蓝色高亮了。
从图中我们可以看出,SLC8A1 在心肌分化过程中存在表达了多种多样的线性转录本,既有保守的也有特异表达的,另外还有新的转录本生成;而 circRNA 是发生在二号外显子上,表达是逐渐升高并在心肌细胞中稳定高表达。
7. circRNA 生物合成
我们已经观察到 SLC8A1 二号外显子形成 circRNA 为经典的剪切模式,即外显子已经有注释;除此之外,Ularcirc 还能观察到非经典的剪切模式,如下图 ApoA4 基因
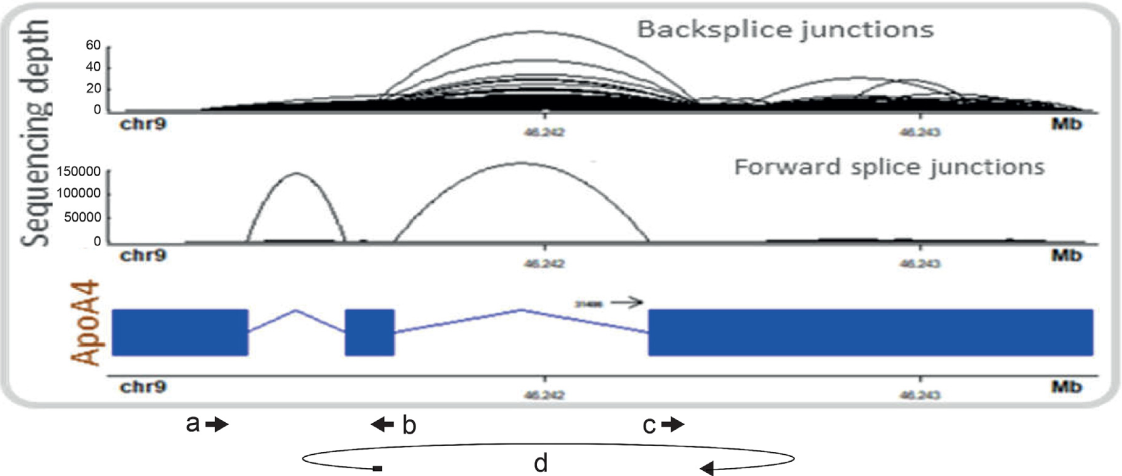
我们可以看到,ApoA4 的 circRNA 的 BSJ 起始与终止位点落在了第二和第三个外显子的内部而不是经典的剪切位点处。
Ularcirc 使用及下载
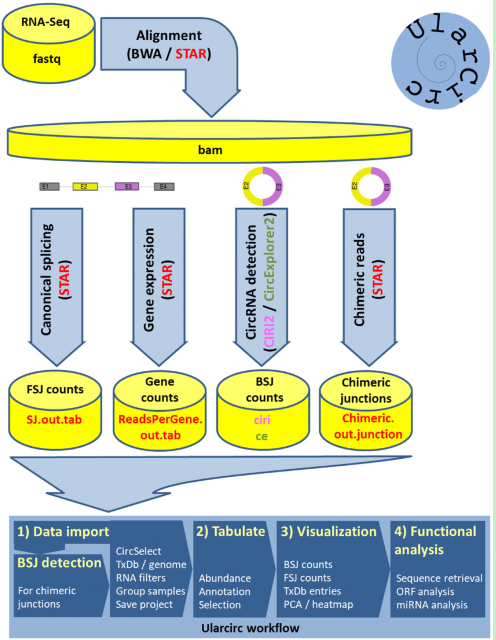
1. 输入
上图给出了 Ularcirc 的流程图,可以看出这款软件是主要依赖于 STAR 的比对结果
- 线性 RNA 需要 SJ.out.tab 与 counts 文件
- circRNA 需要 Chimeric.out.junciton 文件
- 另外,也支持 CIRI2 与 CIRCexplorer2 的结果作为输入
2. 输出
前面功能简介已经大概介绍了 Ularcirc 的结果,在流程图中我们可以看到,Ularcirc 还能提供 PCA 与 heatmap 的图,另外还能进行序列提取。
3. 安装与使用说明
该软件位于 github VCCRI/Ularcirc,其中展示了如何下载与安装该软件。
作者提供了视频教程,然而遗憾的是,我们无法访问 YouToBe。
当然,软件打开之后有一个简单的说明。
4. 使用初体验
安装完 Ularcirc 之后,我尝试了一下作者提供的内置文件,然而不知道为何无法加载基因组注释信息,后面的功能似乎也无法进一步体验。
虽然是可视化软件,但是很多东西还是摸不着头脑,需要进一步探索……
参考文献:
1.Humphreys D T, Fossat N, Tam P P L, et al. Ularcirc: Visualisation and enhanced analysis of circular RNAs via back and canonical forward splicing[J]. bioRxiv, 2018: 318436.
.png)