12月11日,Genome Biology(影响因子13.2)杂志在线发表了吉林大学附属第一医院胡继繁和崔久嵬为共同通讯作者的文章,介绍发现来自FLI1基因的circRNA能参与调控基因启动子的甲基化修饰[1]。

与常规的circRNA研究论文不同,本文作者一开始是希望探索FLI1基因在乳腺癌中表达增高的机制,从FLI1基因的启动子结合分子入手。作者采用了一种基于dCas9(没有内切酶活性的Cas9突变体,依然具有基于gRNA的精确基因组定位功能)的免疫共沉淀分离技术(CasIP),在所钓取的分子中意外发现了来自FLI1基因的由外显子2-4形成的circRNA(作者将其命名为FECR1),作者发现过表达FECR1能促进细胞侵袭。进一步,作者通过RAT分析(基于反转录的分子捕获分析),作者发现了在乳腺癌中受FECR1调控的下游基因,这些基因主要与细胞增殖调控有关。FECR1在FLI1基因的结合位点主要是启动子区的CpG岛,FECR1促进该区域的去甲基化。有趣的是,DNMT1基因也是FECR1的下游基因,但FECR1主要是抑制DNMT1的表达,与此同时,作者还发现TET1也能结合FECR1。这些发现表明FECR1参与了基因的表观遗传调控[1]。本文的故事与前不久单革教授发现的携带内含子序列的EIciRNA促进母基因转录的故事有些类似,但机制模型并不相同。本文的机制模型丰富了我们对circRNA作用机理的认知,值得各位同行学习借鉴。
如何发现FECR1的?
FLI1基因在造血系统肿瘤中存在高表达的情况,作者发现该基因在乳腺癌中也存在高表达,因此作者希望能探索一下这个基因在乳腺癌中受调控的机制。
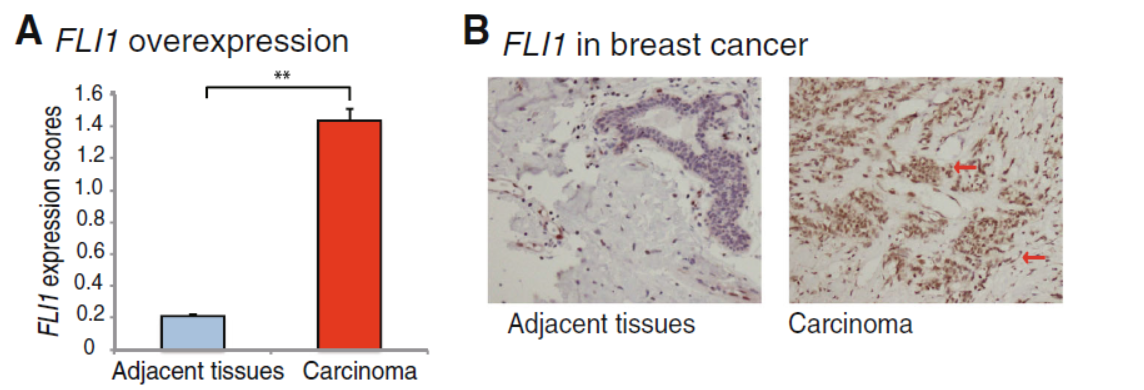
图1 FLI1在乳腺癌中高表达 (来自[1])
分析FLI1启动子区结合的分子是认识该基因表达调控机制的重要技术手段,作者采用了一种称为CasIP的方法,该方法的要点包括:Flag融合的dCas9蛋白,两条靶向FLI1基因启动子的gRNA,为防止RNA降解,作者采用了甲醛固定和原位反转录策略,反转录过程中掺入生物素标记的dCTP,这样就可以通过链霉亲和素纯化cDNA,分析结合在该启动子区的RNA分子。在所得到的结果中意外发现了来自FLI1基因外显子2-4的一种circRNA分子,作者称之为FECR1。
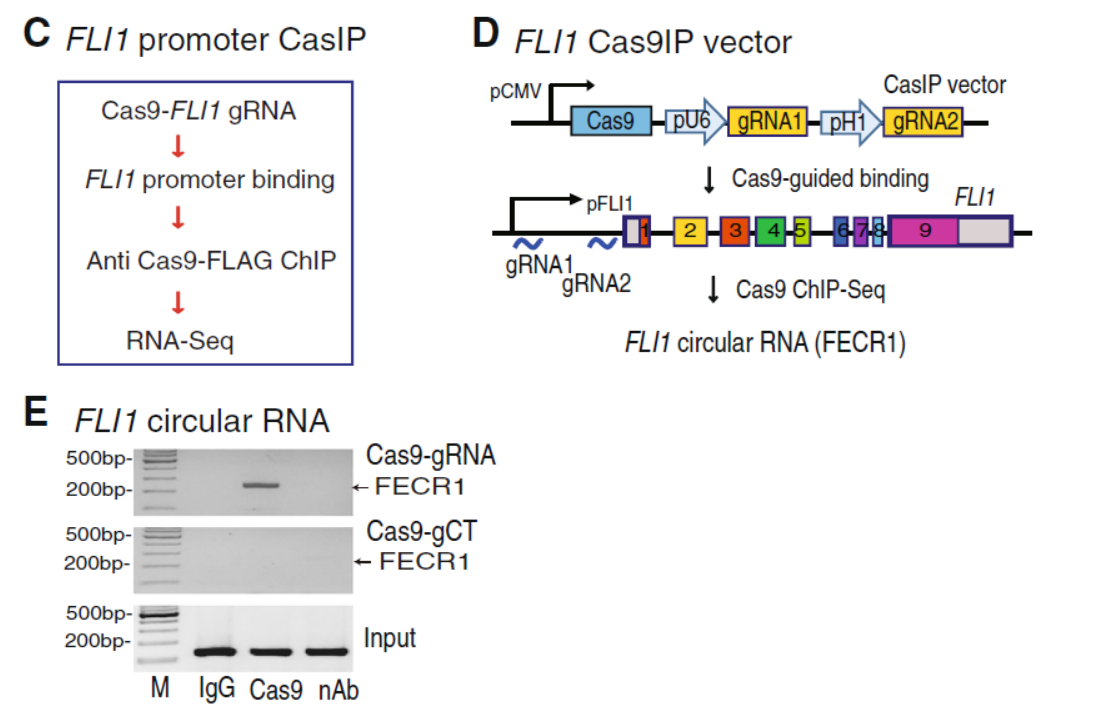
图2 基于CasIP技术发现FECR1在FLI1启动子区 (来自[1])
FECR1鉴定和功能分析
通过RNase R消化实验,Sanger测序进一步鉴定和确认了FECR1的存在,并发现了另一种由外显子2-5形成的circRNA,由于这种circRNA的含量很低,作者没有进一步研究。QPCR分析表明FECR1在乳腺癌细胞系和病人标本中表达较高。
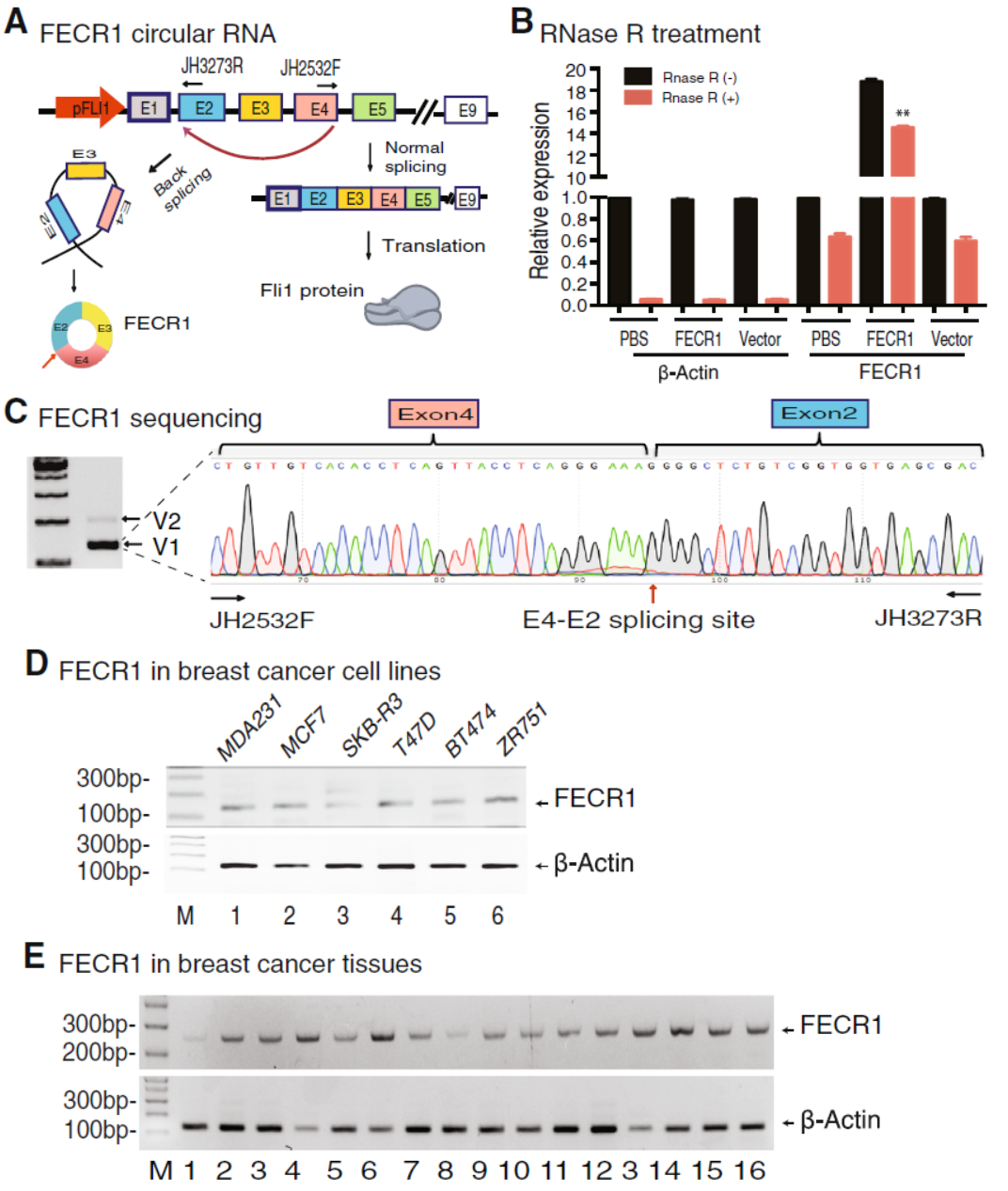
图3 FECR1鉴定与表达分析 (来自[1])
过表达FECR1显著促进乳腺癌细胞的侵袭能力。
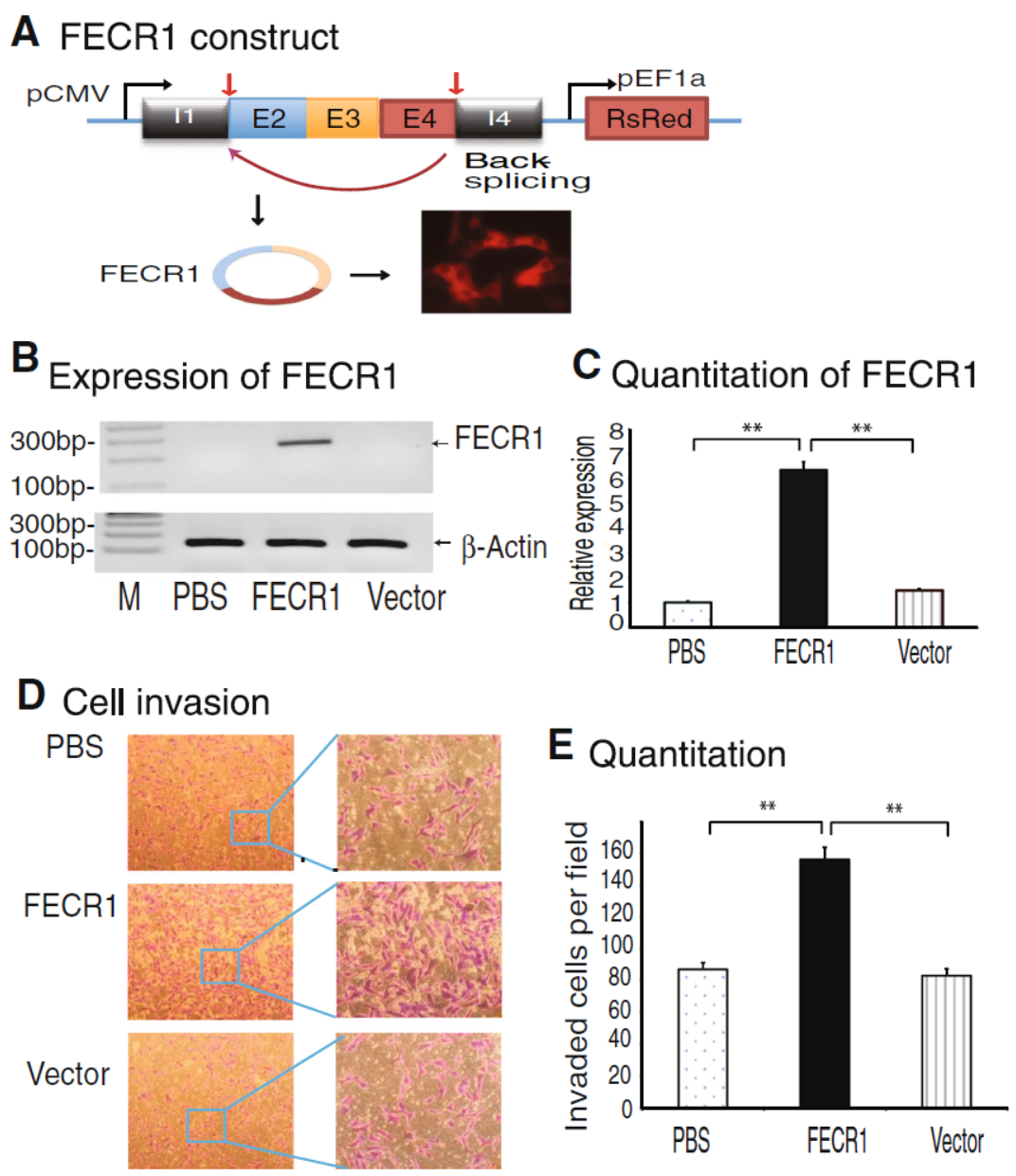
图4 过表达FECR1促进乳腺癌细胞侵袭 (来自[1])
FECR1作用机制分析
既然FECR1是从FLI1基因的启动子区分离发现的,那么这种circRNA分子作用的下游基因是怎样的?作者通过RAT分析(reverse transcription-associated trap)探索了这个问题,RAT分析的要点:原位反转录,掺入生物素标记的dCTP,染色质超声打断,用链霉亲和素进行ChIP分析。RAT-seq分析后发现了受FECR1调控的下游基因,这些基因主要与细胞增殖有关。进一步,基于RAT-PCR分析,作者发现FECR1主要结合在FLI1基因的启动子区,过表达FECR1能明显促进FLI1的表达。
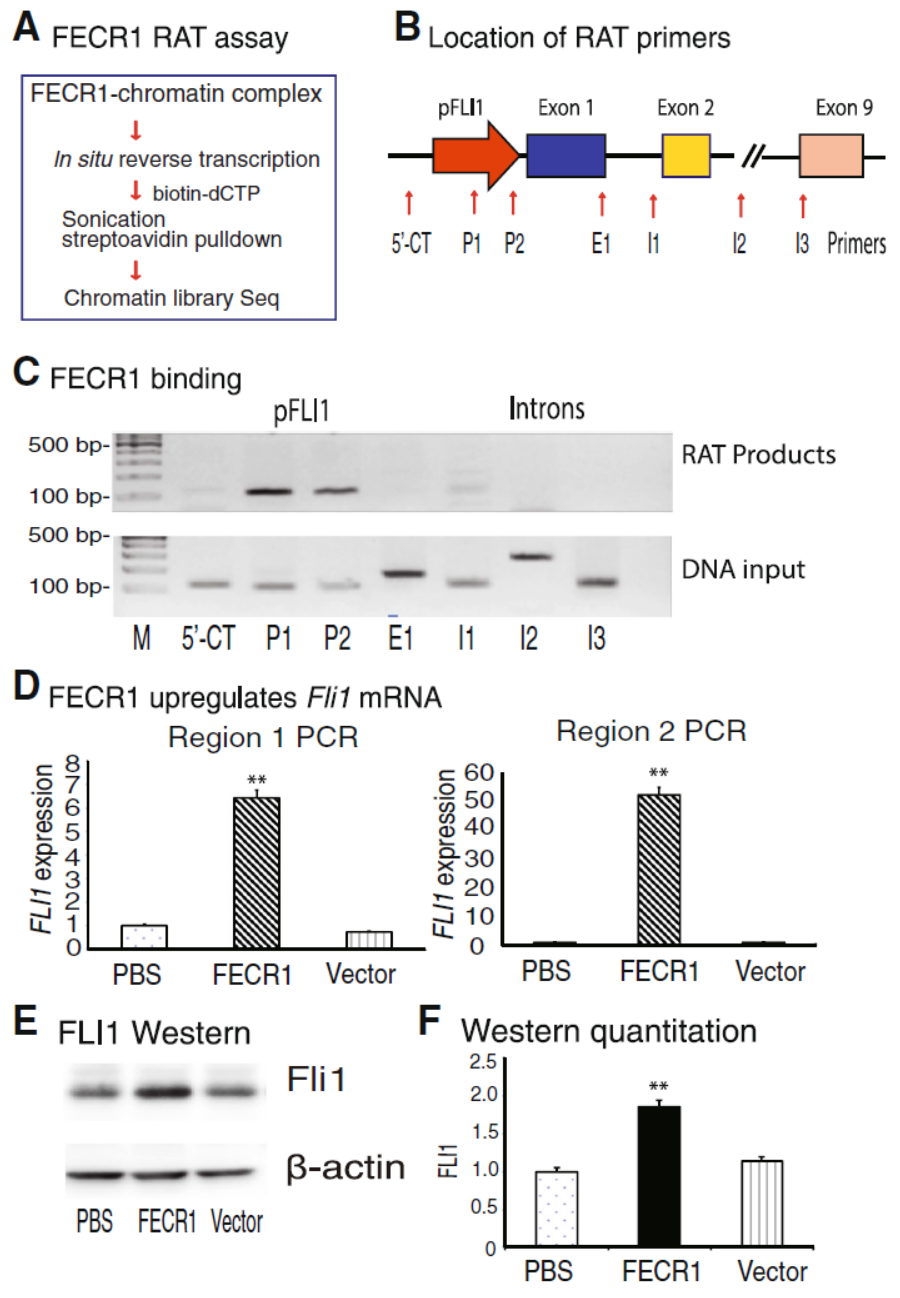
图5 FECR1结合FLI1基因的启动子区 (来自[1])
进一步,作者分析了FECR1结合位点的特征,发现FECR1主要结合在FLI1基因启动子的CpG岛。基于多种DNA限制性内切酶分析重亚硫酸盐处理前后的情况,发现FECR1结合后明显降低所结合区域的DNA甲基化状态。该方法类似于基因组分析中用到的限制性片段长度多态性(RFLP)技术。
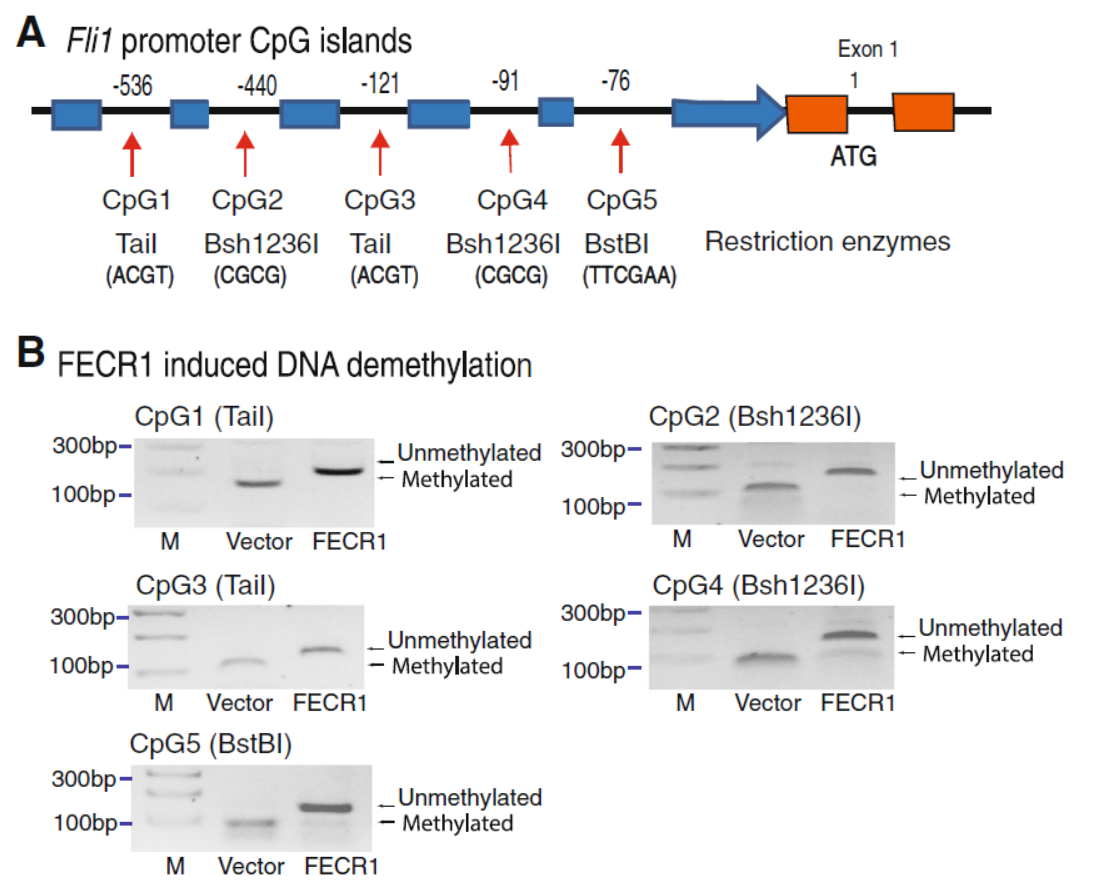
图6 FECR1结合FLI1基因的CpG岛,促进其去甲基化 (来自[1])
DNMT1是常见的参与基因启动子区CpG岛甲基化调控的甲基转移酶,有趣的是,作者在FECR1调控的基因中也找到了DNMT1基因,说明FECR1可以结合到DNMT1基因的启动子区。过表达FECR1明显抑制DNMT1的表达。此外,作者还在TET1抗体的RIP实验中捕获了FECR1分子,说明该circRNA也能与TET1结合。
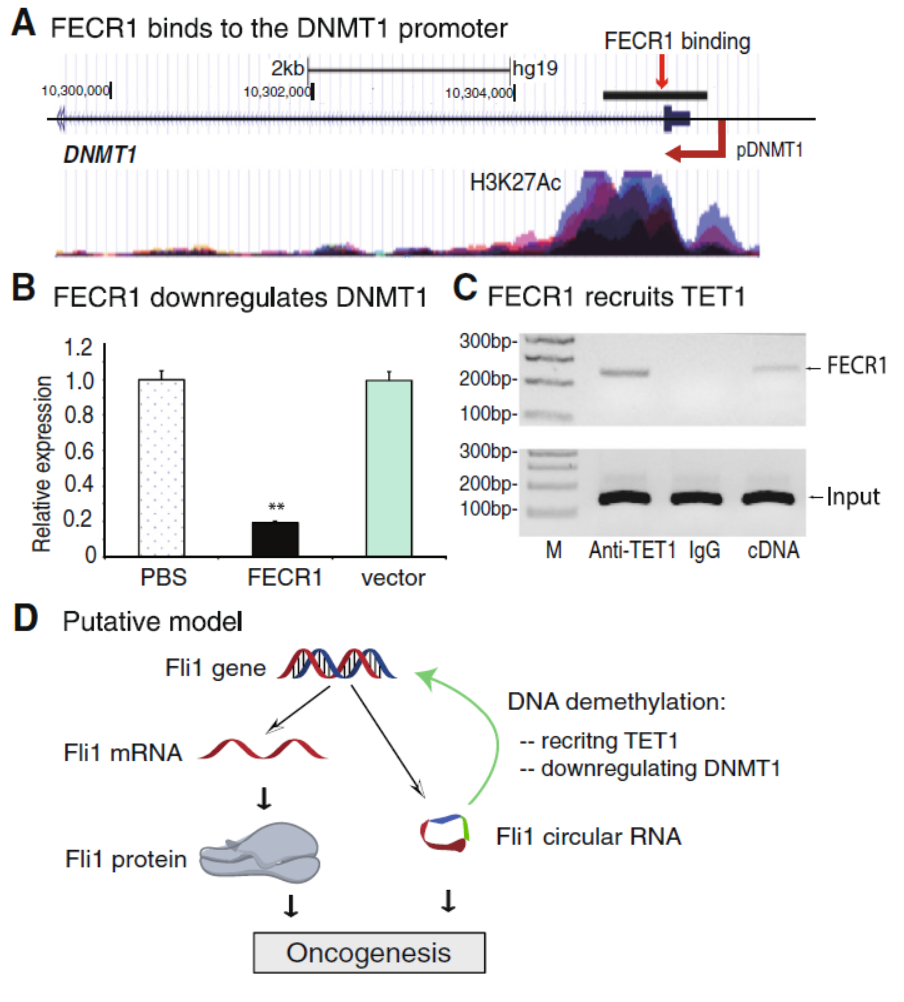
图7 FECR1结合DNMT1启动子,抑制其表达 (来自[1])
本文的故事比较简单,但挺有新意。丰富了我们对circRNA分子功能的认知,非常值得各位同行学习借鉴。
参考文献
1. Chen, N., et al., A novel FLI1 exonic circular RNA promotes metastasis in breast cancer by coordinately regulating TET1 and DNMT1. Genome Biol, 2018. 19(1): p. 218.
.png)