做科研最痛苦的是什么?是每周被导师追问N次实验进展?还是拿到被同行批到怀疑人生的审稿意见?付出巨大的努力却收获不到理想的结果才是最令人痛苦的。明明付出了比别人更多的努力,为什么发表的文章没人家的好?如果能用更短的时间找到功能更强的基因,是不是就能让自己的努力更有意义?同样的工作量,同样的努力,用在研究功能更强的基因和分子上一定会起到事半功倍的效果。
找到功能强大的基因和分子开展研究是生物医学科研的最关键的一步。
如何找到功能强大的基因和分子?
1月13日Nature Genetics在线发表的一篇文章的做法非常值得学习,他们使用Cas-9高通量文库的方法通过设计筛选模型直接得到筛选模型对应的功能基因ARID1A([1])。

文章内容简介:(原文摘要部分直接翻译的)
ARID1A是染色质重塑复合体SWI/SNF的一个亚基成分。ARID1A是ER阳性乳腺癌中SWI/SNF复合体最经常发生突变的亚基。作者发现ARID1在内分泌耐受的ER阳性乳腺癌中ARID1A经常会失活突变。基于CRISPR/Cas9高通量筛选文库,作者发现ARID1A在筛选ER降解剂药物Fulvestrant耐药相关基因中是打分最高的候选基因。ARID1A的失活可明显导致ER降解剂药物耐药,并可促进乳腺癌从ER依赖的luminal型向ER不依赖的basal-like型的转变。机制方面,ARID1A的失活可导致SWI/SNF复合体对几个luminal lineage决定性转录因子的调控作用出现异常,这些转录因子包括ER,FOXA1和GATA3。ARID1A的失活还能在全基因组层面改变ER-FOXA1与染色质的相互作用以及ER依赖的转录活性。本文的工作发现并证明了ARID1A在内分泌治疗中的关键作用,以及ARID1A在维持ER 阳性乳腺癌luminal类型中的作用。
关于原文中如何验证ARID1A的表达和功能的,感兴趣的读者可以直接拜读原文。就像题目所提到的,ARID1A的功能这么强,作者是怎么把它找出来的?下面就简单介绍一下高通量文库筛选技术:
什么是高通量文库筛选技术?
本文的工作非常严谨扎实,最重要的一步就是通过高通量文库基于功能筛选得到ARID1A基因,后面的验证和功能机制研究工作都是基于这个基础而开展的。那么这种高通量文库技术是如何实现基于功能的筛选的?
本文的高通量Cas9文库和工作原理剖析
本文中提到的CRISPR/Cas9高通量筛选文库是比较常用的高通量功能筛选工具,文中用的文库是靶向914种表观遗传调控相关基因的Cas9文库,每个基因设计了平均12条sgRNA。这些sgRNA被批量化合成后一起包装到病毒载体中,并进一步包装成一种病毒混合物(这里面包含了所有设计的sgRNA分子,每一个病毒只携带一种三个RNA序列,因此这个文库病毒是携带所有类型sgRNA的病毒混合物)。作者在此前已经构建好了能稳定表达Cas9蛋白的细胞株,用上面的这个病毒感染这个细胞株,通过调整细胞和病毒滴度的比例,实现理论上一个细胞只接触并被一个病毒分子感染,病毒准确感染了细胞后就可以整合到基因组中。到这一步,细胞群体中会存在感染了不同sgRNA的细胞个体,每个细胞只携带一种,细胞群体就可以包含所有sgRNA类型的细胞。作者让这些感染后的细胞先稳定生长两周,这样每个细胞中sgRNA和Cas9蛋白发挥作用,就会把对应的基因给敲除掉。整个细胞群就会存在敲出了不同基因的细胞。在这个基础上,分别用Fulvestrant处理,DMSO为对照,细胞会因为被敲除的基因不一样而表现出对药物敏感性的变化,最终会体现在细胞比例的变化。最后再通过收集两组细胞的基因组,通过比较药物处理组特异性富集和特异性丢失的基因,就可以找出功能基因。
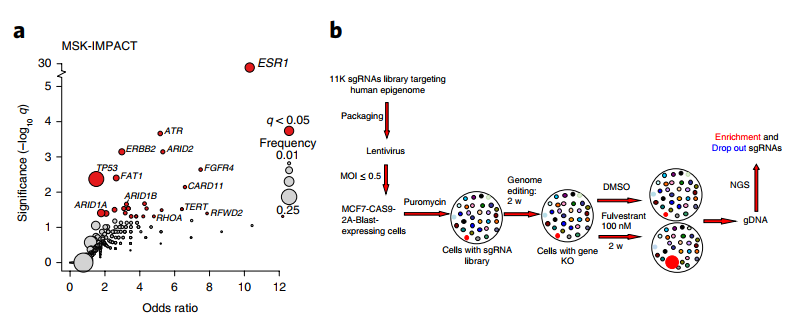
图1 Cas9文库筛选功能基因 ([1])
这种高通量文库筛选体系称为“Pooled Screening”,就是从一群细胞和文库分子中直接筛选候选基因的方法。
Pooled Screening文库应用综述
Pooled Screening文库已经在生物医学研究中发挥了非常重要的作用,在细胞生长,细胞信号通路,病毒感染机制,药物作用机制以及肿瘤免疫,表观遗传等等非常多的领域中得到了广泛应用。不仅仅上面提到的CRISPR/Cas9技术可以实现高通量Pooled Screening文库,shRNA,DNA Barcode分子都可以设计成这种高通量文库。关于高通量文库的技术和应用可以详细阅读下面的这篇综述([2])

作为近年来的明星分子
连续3年荣登生物科学领域新兴前沿方向的TOP1宝座
什么样的高通量筛选文库适合circRNA?
高通量Pooled Screening文库这么好用,能不能用到circRNA的研究中呢?什么样的文库能在circRNA中使用?2019年的一篇Cell文章就曾经用过高通量的shRNA文库进行功能性circRNA的筛选([3])。
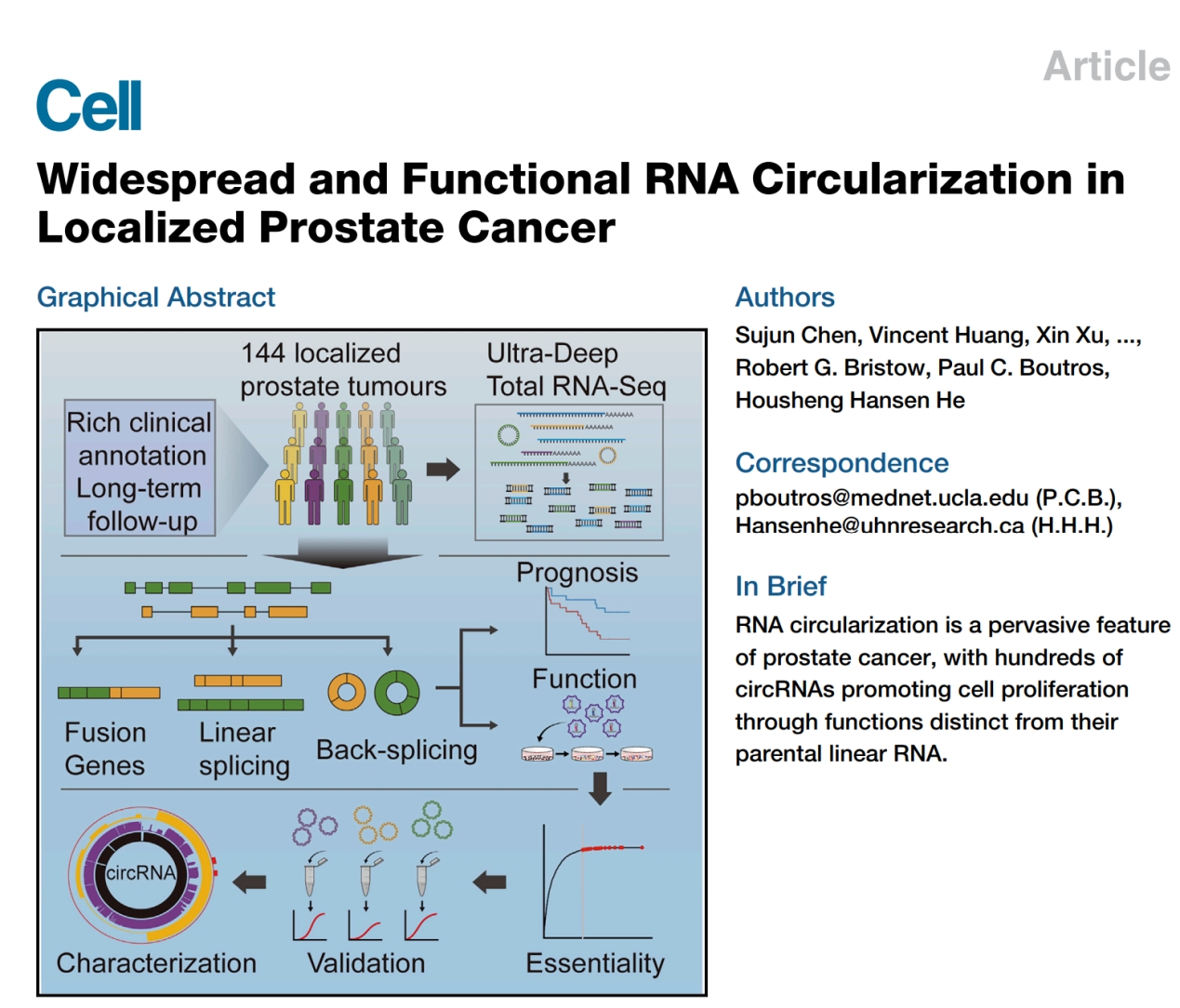
这项工作中作者用到的是针对circRNA的shRNA文库,筛选对前列腺癌生长所必须的essential circRNA,这些essential circRNA被干扰之后,细胞增殖显著抑制甚至细胞会发生死亡([3])。作者在这项工作中共分析到了171种essential circRNA,选择了10个进行干扰和过表达验证,结果表明所筛选到的circRNA在细胞增殖中的作用非常显著。
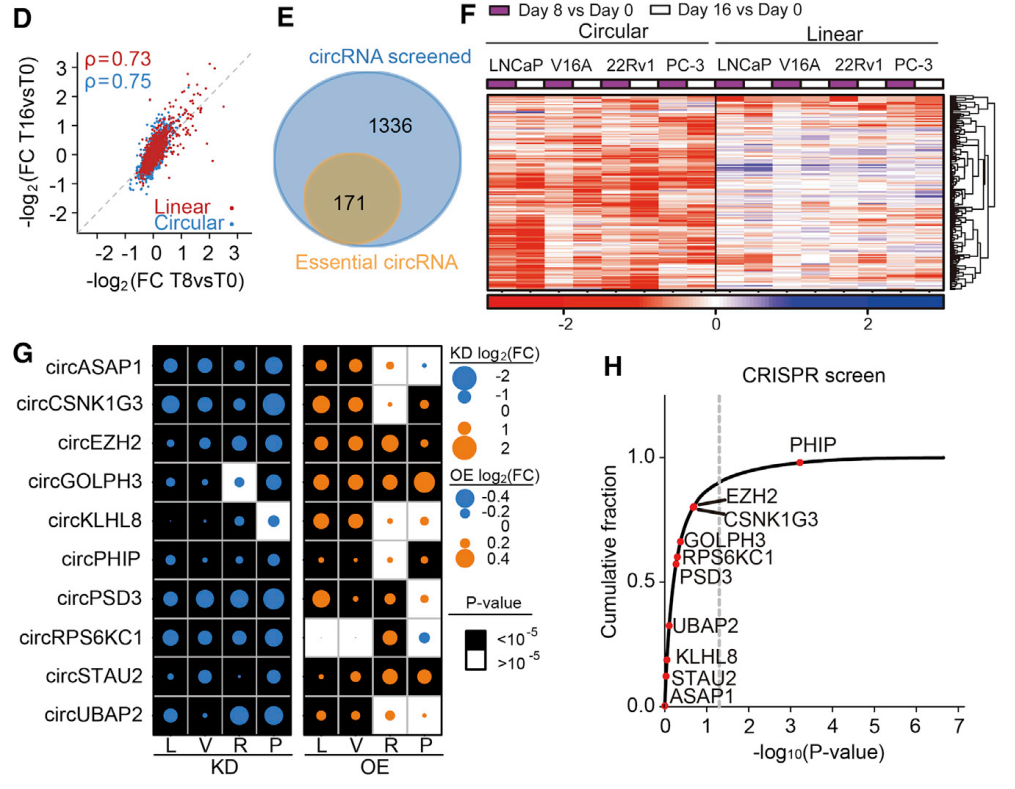
图2 基于shRNA文库筛选前列腺癌essential circRNA ([3])
看到这里,大家一定会眼前一亮,功能这么强的circRNA分子原来也可以用这个办法来找!但需要注意的是,因为绝大部分circRNA是从mRNA的外显子反向剪切形成的,要保证只下调circRNA而不影响对应的mRNA,只能在反向拼接位点来设计,因此只有shRNA才能保证只对circRNA起作用而不影响对应的mRNA。Cas9是mRNA敲除的重要技术,但目前在circRNA中应用还非常有限,只有个别circRNA可以通过设计实验并验证后实现敲除,目前还没法实现全基因组进行大规模的circRNA敲除而不影响对应的mRNA,因此Cas9文库还不适合circRNA。目前只有shRNA文库适合功能circRNA的筛选!
参考文献
1. Xu, G., et al., ARID1A determines luminal identity and therapeutic response in estrogen-receptor-positive breast cancer. Nat Genet, 2020. 52(2): p. 198-207.
2. Schuster, A., et al., RNAi/CRISPR Screens: from a Pool to a Valid Hit. Trends Biotechnol, 2019. 37(1): p. 38-55.
3. Chen, S., et al., Widespread and Functional RNA Circularization in Localized Prostate Cancer. Cell, 2019. 176(4): p. 831-843 e22.
.png)