简介
为了解决这个令广大科研人员苦恼的问题,浙江大学智能创新药物研究院周展教授团队开发了一个工具——DeepCIP,该工具采用了多模式深度学习方法专门对circRNA IRES进行预测,将更好地帮助我们研究circRNA的编码潜力以及提升circRNA药物的设计能力。
作者以预印本的方式于2022年10月发表了一篇题为 “DeepCIP: a multimodal deep learning method for the prediction of internal ribosome entry sites of circRNAs” 的文章。

算法优势
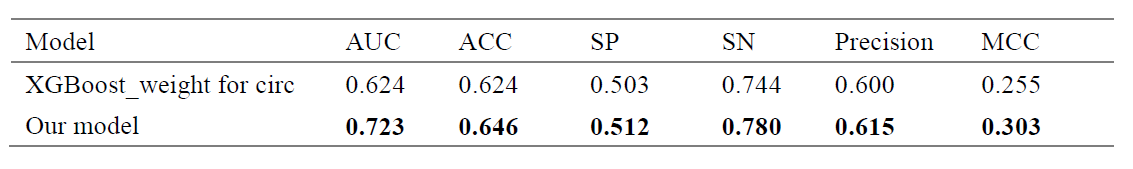
表1.DeepCIP和XGBoost_weight对circRNA IRES预测的比较结果
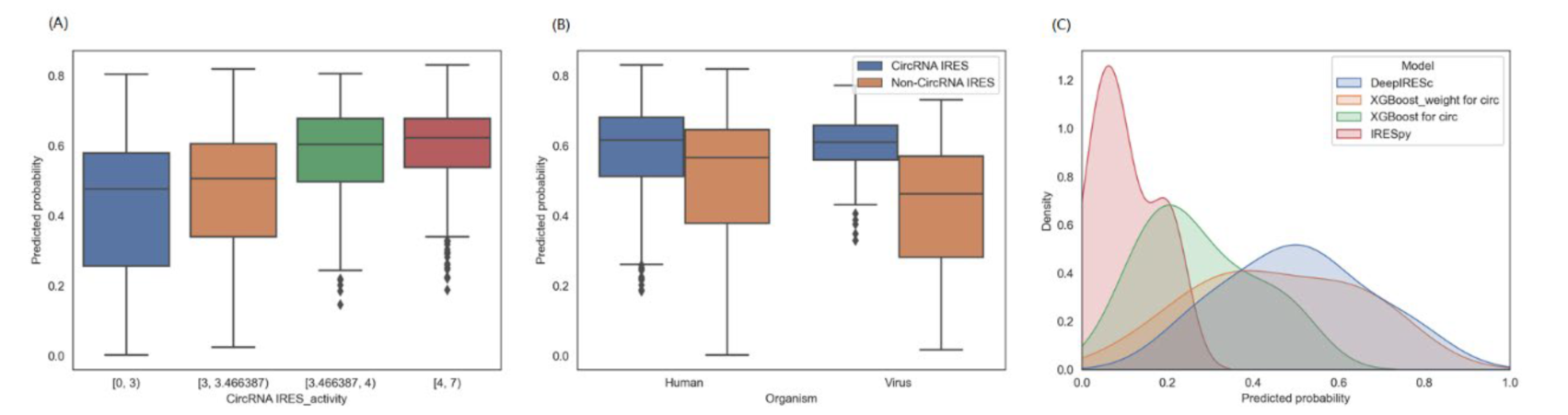 图1.DeepCIP与其他模型预测效果对比
图1.DeepCIP与其他模型预测效果对比
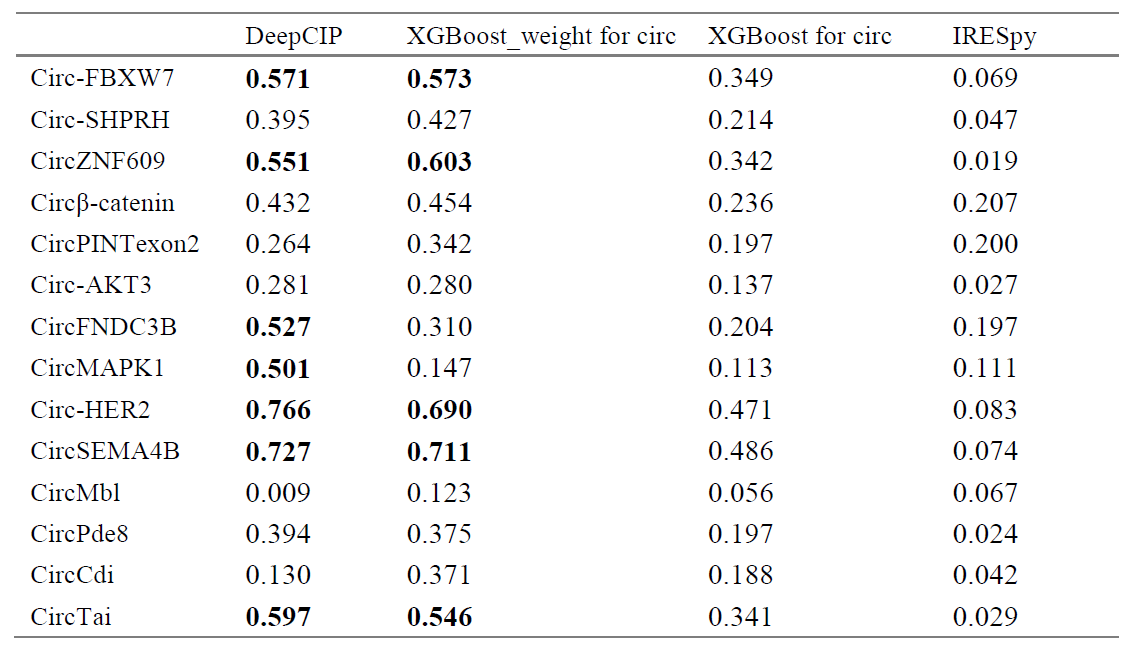
表2.本地真实数据集上不同模型的预测结果
方法策略
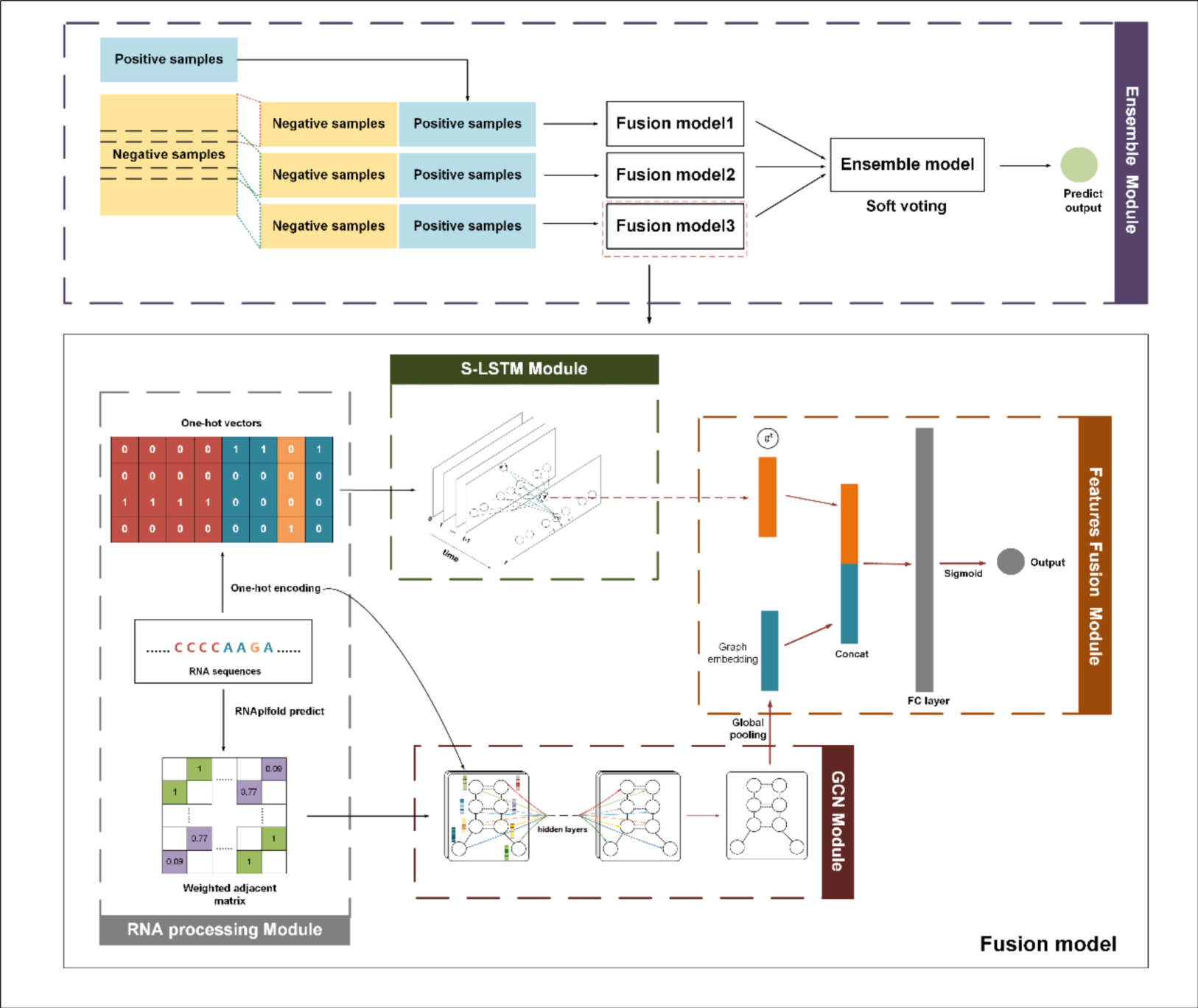 图2.DeepCIP的架构
图2.DeepCIP的架构
小结
以上是小编对DeepCIP做的一些简单介绍,作为第一款专门用于circRNA IRES预测的工具,DeepCIP的实用性可圈可点,感兴趣的朋友们可以尝试安装使用。
转载请联系邮箱授权:circRNA@163.com
.png)