circRNA 也有翻译潜能的事件被报道后,circRNA 的翻译很快就成了大家研究的热点。然而蛋白质翻译这个领域的研究一直比较硬核,更别说 circRNA 这一类分子背景还不够硬。
然而,金秋十月是收获的季节,circRNA 翻译研究又多了一把利刃 CircCode —— 还是基于机器学习的,由陕西师范大学的 Peisen Sun 与 Guanglin Li 共同开发。虽然之前也有 circRNA 翻译相关工具,比如 CircPro 或 circtools,但都是 mRNA 的“老黄历”。
实际上,这款工具的研究思路很简明,作者也为我们提供了一个非常漂亮的流程图
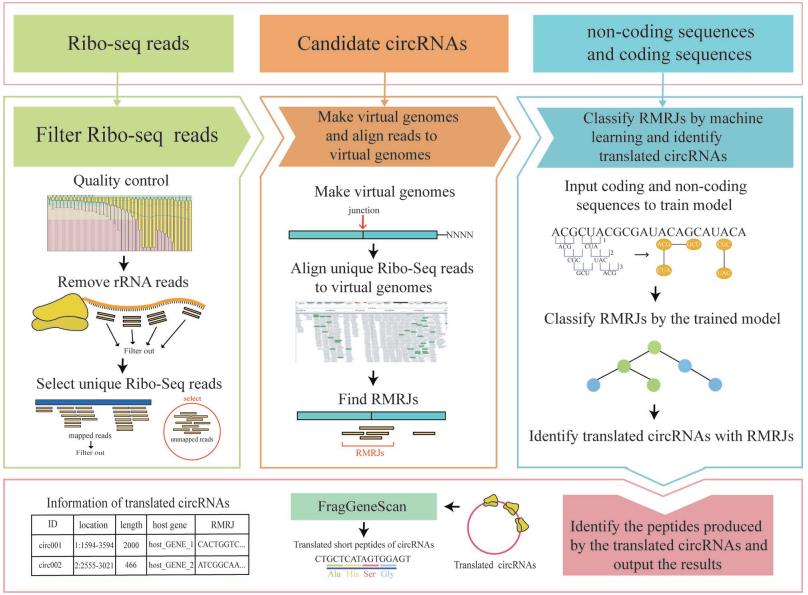
- 核糖体测序 reads 质控后过滤,保留没有比对上的 reads
- 将想要研究的 circRNA 序列以 junction 为中心提取 100nt 作为虚拟的参考基因组,然后将第一步保留的 reads 比对到参考基因组上。最后将跨 junction 位点的 reads 保留作为 RMRJs(Reads Mapped Region on a Junction),实际上是作为翻译的候选 circRNA。
- 通过机器学习工具 BASiNET 确定 RMRJ 是否可翻译,确定可翻译的 circRNA。
- 最后用 FragGeneScan 预测 circRNA 的 ORF 及多肽。
除了 PPT 做得好,CircCode 也需经受实际数据的考验。
- 作者从 RPFdb 数据库下载了人类与拟南芥的核糖体数据集,并用 CIRCPedia 与 PlantcircBase 所有 circRNA 作为 CircCode 的输入,最后识别到了大量可翻译的 circRNAs(人类 3610 个,拟南芥 1569个)。
- 父基因功能富集分析表面它们参与了蛋白质加工等生物学过程。
- 为了检验精确性,软件 GenRGenS 训练了已发表的可翻译的 circRNA 序列来测试 CircCode,最后 FDR = 0.0027。
- 已发表可翻译的人类 circRNA 中,有 60% 被 CircCode 所识别。
- 与 CircPro 相比, SRR3495999 数据中 CircPro 识别了 44 个可翻译的 circRNAs,而 CircCode 却识别到了 76 个。
最后,工具是基于 Linux 系统的,源代码及使用方法发布在 github 上。
作者虽然对软件的准确性与敏感性作了分析与比较,然而由于目前可翻译 circRNA 的数据太少,只能留给时间来检验。
总的来说,circRNA 研究又有了新工具。
.png)