circRNA 虽然被发现具有重要的生物学功能,并且与疾病的发生发展密切相关,但是目前对 circRNA 研究的手段仍停留在 mRNA 研究领域。如何、怎样、用神马策略来揭开 circRNA 在细胞和组织中如何度过它的一生,真可谓让全世界的爱好者绞尽了脑汁。
从最初的“达芬奇手术机器人”到现在上海儿科“AI医生”正式上岗,人工智能的发展可谓开启了生物医疗的新时代。而在基因领域,人工智能早已不遑多上,机器学习早已占领了生物学信息学的每一寸土地;而当前人工智能的核心驱动 —— 深度学习也已应用于识别基因中的突变,例如 DeepVariant,为疾病的早期筛查打下了基础。
相信大家对将人工智能应用于 circRNA 的研究跃跃欲试,这不,美国路易斯维尔大学 Juw Won Park 团队就在7月4日发表了一个工具 circDeep 用于分辨 circRNA 与其他非编码 RNA,目前唯一将深度学习应用于 circRNA 预测的工具。
可能越来越便宜的测序与众多的生物信息学工具给大家一个“错觉”:circRNA 的鉴定是一件容易的事儿。然而,当仔细品味转录组学时,会发现我们对转录组了解的真不多,就连某个基因有多少转录本而每个转录本时空效应是怎样的,可能都不清楚。circRNA 的检测也是这样的,我们只是达到了一个阶段,然而由于生物环境的复杂性以及非编码 RNA 低表达的特征,circRNA 特征刻画并不完整。
circDeep 的作者认为,鉴定 circRNA 需要三个基本步骤:
- 区分 RNA 是否编码蛋白质
- 区分 RNA 的长短
- 从长链非编码 RNA 中区分出 circRNA
在这之前,只有两个计算工具用于刻画 circRNA
- PredcircRNA
- 该软件利用多核学习框架(multiple kernel learning framework)训练保守性分数、ORF 等多个特征从而识别 circRNA
- H-ELM
- 利用分层极限学习机(hierarchical extreme learning machine)与特征选择区分 circRNA 与其他 lncRNAs
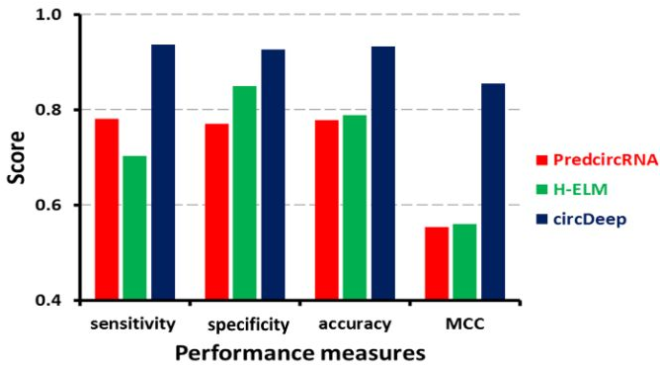
两种方法的精确度都没达到 80%,并且难以用特征描述 circRNA 的结构特性。
circDeep 将反向互补匹配特征(Reverse Complemetary Matching descriptor)、不对称卷积神经网络(Asymmetric Convolution Neural Network)以及长短记忆(ACNN-BLSTM)序列特征(ACNN-BLSTM sequence descriptor) 和保守性特征(conservation descriptor)融合成高度抽象的特征,提出了一个点对点(End-to-End)的深度学习框架,
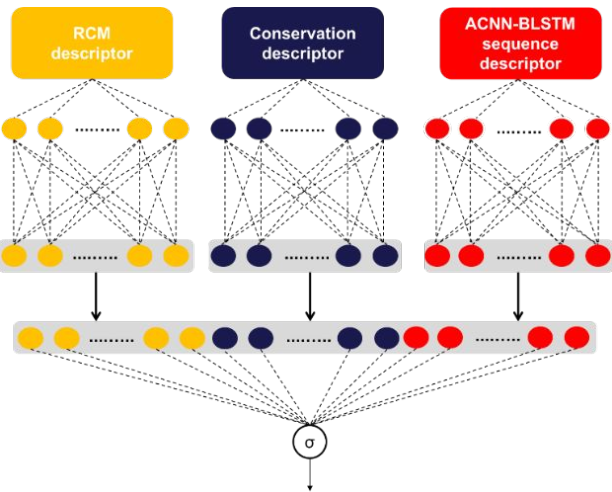
与 PredcircRNA 以及 H-ELM 用相同数据集比较显示,circDeep 具有最优的性能。
最后,作者将代码以及使用说明公开到了GitHub circDeep,并提供了相应的例子供大家使用。
加培训班的信息
.png)