环状RNA广泛存在不同的肿瘤中,目前高通量测序及计算方法,主要针对环状RNA的环化拼接位点进行鉴定,很难确定环状RNA内部的外显子和内含子序列剪切方式及组成,所以对环状RNA的内部结构难以明确。这将导致一些肿瘤特异性的circRNA可变剪切体(CIRC-AS)被忽略。武汉大学的何春江教授团队近日开发了一种新的算法——circSplice,可以特异性地识别circRNA内部可变剪切事件,并比较多样本组间circRNA可变剪切体差异(之前没有工具可实现circRNA可变剪切体组间差异比较)。该研究作者进一步在透明细胞肾癌和膀胱癌测序数据中利用circSplice分别检测到4498和2977个circRNA可变剪切体,后续RT-PCR实验结果也证实了circRNA可变剪切体的存在,进一步对circRNA可变剪切体的生物功能进行富集分析发现,癌症特异性CIRC-As的不同模式存在潜在重要功能,对多种肿瘤特异性环状RNA亚型的调控和功能研究具有重要意义。
目前,关于circRNA可变剪切体的研究还很匮乏,尤其是circRNA可变剪切体跟生物学功能或疾病特征关联的研究更少,因此何教授这一全新成果为探索circRNA可变剪切体的功能研究提供了非常便利的工具,下面我们一起来了解下circSplice算法的工作原理和运行机制。
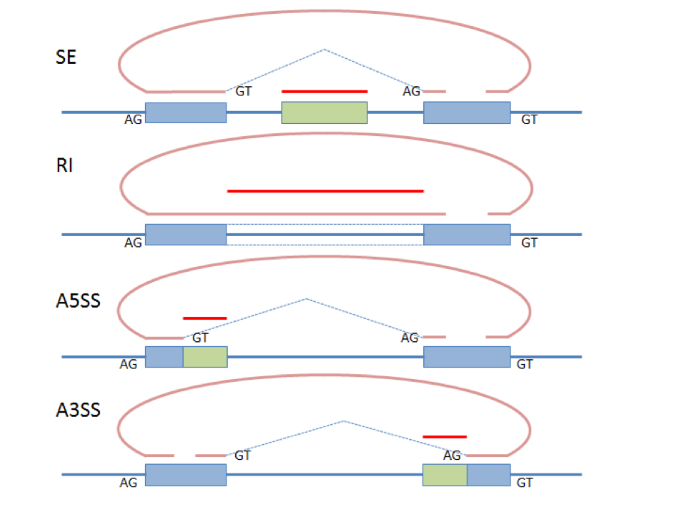
CircSplice是一种比较多样本间circRNA可变剪切(circ-AS)的新算法。CircSplice可以检测反向剪切read(BSJ)及其配对read中的4种AS类型,包括外显子跳跃(SE)、内含子保留(RI)、选择性5’剪切(A5SS)、选择性3’剪切(A3SS),预测过程充分考虑了GT-AG/CT-AC原则,同时独创性的支持不同样本间circ-AS的比较,比如,用户可以在癌和癌旁中检测癌症特异的circ-AS。CircSplice使用Perl编写,并且在Unix以及Mac OS X中进行了测试,在128GB内存和2.2GB CPU的机器上运行一个12G的样本仅需要10分钟。
一.CircSplice检测circ-AS示意图
二.CircSplice 工作流程:
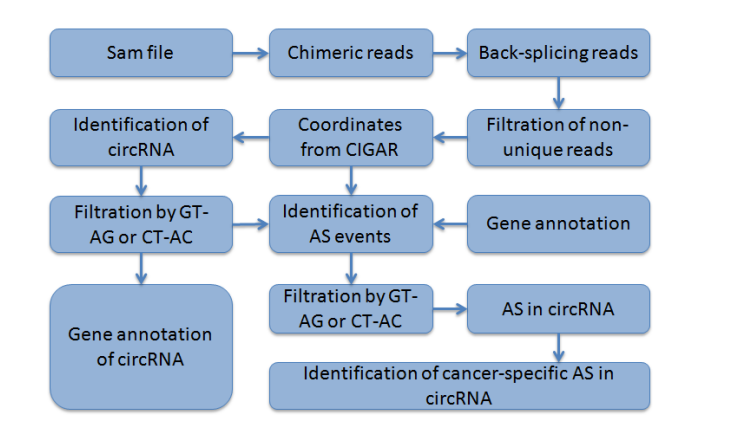 1、通过STAR比对至参考基因组序列,得到chimeric reads。
1、通过STAR比对至参考基因组序列,得到chimeric reads。
2、保留那些比对上同一染色体和链的chimeric reads,通过CIGAR值判断reads的坐标。
3、识别BSJ,去除另一端落在供体和受体位点之外的reads,以确保另一端也来自circRNA。
4、通过剪切位点GT-AG或者CT-AC对BSJ进行过滤和识别。
5、对BSJ进行基因注释。
6、将BSJ和其另一端reads比对到基因的外显子或内含子,结合基因注释进行4种剪切类型的判断。允许2bp容错。另外要求代表剪切的read的junction一端坐标与注释外显子的一端坐标一致,以减少假阳性预测。
7、通过GT-AG或CT-AC原则对可变剪切事件进行过滤。
8、计算可变剪接事件的reads数,并进行标准化(支持可变剪切的reads数*106/总chimeric reads数)。
9、根据坐标融合不同样本的剪切事件,比较样本间的环状RNA剪切事件。允许2bp容错。
Circsplice是开源工具可以从
http://gb.whu.edu.cn/CircSplice或https://github.com/GeneFeng/CircSplice获取,网页提供了详细的介绍。
参考文献:
Feng J , Chen K , Dong X , et al. Genome-wide identification of cancer-specific alternative splicing in circRNA[J]. Molecular Cancer, 2019, 18(1):35.
.png)