随着高通量测序技术的广泛应用,研究人员可以针对不同的肿瘤类型进行高通量测序,并获得相应肿瘤的基因组数据,著名的肿瘤数据库TCGA、Oncomine、ICGC等就是很好的例子,但是每个肿瘤数据库都有其特色,如何能够综合起来分析多种肿瘤的基因数据,更好地、客观地分析某些基因对于多种肿瘤发生发展的影响和预后。接下来,将为小伙伴介绍一下很强大的综合性数据库——cBioPortal,不需要注册登录即可免费使用的nice数据库。
cBioPortal for Cancer Genomics是一种开放获取的开源资源,用于多个癌症基因组学数据集的交互式探索。cBioPortal显着降低了复杂基因组数据与癌症研究人员之间的获取障碍,促进快速、直观、高质量地获取大规模癌症基因组学项目的分子谱和临床预后相关性,并使研究人员能够将这些丰富的数据集转化为生物学见解和临床应用。
cBioPortal网站目前存储DNA拷贝数数据(每个基因的假定,离散值,例如“深度缺失”或“扩增”,以及log2水平),mRNA和microRNA表达数据,非同义突变,蛋白质水平和磷蛋白水平(RPPA)数据,DNA甲基化数据和有限的临床数据。
如何使用好cBioPortal?
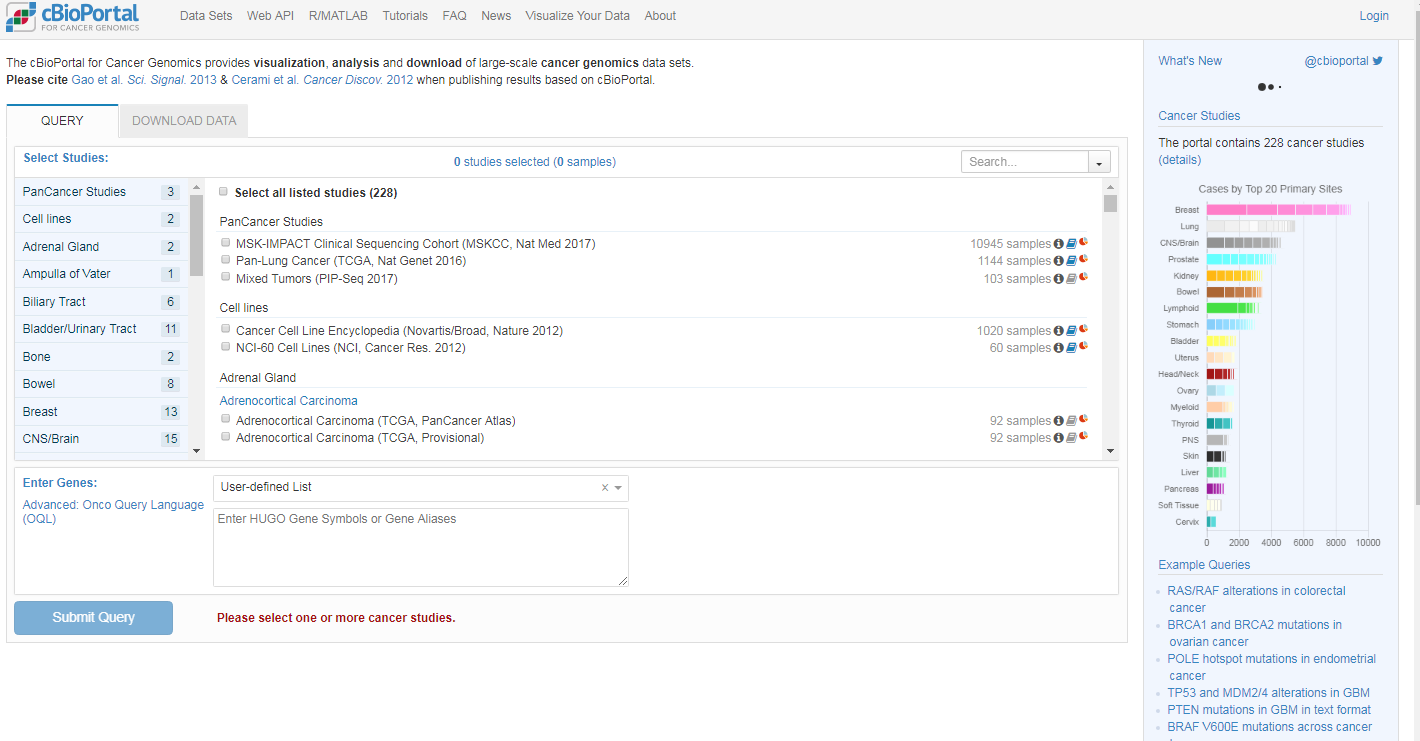
- 首先登陆cBioPortal for Cancer Genomics官方网站http://www.cbioportal.org/,界面如下;
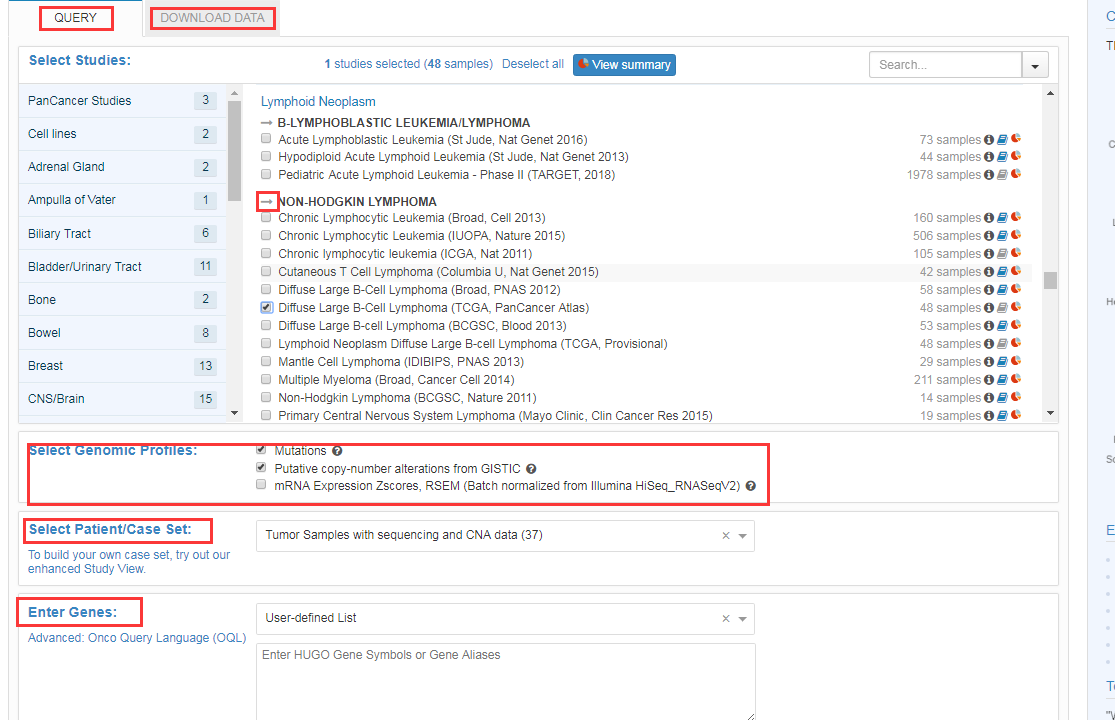
- 现以弥漫性大B淋巴瘤为例进行检索,界面的左上角有查询界面和数据下载两大功能,首先选择好研究肿瘤的类型,向右箭头可以对该项目进行全选,勾选不同的数据库会出现不同的“Genomic Profiles”,其中的mRNA表达谱尤为重要,记得勾选,后续可以进行多项分析和预后相关预测。另外“Patient/Case Set”根据需要选择,最后输入你想研究的基因;
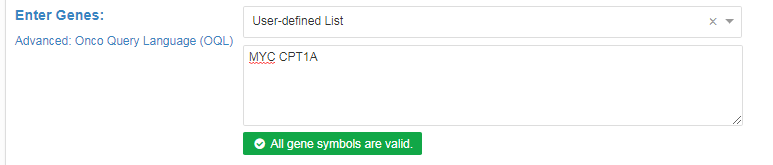
- 可同时输入多个基因名称,以空格隔开即可,系统会检验你输入的基因名称是否正确或者数据是否存在这个基因的表达,出现绿色标签“All gene symbols are valid”,说明你输入基因正确,如
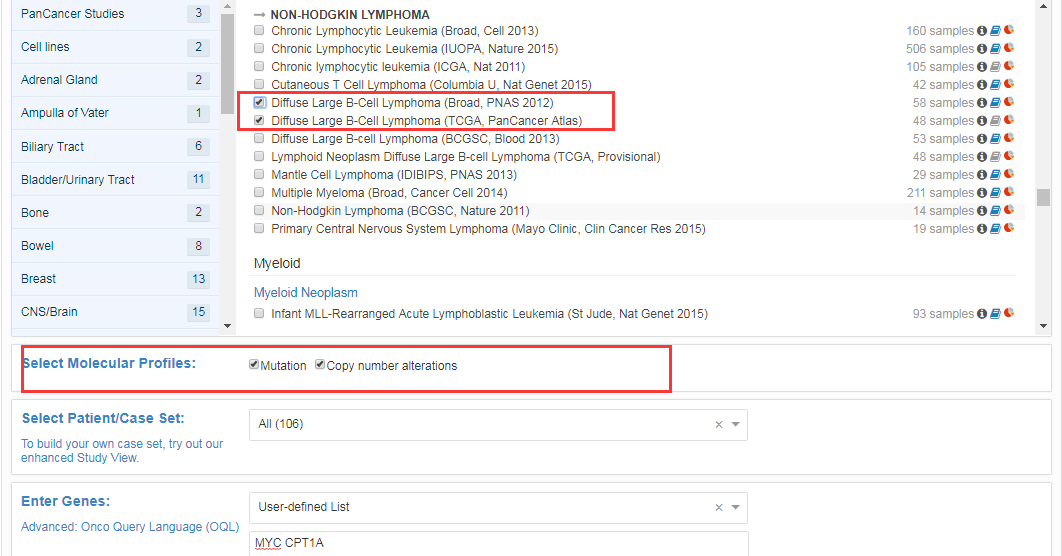
- 建议只勾选其中一种肿瘤分析,因为勾选两种以上的肿瘤时,“Select Molecular Profiles”无法出现mRNA表达谱勾选项,导致查询结果无法分析mRNA的表达等项目,可能是由于不同数据集来源导致的bug;
- 查询结果如下,myc基因在DLBCL肿瘤病人中的基因改变类型的比例情况;
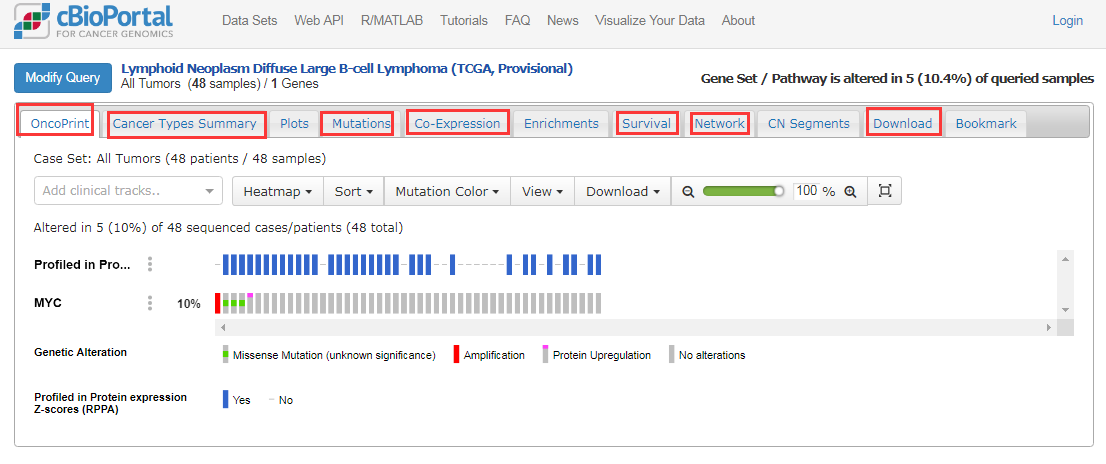
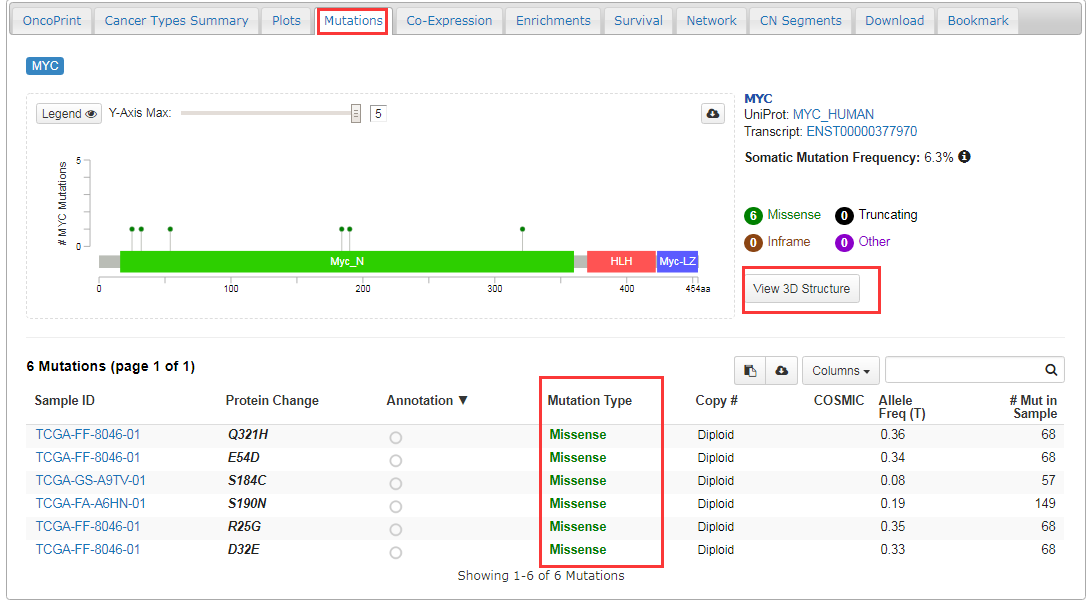
- 点开“mutations”项,可查看MYC在DLBCL肿瘤病人中的基因突变的具体情况,还可查看myc蛋白的3D结构;
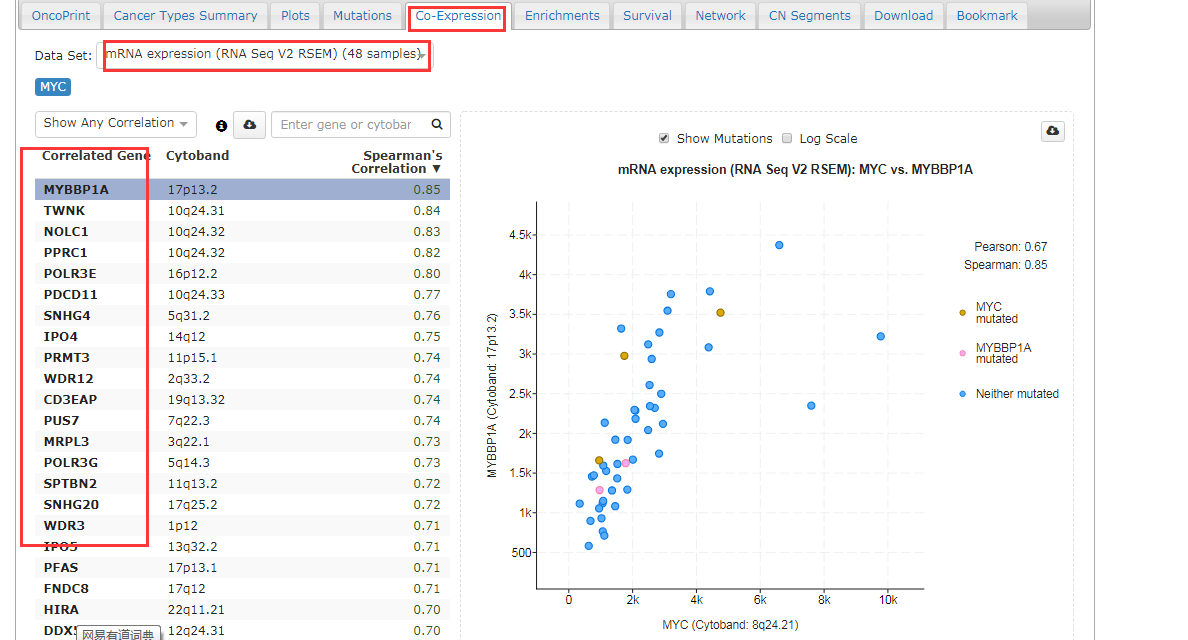
- 点开“co-expression”,可查看MYC基因和其他相关基因的共表达分析情况,选择相应的基因,右边会出现表达正相关的文章级别图;
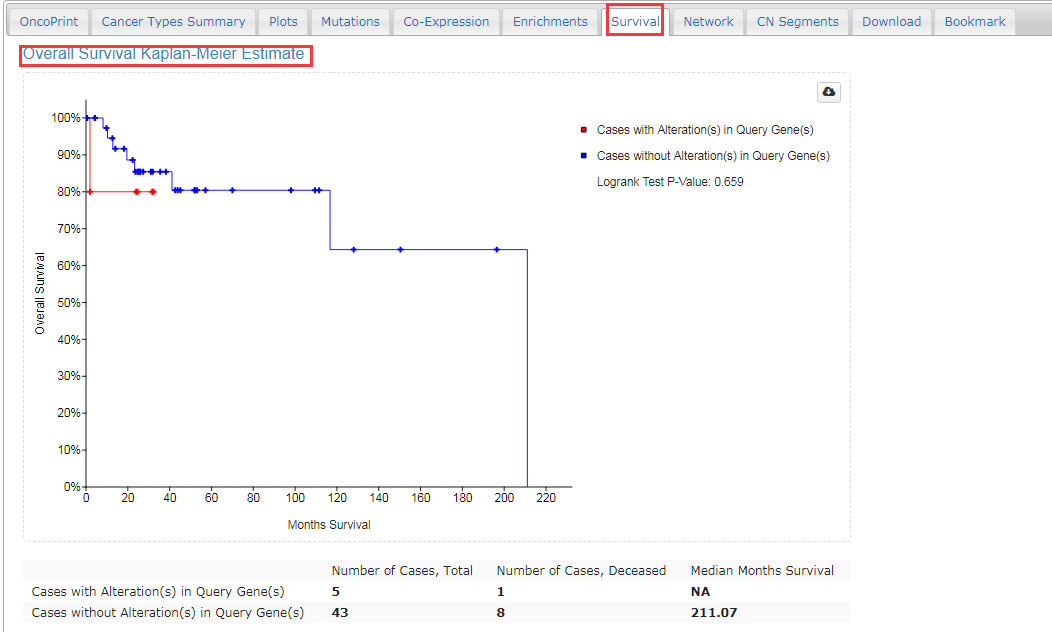
- 点开“Survival”,可查看myc基因是否发生突变与病人的OS、DFS的相关性,这个对于研究基因的临床相关性和临床应用有重要的作用;
9.点开“network”,可查看myc基因与该肿瘤中最频繁发生改变的50个基因的调控网络关系;
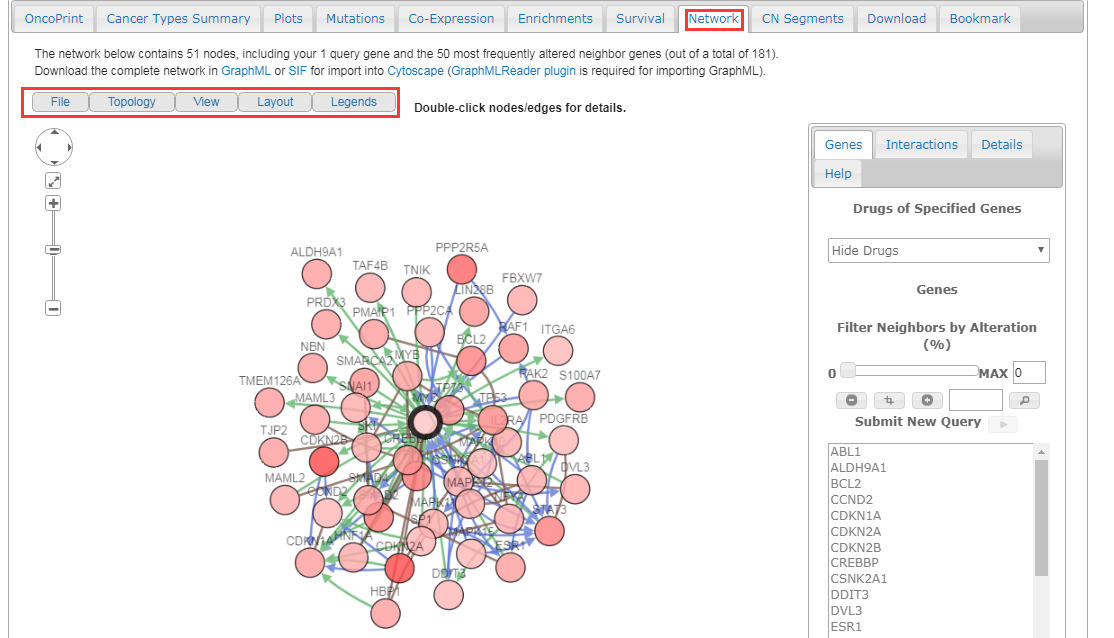
总结:以上几个功能是cbioportal的重要功能,当然还有很多小功能,如查询基因在肿瘤中突变类型的总结分析、点图展示、突变基因的enrichment分析和基因突变数据下载等等,在这里就不一一赘述了,小伙伴如果感兴趣的话,可以先学习一下怎么使用该数据库,在深入挖掘它的功能,有些功能真的好强大,有些图就是文章级别的图,可以直接应用于发表paper,但是需要引用,参考网站的说明即可。如果小伙伴觉得这些内容分享还可以,请多多点赞,转发,多多关注该公众号哦哦!!

.png)