
2019年10月10号,BioRxiv在线发表了一篇关于SRSF3在circRNAs生成过程中作用的论文,文章的通讯作者为费城儿童医院的Andrei Thomas-Tikhonenko博士。
生物体主要通过两种机制来调控circRNAs的生物发生:一种是与侧翼内含子序列中的Alu元件相关,即,当两个外显子通过侧翼内含子中所含的反向互补的Alu元件彼此靠近时,某些circRNAs会被反向剪接。另一种与侧翼内含子序列中包含RNA结合蛋白的结合基序,即,当与反向剪接外显子侧翼内含子结合的RNA结合蛋白(RBP)发生二聚化时,就会发生外显子的反向剪接。
在这篇研文章中,作者分析了P493-6细胞(Burkitt淋巴瘤的模型细胞系)的circRNAs,发现2/3的circRNAs侧翼内含子中包含Alu元件,另外1/3不包含Alu元件。那这些侧翼内含子序列不包含Alu元件的circRNAs是如何形成的?是由RBP结合蛋白介导的吗?
作者通过分析不含Alu元件的circRNAs的侧翼内含子序列是否含有已知的与RBP蛋白结合的基序,发现这些circRNAs的侧翼内含子序列包含多个RBP结合基序,这些RBP包括SRSF3和PTBP1,表明RBP介导P493-6细胞中1/3的circRNAs的生物发生。哪个RBP在circRNAs生物发生中起主要作用?考虑到SRSF3可以促进mRNA的可变剪切,而大多数circRNAs为外显子circRNAs,因此本文主要关注SRSF3在circRNAs生物发生中的作用。怎么验证SRSF3确实在circRNAs生物发生中起作用?本文通过干扰P493-6细胞中的SRSF3的mRNA水平,通过RNA-seq,发现926个基因在干扰系和对照组之间有不同的可变剪切。重要的是,这些基因中1/5对应于P493-6细胞中鉴定的circRNAs的亲本基因,表明SRSF3确实在circRNAs生物发生中起重要作用。
此前关于circRNA与SRSF之间关系的研究论文有两篇:
Barbagallo, D., Caponnetto, A., Cirnigliaro, M., Brex, D., Barbagallo, C., D’Angeli, F., et al. (2018). CircSMARCA5 inhibits migration of glioblastoma multiforme cells by regulating a molecular axis involving splicing factors SRSF1/SRSF3/PTB. International journal of molecular sciences, 19(2), 480.
Barbagallo, D., Caponnetto, A., Brex, D., Mirabella, F., Barbagallo, C., Lauretta, G., et al. (2019). CircSMARCA5 regulates VEGFA mRNA splicing and angiogenesis in glioblastoma multiforme through the binding of SRSF1. Cancers, 11(2), 194.
这两篇文章均报道一个circRNA CircSMARCA5调控SRSF3/1剪切因子,目前还没有SRSF调控circRNAs生物发生的报道。
下面就让我们一起学习一下本文:
P493-6细胞中表达的环状RNA
在本文中,作者选用最保守和准确的工具mapSplice2在P493-6细胞中共鉴定了来自957个亲本基因的1324个circRNAs。随后,作者通过比较P493-6细胞和相似的MC116细胞的亲本基因和circRNAs,验证鉴定的circRNAs。两个细胞系中非重叠的circRNAs可能是由于使用的技术和算法不同导致的。有趣的是,一些非重叠circRNAs是之前研究中未报到的,表明在P493-6细胞中存在新的circRNAs.
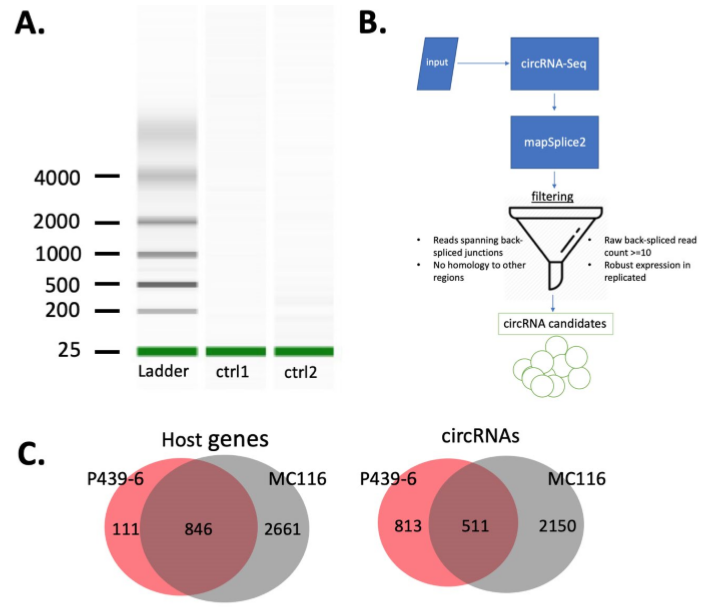
图1 P493-6细胞中鉴定的所有circRNAs
接下来,作者分析亲本基因和circRNAs之间的对应关系,发现三分之一的亲本基因会形成多个circRNAs,并列举一个亲本基因形成一个circRNA,以及一个亲本基因形成多个circRNAs的具体例子。
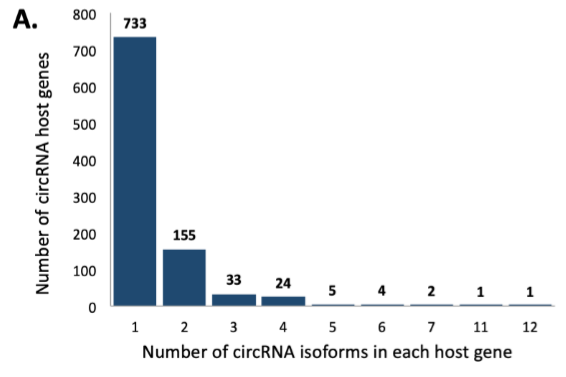
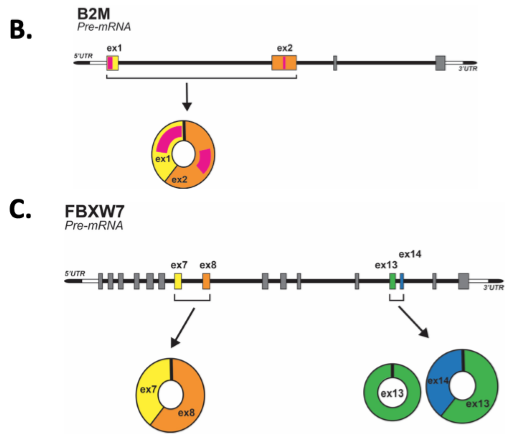
图2 circRNAs的形式
CircRNAs侧翼内含子序列中包含Alu元件和不包含Alu元件的比较
为了检测先前鉴定的circRNA生物发生模型,作者试图分析Alu元件与反向剪接之间的关系。首先在候选circRNAs数据库中,查找形成circRNAs的两个外显子侧翼内含子序列中的Alu元件,结果,有840个circRNAs的侧翼内含子中包含Alu元件,而484个circRNAs不包含,表明只有一部分circRNAs遵循先前的模型。
SRSF3在circRNAs形成过程中的作用
对于上述现象,作者假设是RBP参与了circRNAs的生物发生。使用MEME工具,作者分析了侧翼内含子(包含链接外显子的250 bp)的RNA结合基序,发现多个RNA结合基序,对应的RBP包括PTBP1和SRSF3等。另外,作者也发现这些RNA结合基序结合相应的RBP蛋白有选择性,特别是对PTBP1和SRFS3这两个RBP蛋白。并且,这些区域可能与多个RBPs在不同的核苷酸位置结合,从而揭示了位置结合偏差,导致同一亲本基因产生不同的circRNAs。由于大多数circRNAs是外显子circRNAs,而SRSF3通常可促进外显子包含,因此本文专注于这一特殊的剪接因子。
图3 circRNAs侧翼内含子序列中Alu元件和RNA结合基序的分布
随后,作者试图确定SRSF3的可变剪接靶标。为了模拟SRSF3的下调并确定受影响的mRNA和circRNAs,作者在P493-6细胞系中通过siRNA敲低SRSF3 的mRNA,同时以非靶向siRNA做对照。为了消除对单个siRNA的依赖,并平均它们的脱靶效应,靶向和非靶向分别设计4个siRNAs。结果,通过RT-qPCR和Western印迹确认SRSF3基因的敲低。然后,对SRSF3敲低最显著的72小时样品进行RNA-seq和MAJIQ分析,发现926个mRNA在对照和SRSF3敲除之间进行了不同的剪接,从而确定了SRSF3的靶标。其中,超过五分之一的mRNA也对应于本文鉴定的circRNAs,包括那些侧翼内含子序列中有SRSF3结合基序的circRNAs。 总之,这些数据表明SRSF3的下调不仅影响目标mRNA的剪接,也影响circRNAs的反向剪接。
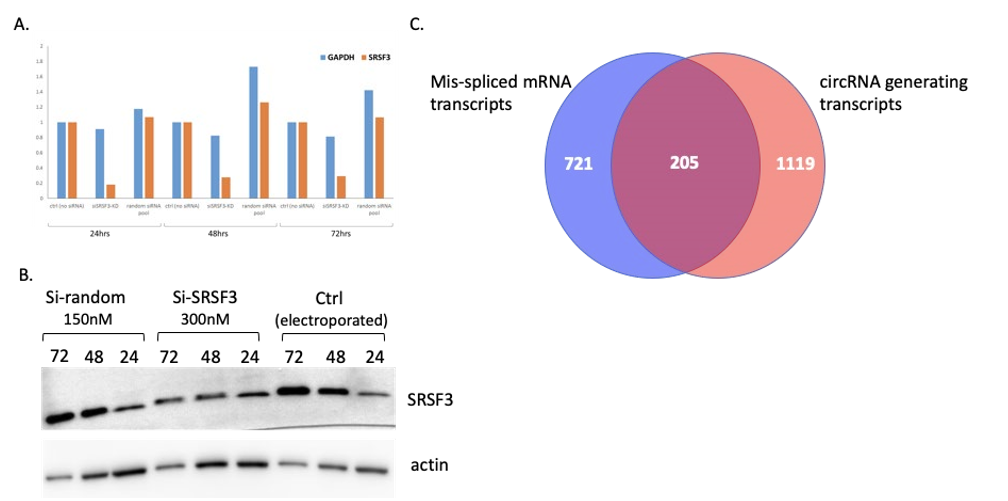
图4 P493-6细胞敲除SRSF3基因的鉴定及RNA-seq分析
参考文献
Ammar S. Naqvi, Mukta Asnani, Kathryn L. Black, Katharina E. Hayer, Deanne Taylor, Andrei Thomas-Tikhonenko. The role of SRSF3 splicing factor in generating circular RNAs. 2019. BioRxiv.
.png)