值此一元复始,万象更新之际,山人预祝各位同仁在新的一年里身体健康,阖家幸福,万事如意!
2月14日,Gene杂志在线发表了俄罗斯科学院分子遗传研究所Ivan B. Filippenkov为通讯作者的文章,介绍鞘磷脂合酶1(SMS1)基因可变剪切与circRNA形成机制的研究工作[1]。

当基因中包含超长的内含子时(长度大于20kb),RNA拼接过程将呈现为多步拼接的方式(multi-step splicing),其中会出现递归外显子(recursive exon,RS-exon)。递归外显子可能被最终完全切除,也可能会保留到最终的成熟产物中。绝大部分携带RS-exon的转录终产物会经过胞内nonsense-mediated decay途径降解,但也有一些基因会保留它们。
SMS1基因总长度达到320kb,包括至少20个外显子,编码区位于外显子7-11。作者之前研究曾发现该基因具有可变的启动子和Poly(A)信号,也包含携带RS-exon的转录终产物,其中包括携带RS-exon的circRNA分子。
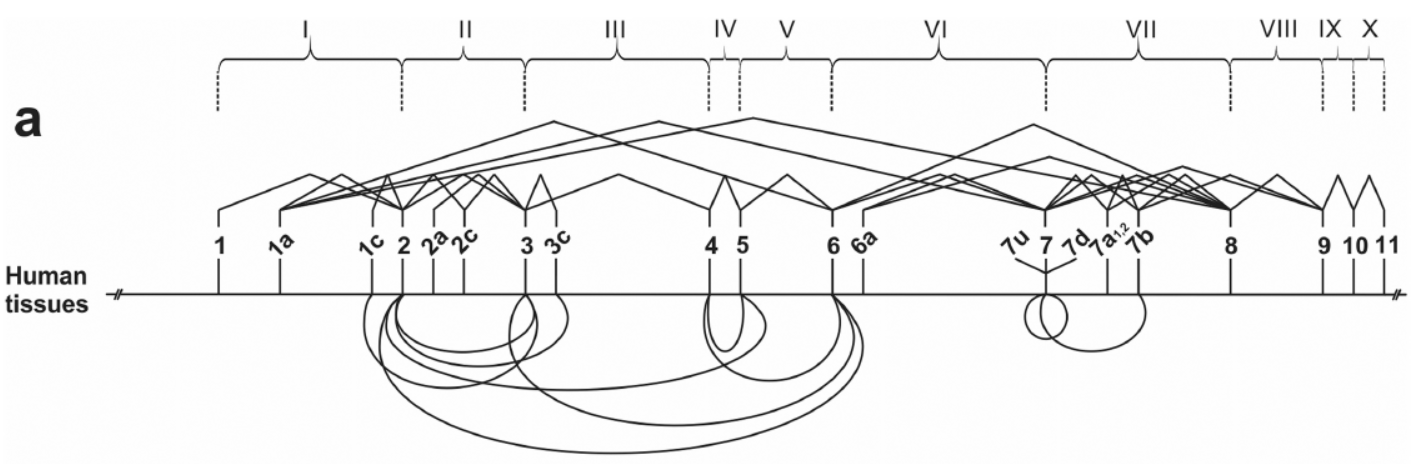
图1 SMS1基因已报道的外显子拼接方式 (来自[1])
本文主要基于靶向捕获测序技术(RNA-Capture Seq)分析了SMS1基因的转录产物,发现了一些新的外显子和可变剪切形式以及组织特异性的可变剪切和circRNA形成有关的现象。本文的结果有助于增加对组织特异性circRNA表达以及circRNA可变剪切的形成机制的认识。
本文作者发现的SMS1基因递归外显子信息列表如下:
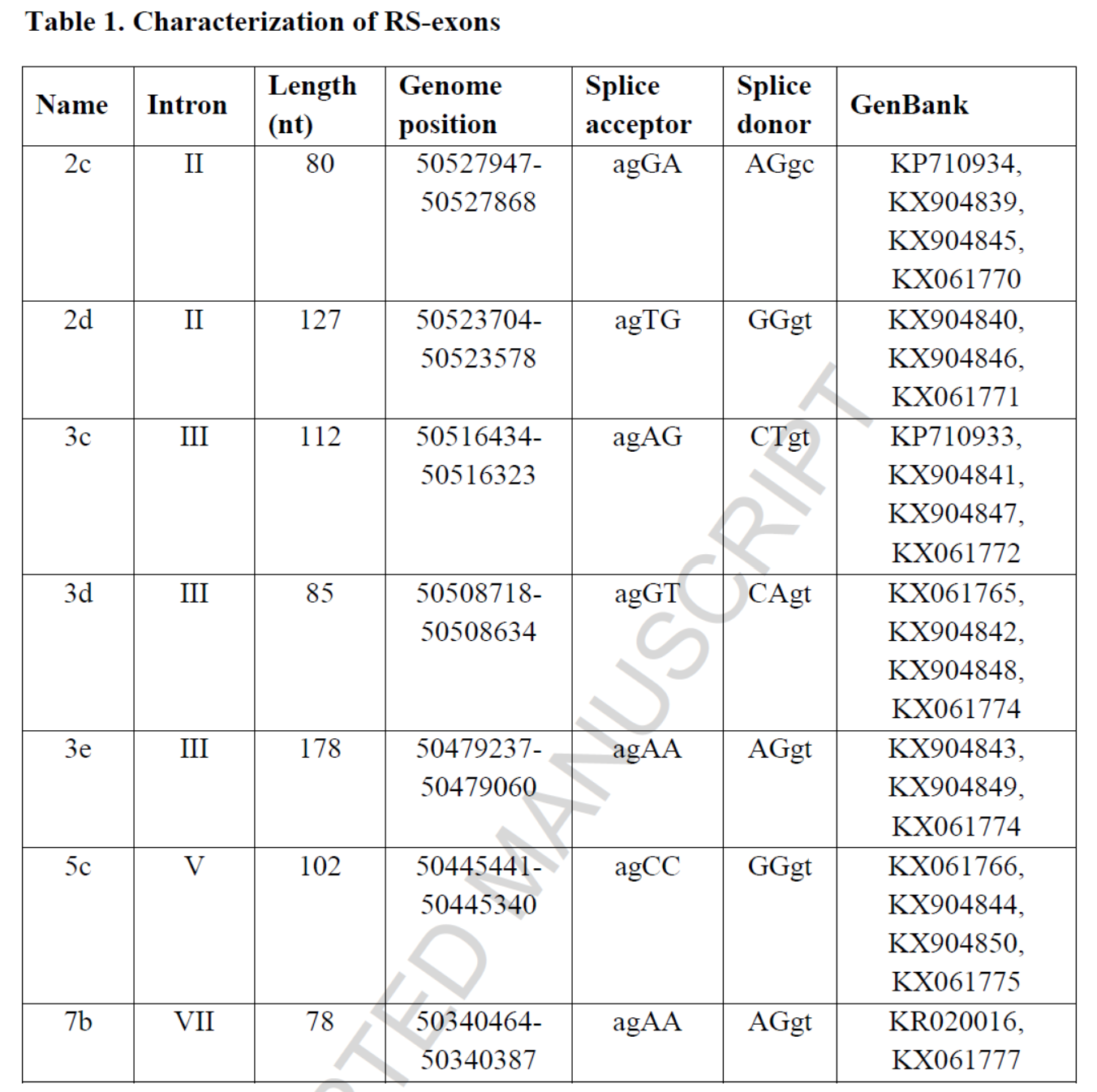
表1 SMS1基因来源递归外显子信息 (来自[1])
作者如何研究和发现这些递归外显子的?
RNA-Capture Seq是一种外显子测序技术,概括而言,就是基于已知的外显子序列设计靶向捕获探针,分离特定的RNA片段并扩增富集,然后进行高通量测序分析[2]。
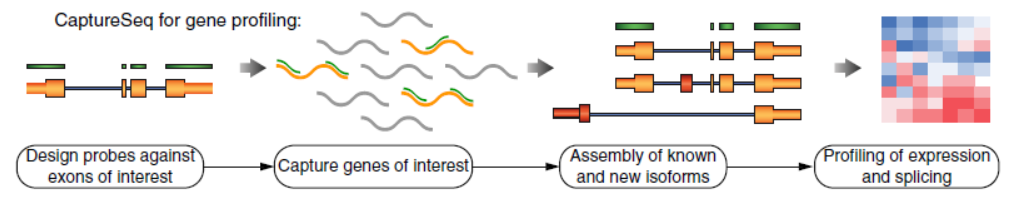
图2 RNA-Capture Seq技术 (来自[2])
基于RNA-Capture Seq测序分析,在测序结果中发现98%的Reads mapping到了SMS1基因的内含子区域。为方便探讨可变剪切的机制,作者挑选了不含重复序列的区域,选择1-11外显子的区域进行深入分析。本文作者在前额叶皮质组织中发现了13种新的可变剪切方式,其中12种包含了内含子中来源的外显子序列,1种新的位于5’区域上游的序列,称为U1外显子。白细胞中则发现了15种新的可变剪切形式,其中8种包含了内含子来源的序列,4种为丢失某个外显子的形式,还有3种包含了上游的新外显子序列(U1, U2, U3)。
从两种样品的测序结果中除了内含子4, 9和10种没有发现新的外显子序列外,其余的内含子中都包含有可保留为外显子序列的片段。大脑前额叶皮质和白细胞样品的测序结果中内含子1,3和8的可变剪切方式有非常大的差别。
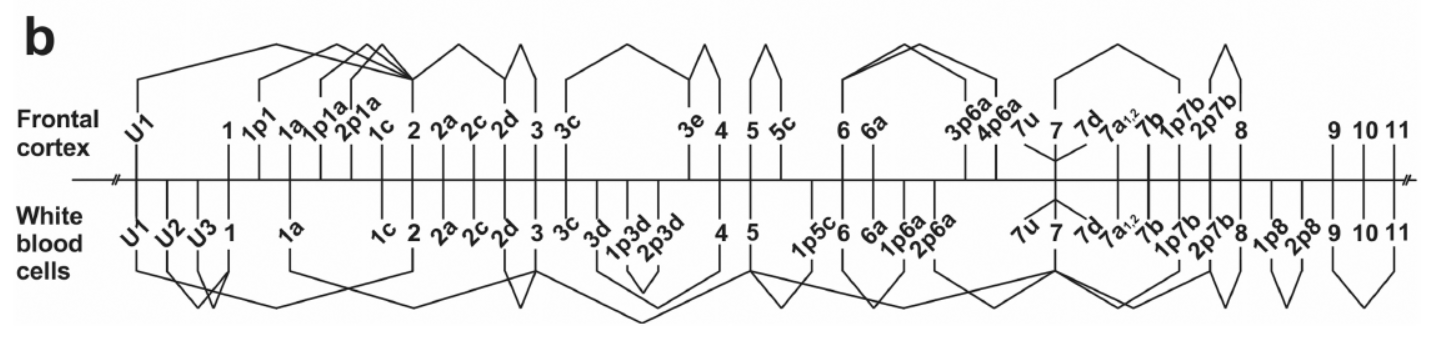
图3 人类大脑前额叶皮质和白细胞中SMS1基因可变剪切差异 (来自[1])
上述的分析结果是基于高通量测序的,需要进行RT-QPCR验证分析。作者设计了不同可变剪切体对应外显子的引物,进行了半定量PCR和QPCR分析,证明了对应的可变剪切产物的存在。
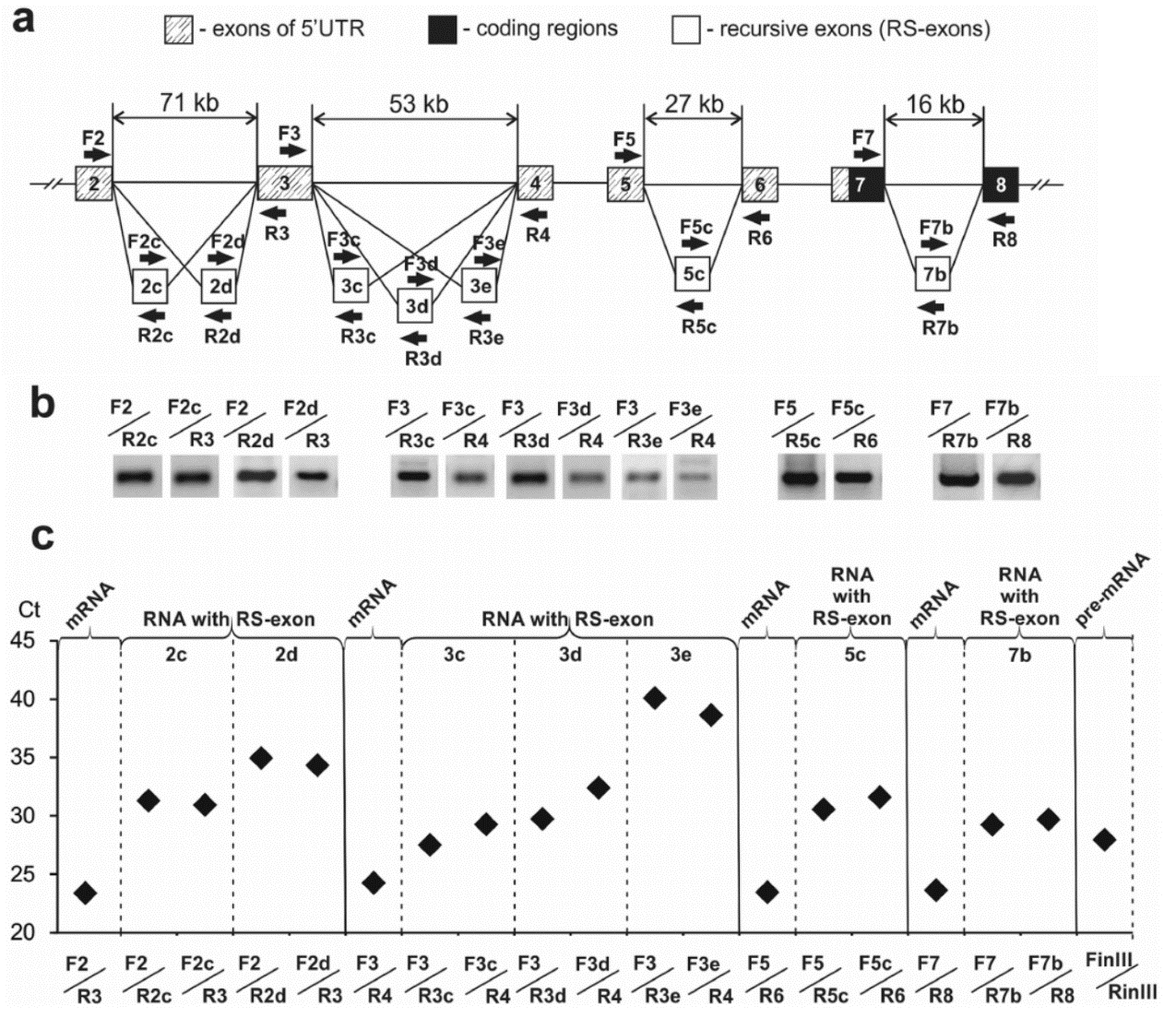
图4 SMS1基因可变剪切的PCR验证(来自[1])
SMS1基因的递归外显子也可在对应circRNA中存在
作者在之前的研究中就曾发现SMS1基因的递归外显子(2c, 3c和7b)可最终保留在对应的circRNA分子中。本文新发现的递归外显子是否也可存在与circRNA中?作者通过RNase R消化的方式鉴定到这些递归外显子的确可以在circRNA产物中存在。作者还在更多组织来源的RNA样品中进行了分析,结果表明特定的circRNA在不同组织中的表达差异非常明显。
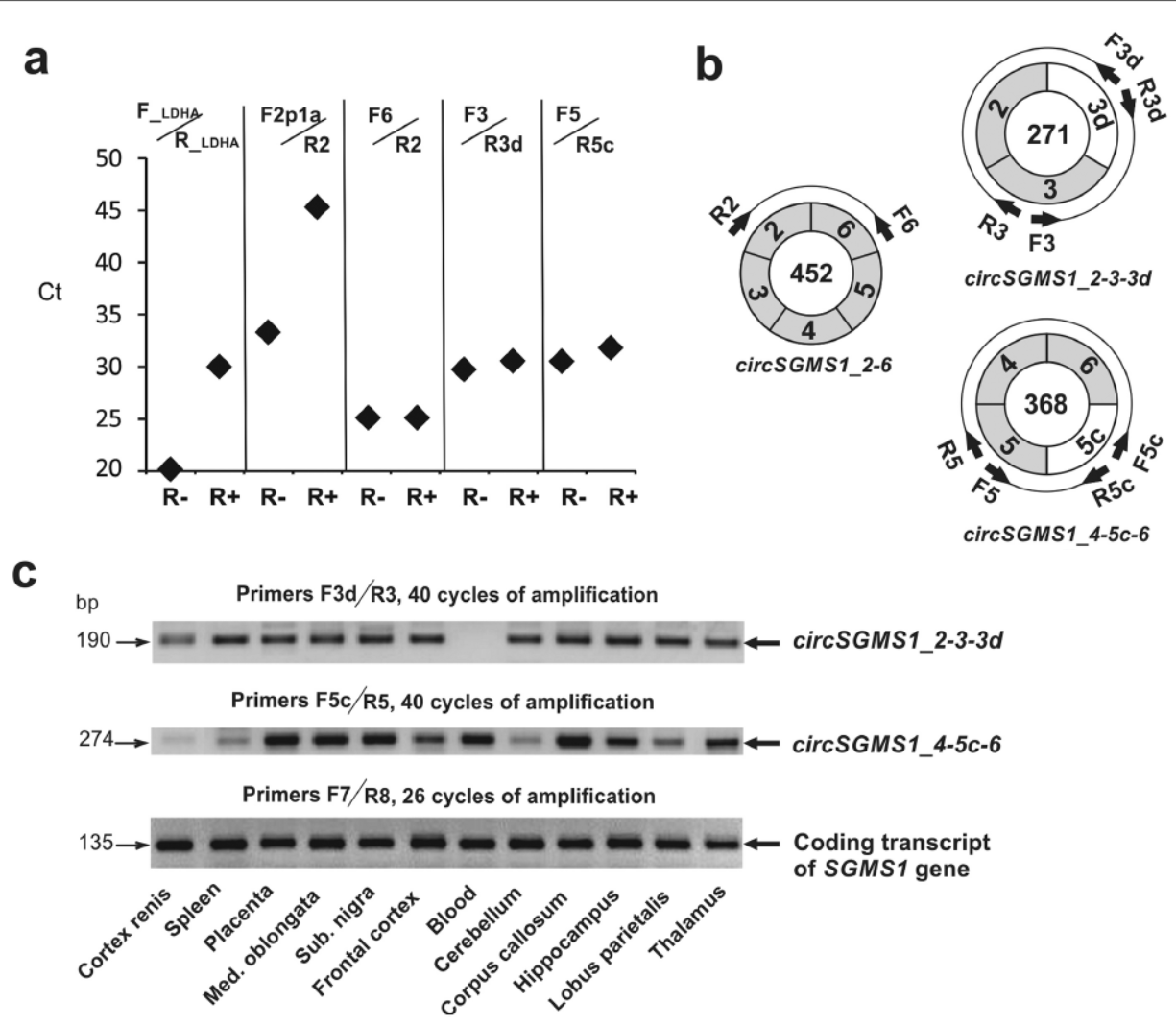
图5 递归外显子也可在circRNA中存在(来自[1])
根据已报道的和本文的发现,作者描绘出了SMS1基因来源的circRNA形成示意图。
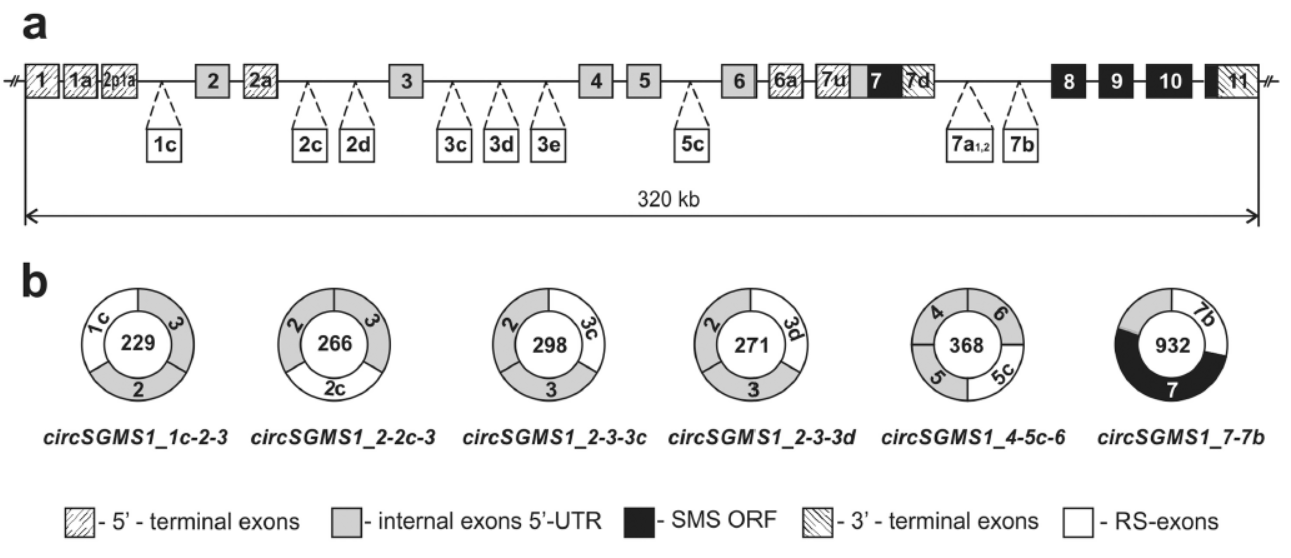
图6 SMS1基因来源的circRNA(来自[1])
基于这些circRNA所对应的内含子,作者描绘了SMS1基因通过多步拼接的方式形成circRNA的机制示意图。
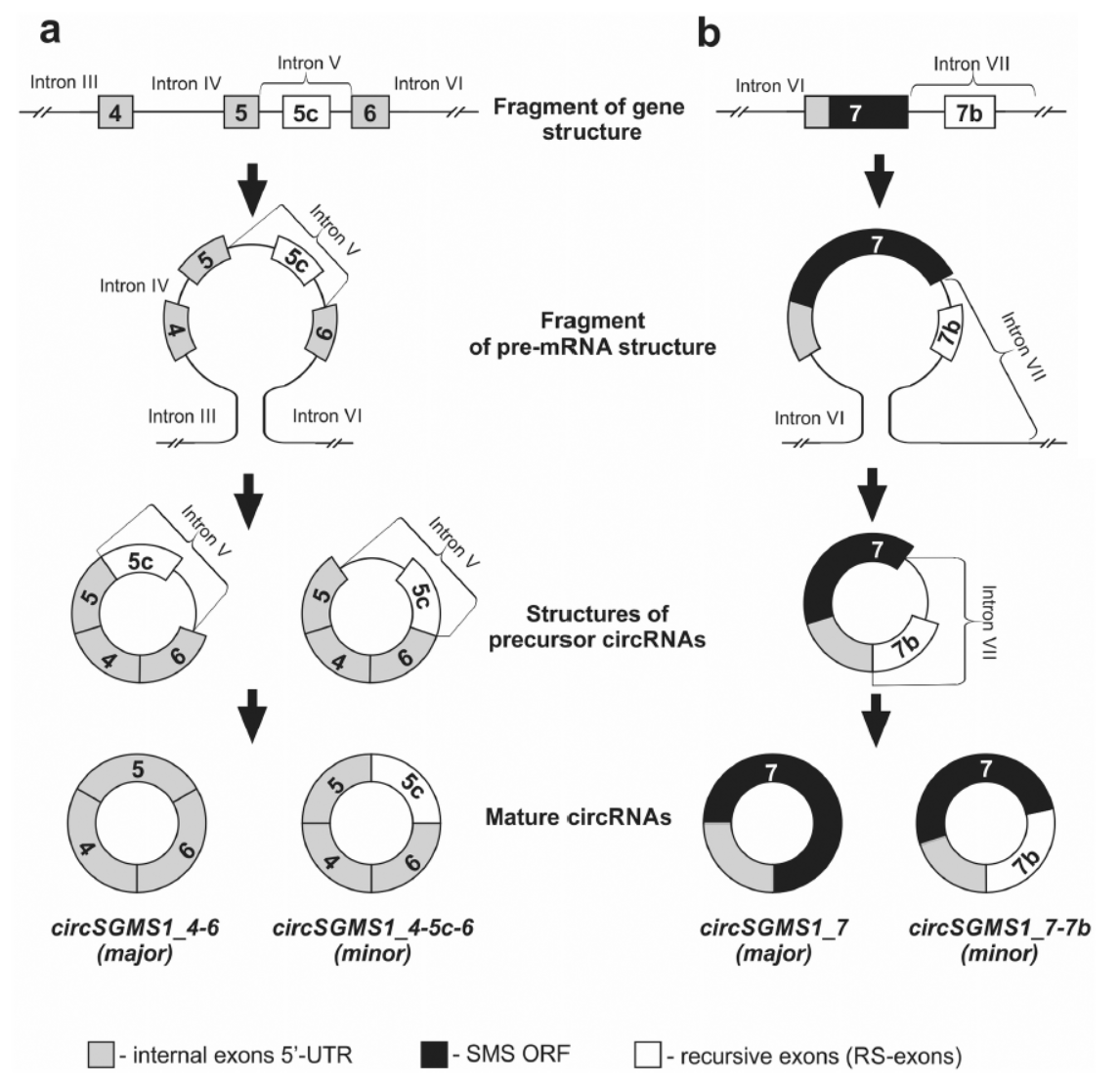
图6 SMS1基因来源的circRNA形成机制(来自[1])
SMS1基因的递归外显子在大鼠和小鼠中保守
在UCSC数据库中作者分析了人类SMS1的递归外显子2d, 3c, 3e 和7b在大鼠和小鼠中的保守性,发现这些外显子均非常保守。RT-QPCR也验证了在大鼠和小鼠脑组织来源的RNA中可检测出对应的可变剪切产物。
本文是基因可变剪切的研究报道,其中涉及到了circRNA的可变剪切问题。从本文的结果来看,circRNA的形成过程是基因转录过程伴生的,circRNA中存在的可变剪切外显子或许也是线性RNA剪切过程中会出现的过渡形式所携带的。这为深入理解基因的可变剪切,circRNA的可变剪切,组织特异性的可变剪切和表达状态提供了非常好的借鉴。
参考文献:
1. Filippenkov, I.B., et al., Multi-step splicing of sphingomyelin synthase linear and circular RNAs. Gene, 2018.
2. Clark, M.B., et al., Quantitative gene profiling of long noncoding RNAs with targeted RNA sequencing. Nat Methods, 2015. 12(4): p. 339-42.
.png)