目前circRNA的高通量鉴定基本都是基于识别反向拼接位点(Junction Point)而进行的,而就在1月27日的Nucleic Acids Research杂志在线发表了一篇研究论文,报道非共线性(Non-Co-Liner, NCL)转录产物不仅包含传统的circRNA,还可能是分子间反式拼接的产物。该文章的通讯作者是台湾中央研究院基因组研究中心的Trees-Juen Chuang[1]。

相关概念和背景介绍
什么是非共线性转录产物?
绝大多数真核生物的mRNA的外显子会按照基因组中上下游的顺序拼接形成最终的成熟mRNA,但有些特殊的情况下会出现与基因组序列顺序不一致的转录产物,比如大家都感兴趣的circRNA,这种产物就成为非共线性(Non-Co-Liner, NCL)转录产物。
NCL产物理论上有两种形式:同一分子内反向拼接形成的circRNA和分子间反式拼接的产物,本文称为反式拼接RNA(trans-spliced RNA, tsRNA)。
什么是反式拼接?
经典遗传学有“顺反子”的概念,顺式(cis-)作用和反式(trans-)作用也是从这一概念延伸而来。简而言之,顺式作用可以理解为同一个分子的上下游相互的作用即为顺式作用。而不同分子之间的作用就可以理解为反式作用。反式拼接就是反式作用的一种形式,也就是两个不同的RNA分子之间拼接到一起的情况。
不难理解,circRNA研究中的反向拼接其实是一种顺式作用的拼接形式,是同一个分子内的RNA选择性拼接方式。而本文中所提到的反式拼接其实是两个RNA分子最终拼接到一起的情形:
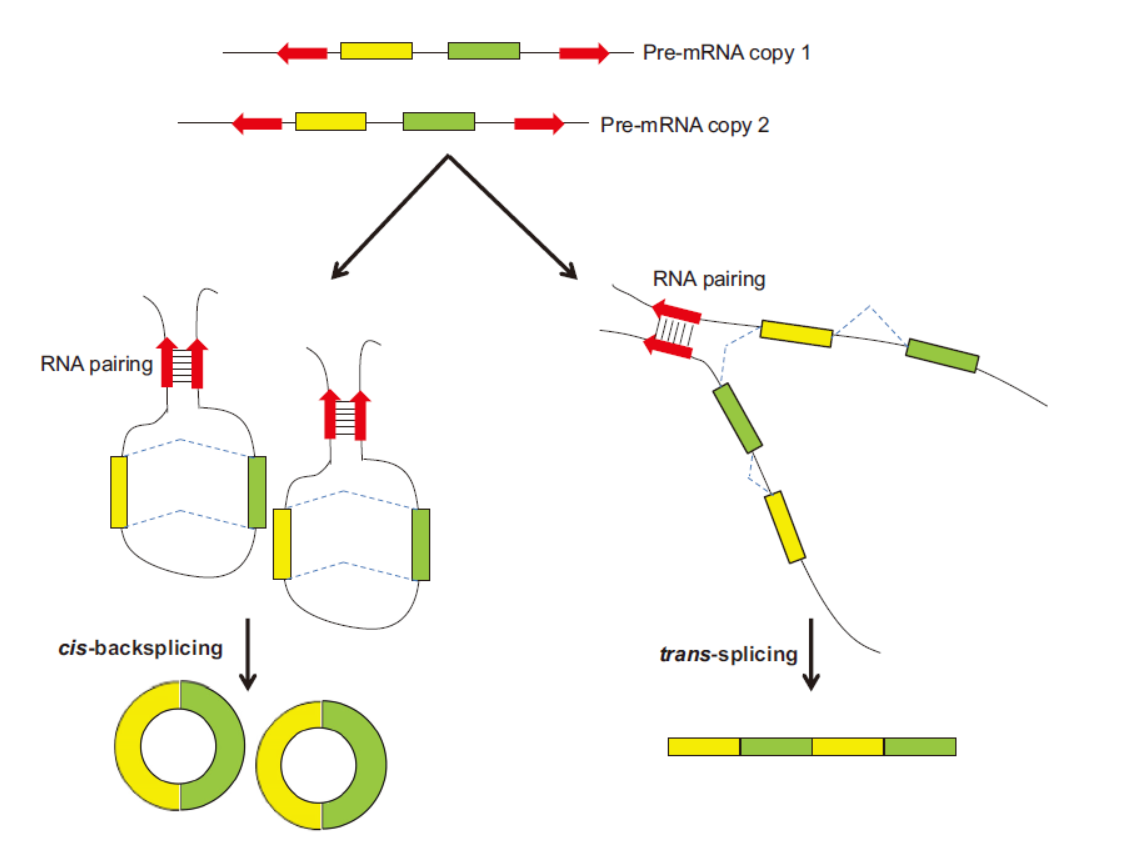
图1 反式拼接与反向拼接 (来自[1])
从示意图中也能看出,在反式拼接的产物(trans-spliced RNA, tsRNA)也存在与circRNA一样的Junction Point!这可不是闹着玩的,这意味着我们需要反思:我们正在辛辛苦苦研究的circRNA会不会是tsRNA,或者混杂着tsRNA?
本文的作者是如何发现并证明tsRNA的?怎样区分tsRNA和circRNA?tsRNA有什么特征和规律? tsRNA对circRNA的研究会带来什么影响?带着这一系列问题,让我们一起来学习一下这篇文章:
如何发现tsRNA的?
本文一开始是为了探索NCL转录产物存在的可能性及表达丰度的问题,首先选择了ENCODE中的7种人类细胞株的信息,这些细胞株均对应有胞质/核亚细胞组分的分析及Poly(A)-selected和Non-Poly(A)-selected 的测序数据。
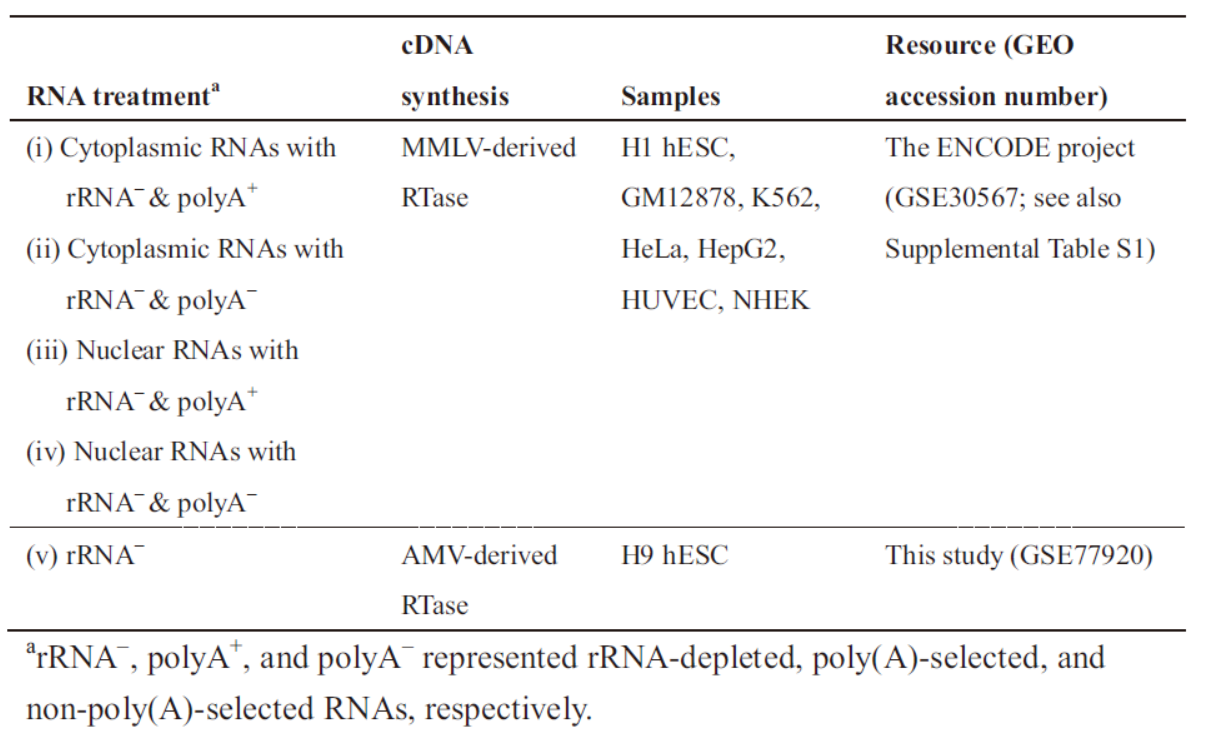
表1 本文用到的细胞测序数据 (来自[1])
作者使用的工具是NCLscan,主要分析了基因内的符合已注释的外显子边界的情况。NCLscan是本文作者在2015年发表的一个专门分析基因非共线性转录产物的工具[2]。为提高所分析的结果的可信度和准确性,专门挑选了基因内部的,符合已知的外显子序列边界的拼接产物进行分析。七种细胞中分别分析得到了2000~9000种不等的NCL转录产物。这些NCL产物对应于6731种基因,其中绝大多数(6542种,占97%)为蛋白编码基因。归一化处理后发现NCL的占比明显低于常规线性转录本,大约10-20倍的差距。hESC是NCL最高的细胞类型。(难道NCL与细胞的多能性有关?还不得而知)
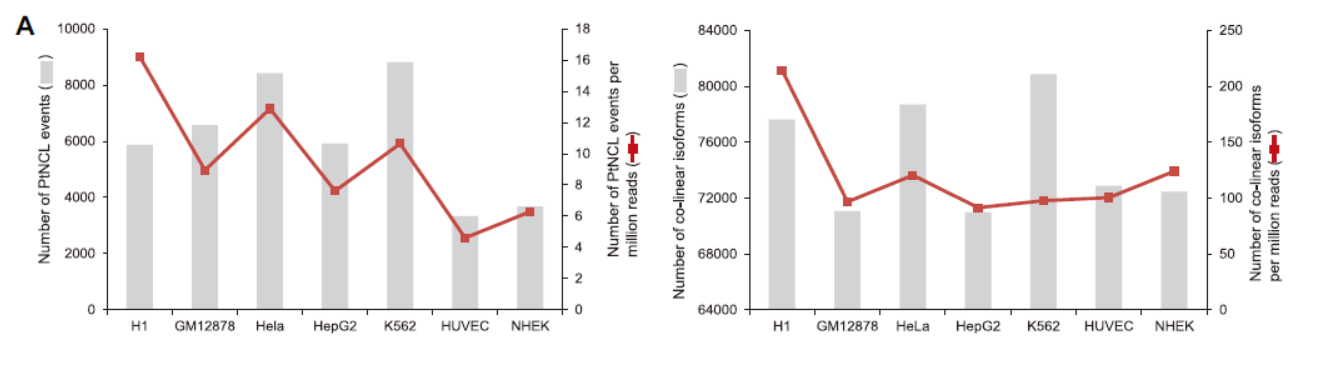
图2 NCL产物与对应基因的表达情况 (来自[1])
NCL的产物可分为分子内NCL产物(circRNA)和分子间NCL产物(tsRNA),两者最明显的区别是tsRNA是线性的RNA分子,有典型的Poly(A)结构,而circRNA没有。因此在测序数据中通过比较Poly(A)-selected和Non-Poly(A)-selected 的测序结果就可以有效的区分二者。tsRNA形式仅存在于Poly(A)-selected的测序结果中,circRNA形式仅存在于Non-Poly(A)-selected的测序结果中,两种测序数据都能找到的是同时存在两种形式NCL的位点。
tsRNA有什么特征?
接下来作者分别从NCL位点对应的基因组分布情况,与所对应基因的关系等不同角度分析了NCL产物的特征,进而分析circRNA和tsRNA的特征。概括而言,NCL具有以下几个特点:(1) NCL的发生并非完全随机的,存在热点;(2) NCL位点更倾向于分布在SCE元件覆盖区域;(3) 大部分NCL位点倾向于形成circRNA相关的形式,其中一部分会同时有circRNA和tsRNA两种形式,只有非常少的NCL位点是仅仅形成tsRNA形式的;(4) 反向互补Alu元件能够促进tsRNA和circRNA的生成;(5) 相对于circRNA形式,tsRNA表达与常规线性产物表达量相关性更强,但丰度更低,稳定性也较差;(6) tsRNA和circRNA的亚细胞分布有一定的区别。
1. NCL存在热点:
在能检测到NCL产物的基因中,约32-50%的基因存在两个或多个NCL的产物,说明NCL的过程不是随机的,存在热点位点。在所有检测到的24498个NCL信号中,23.5%的供体端和26.7%的受体端存在多种NCL产物形式,说明这些位点能够形成不同的NCL产物。
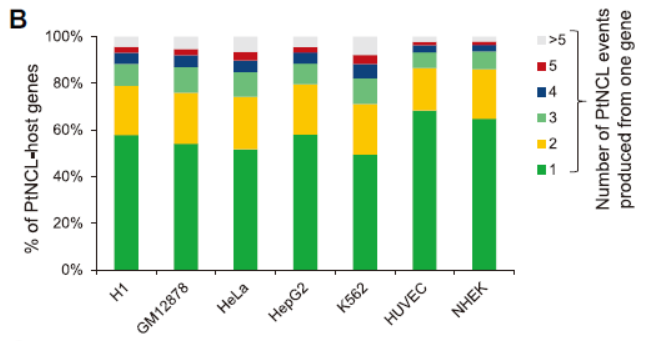
图3 NCL存在热点 (来自[1])
2. NCL位点更倾向于分布在SCE元件覆盖区域:
通过进一步分析筛选到的NCL位点的序列特点,作者发现NCL位点倾向于分布在结构基因的SCE元件区域。SCE元件(Synonymous constraint elements)是描述蛋白编码基因进化特征的一个概念,基于进化保守性分析和群体遗传分析,一些特定的编码序列会存在相对较低的同义密码子多态性,这些区域就定义为SCE元件。SCE元件所对应的编码蛋白的功能还没有完全研究清楚,但或许与蛋白的高级结构或RNA的高级结构及转录调控等有一定的关系[1, 3]。通过相关性分析表明,NCL位点更倾向于分布在SCE元件的覆盖区域。这一特征还体现在能鉴定到过个NCL的热点位点比仅有一次NCL的位点更高,同一位点出现的Reads数越高的,越倾向于对应在SCE覆盖区域。
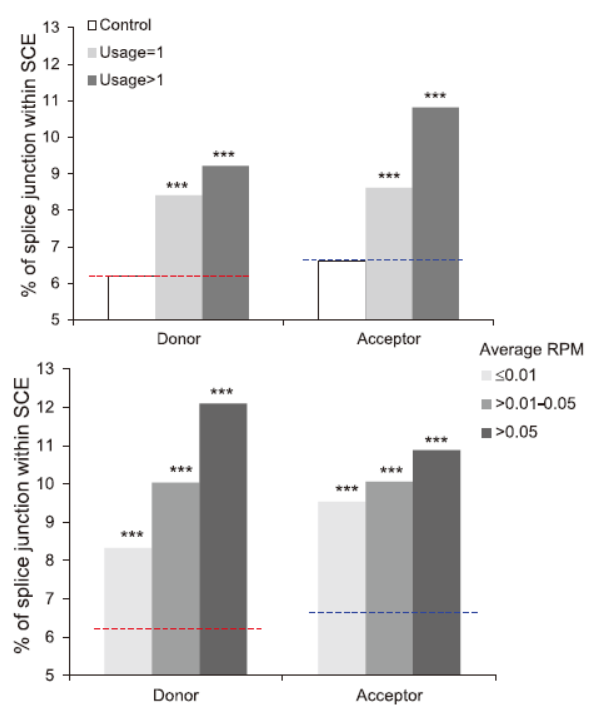
图4 NCL位点更倾向于分布在SCE元件覆盖区域 (来自[1])
进一步,作者比较了NCL产物与对应的常规线性转录本产物的丰度的差异情况,结果表明NCL的表达丰度普遍非常低,绝大部分均明显低于经典的线性转录本。但作者也发现了362个丰度相对较高的NCL产物(RNCL >0.1),并且有34个NCL产物的丰度甚至高于对应的经典的线性转录本。这些高表达的NCL产物有一定的细胞特异性分布。
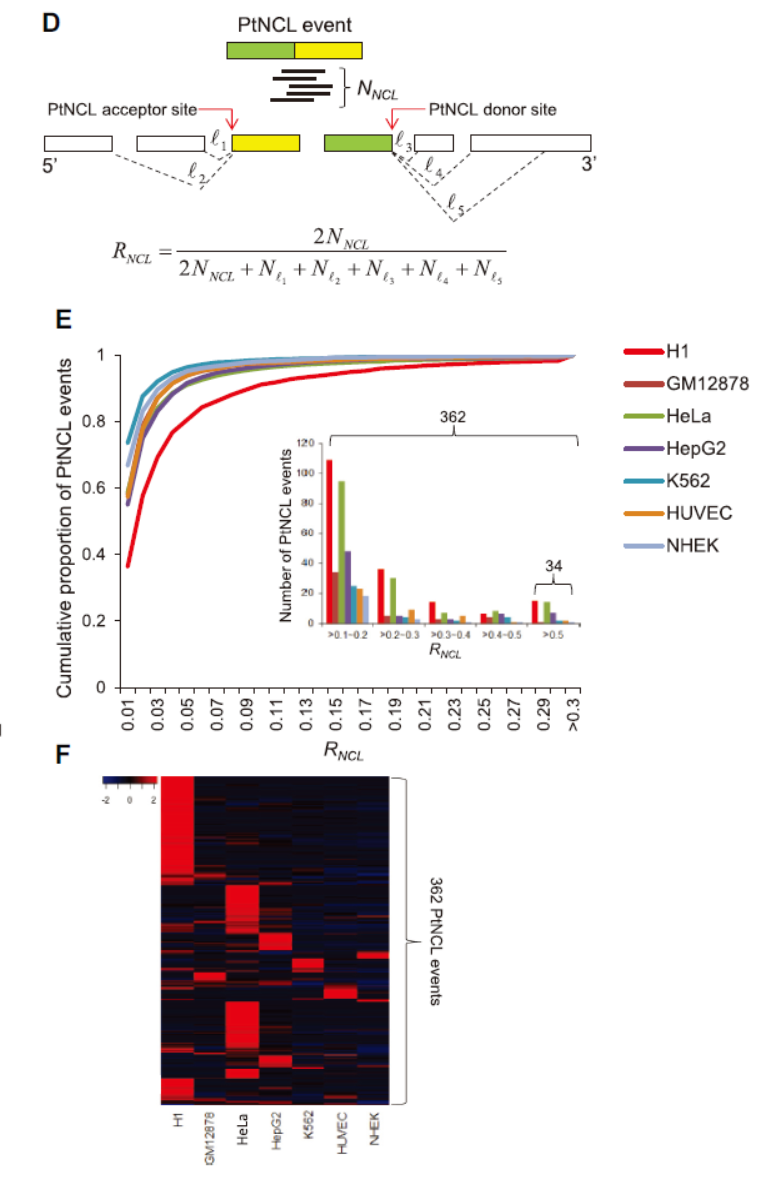
图5 NCL产物表达量分析(来自[1])
3. NCL位点形成circRNA和tsRNA的倾向性有所不同:
基于比较同一细胞的Poly(A)-selected和Non-Poly(A)-selected 的测序结果,将NCL产物分为三种不同的类型:仅有tsRNA产物的(TS-only)、仅有circRNA产物的(circRNA-only)和两者都有的(TS-circRNA),分析表明,circRNA-only占到总NCL产物类型的61-79%,TS-circRNA占到20-35%,TS-only不到5%。将不同测序数据基于Reads数归一化后分布依然类似。
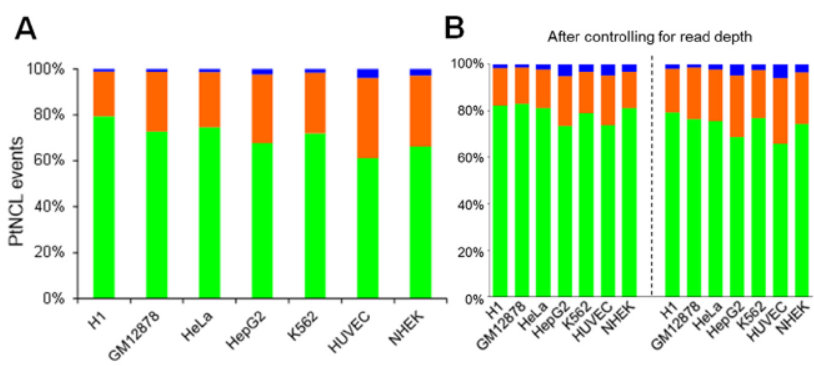
图6 不同NCL产物的表达分布情况 (来自[1])
上述结果都是直接下载了已经上传的测序数据分析得到的,还不足以证明tsRNA否真实存在,一个关键的技术问题是会不会是由于实验过程中的一些不确定因素造成的干扰信号?比如会不会是反转录过程中RNA模板分子的转换(template switching)造成的?或者样品制备过程中处理不彻底而掺杂有非特异性的RNA并最终造成结果的干扰?要证明tsRNA的真实存在,必须排除这些干扰情况的可能性。作者用人的胚胎干细胞H9进行了AMV反转录后测序,分析NCL情况,与已有的H1(MMLV反转录的结果)进行比较分析,找出两者均检测到的NCL,结果表明三种类型的分组丰度分布与上述结果基本一致,可以证明在不同反转录条件下均能找出tsRNA的存在证据。
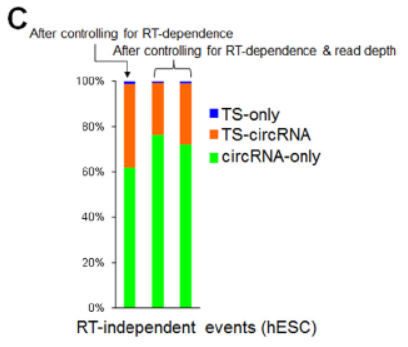
图7 不同反转录条件验证三类NCL的表达分布情况 (来自[1])
针对样品制备过程可能存在的处理不彻底或样品污染等不确定因素造成的干扰,作者设计了三个方案进行分析。首先利用信息学工具结合统计分析样品处理条件的干扰问题,接下来直接通过实验验证样品处理过程中这些可能的干扰因素是否能影响检测的结果,最后利用三代测序的方法验证了tsRNA的真实存在。
我们首先来看如何利用信息学工具结合统计分析样品处理条件的干扰问题。从已有的测序数据中分别分析三类NCL在Poly(A)-selected和Non-Poly(A)-selected测序数据中的表达情况。挑选ES细胞中不受反转录条件影响的NCL进行比较,选择了两个分析条件:首先,从非ES细胞的Non-Poly(A)-selected测序数据(理论上对应于circRNA相关的NCL形式)中找出对应的在ES中表现为TS-only和TS-circRNA的NCL位点,如果这些位点是Poly(A)去除不彻底导致的,TS-only和TS-circRNA两种形式的NCL的含量应该是比较接近才行,然而,结果表明两者具有非常显著的差异。反过来,在非ES细胞的Poly(A)-selected测序数据(理论上对应于liner RNA形式的tsRNA)中找出对应的在ES中表现为circRNA和TS-circRNA的NCL位点,如果这些位点是Poly(A)富集过程中掺杂的circRNA形式的分子,所对应的circRNA和TS-circRNA含量也应该是接近的才合理,结果也表明两者差异也非常显著。综合两种分析,这些差异并不能仅仅用样品处理过程的不确定因素来解释。
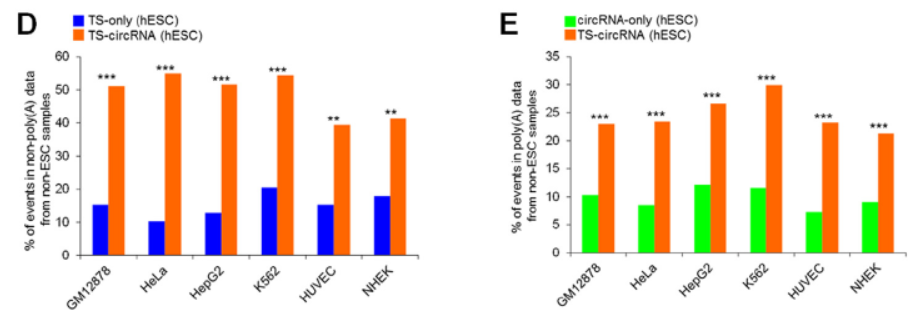
图8信息学结合统计分析样品处理条件的干扰问题(来自[1])
实验验证方案,作者挑选了H9中存在的42种TS-circRNA形式的NCL进行验证,QPCR结果表明它们都可明显耐受RNase R的消化,说明存在circRNA的形式。与此同时,它们均能在Poly(A)-selected的条件下(利用oligo-dT进行Pull down)被富集,说明存在Poly(A)尾巴,也有tsRNA的形式,在此基础上进行RNase R消化后几乎所有的NCL都被消化掉了。但TBC1D31的似乎有点奇怪,原因不明。这个实验就清楚的证明了TS-circRNA形式的NCL在细胞中既能被加工成circRNA,也可以被加工成线性的tsRNA。
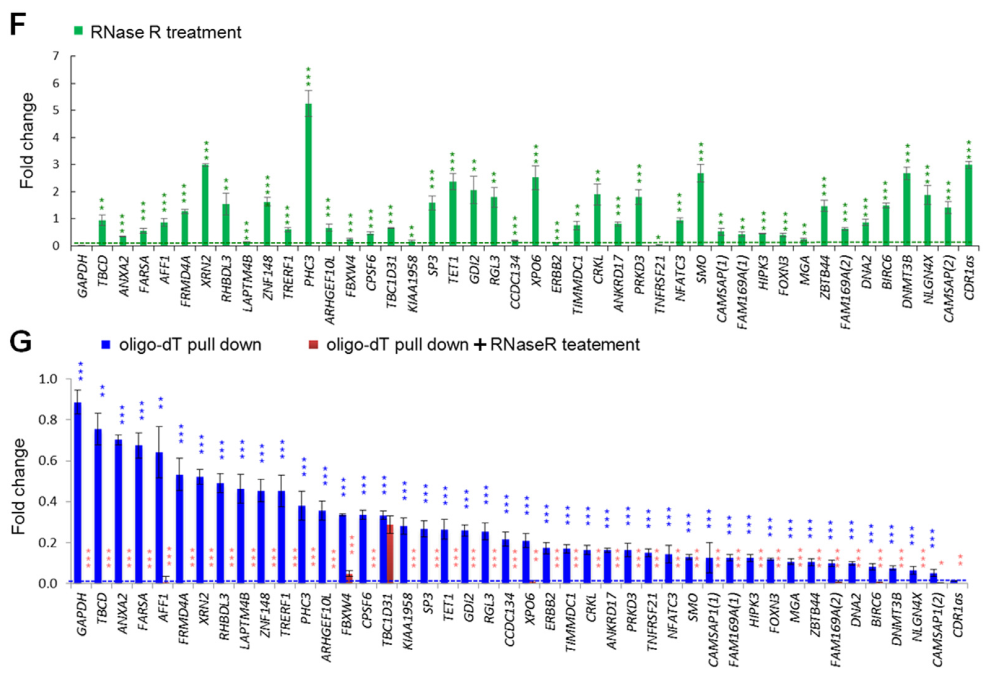
图9 H9细胞中TS-circRNA鉴定分析(来自[1])
最后,作者借助三代测序进行了TS-circRNA的测序分析。在Hela细胞中通过Poly(A)-selected建库测序,在牛津孔MinION测序仪中进行测序分析。挑选了FARSA、 HIPK3 和CAMSAP1的TS-circRNA进行分析,找到了有别于circRNA形式的线性tsRNA来源的Reads。说明细胞的确存在内源性tsRNA。
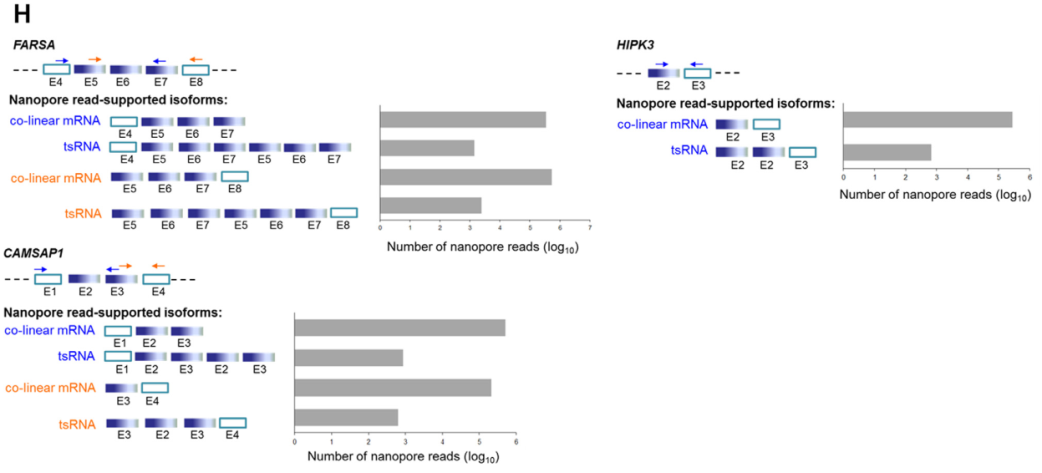
图10 三代测序验证内源性tsRNA(来自[1])
4. tsRNA与circRNA的形成机制相似
经过汇总统计,共分析到8659种可形成tsRNA的NCL位点,其中绝大部分(93%)同时也存在circRNA形式,仅有7%为TS-only的形式。进一步的分析表明,tsRNA和circRNA类似,也倾向于在基因的中间位置的外显子中形成,外显子和对应的内含子长度分析也显示与circRNA的形成有相似之处。值得一提的是,tsRNA的形成也与反向互补Alu元件有密切关系。
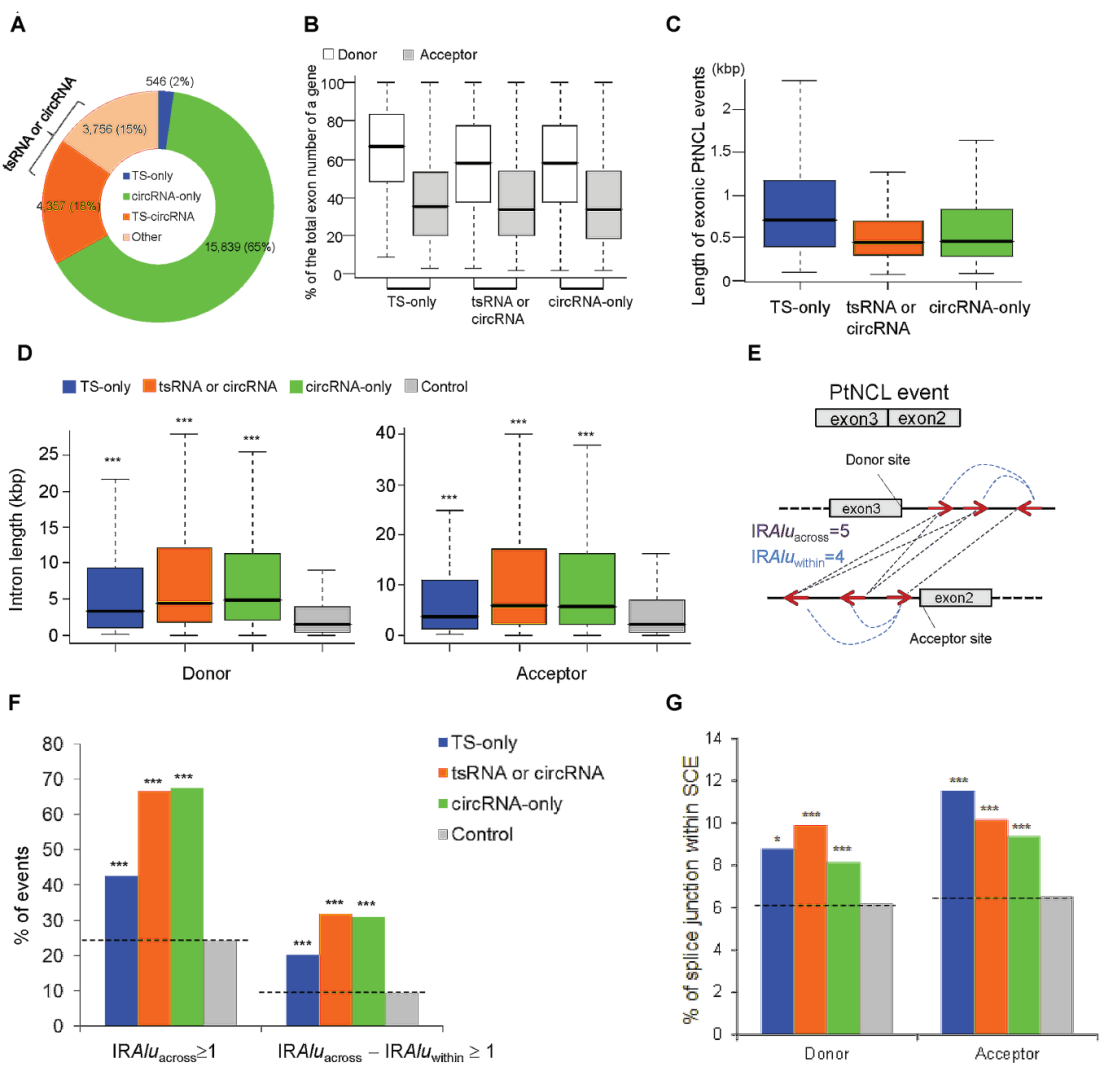
图11 tsRNA与circRNA的形成机制相似(来自[1])
接下来,作者通过构建特定TS-circRNA位点的载体分析反向互补Alu元件对这类位点形成tsRNA或circRNA的影响情况。选择了POLR2A基因进行分析。RT-PCR,RNase R消化分析,结合Poly(A)-selected和Non-Poly(A)-selected及三代测序分析表明该基因同时存在tsRNA和circRNA两种NCL形式。基于人工构建的载体证明,tsRNA和circRNA的形成均依赖反向互补Alu元件。
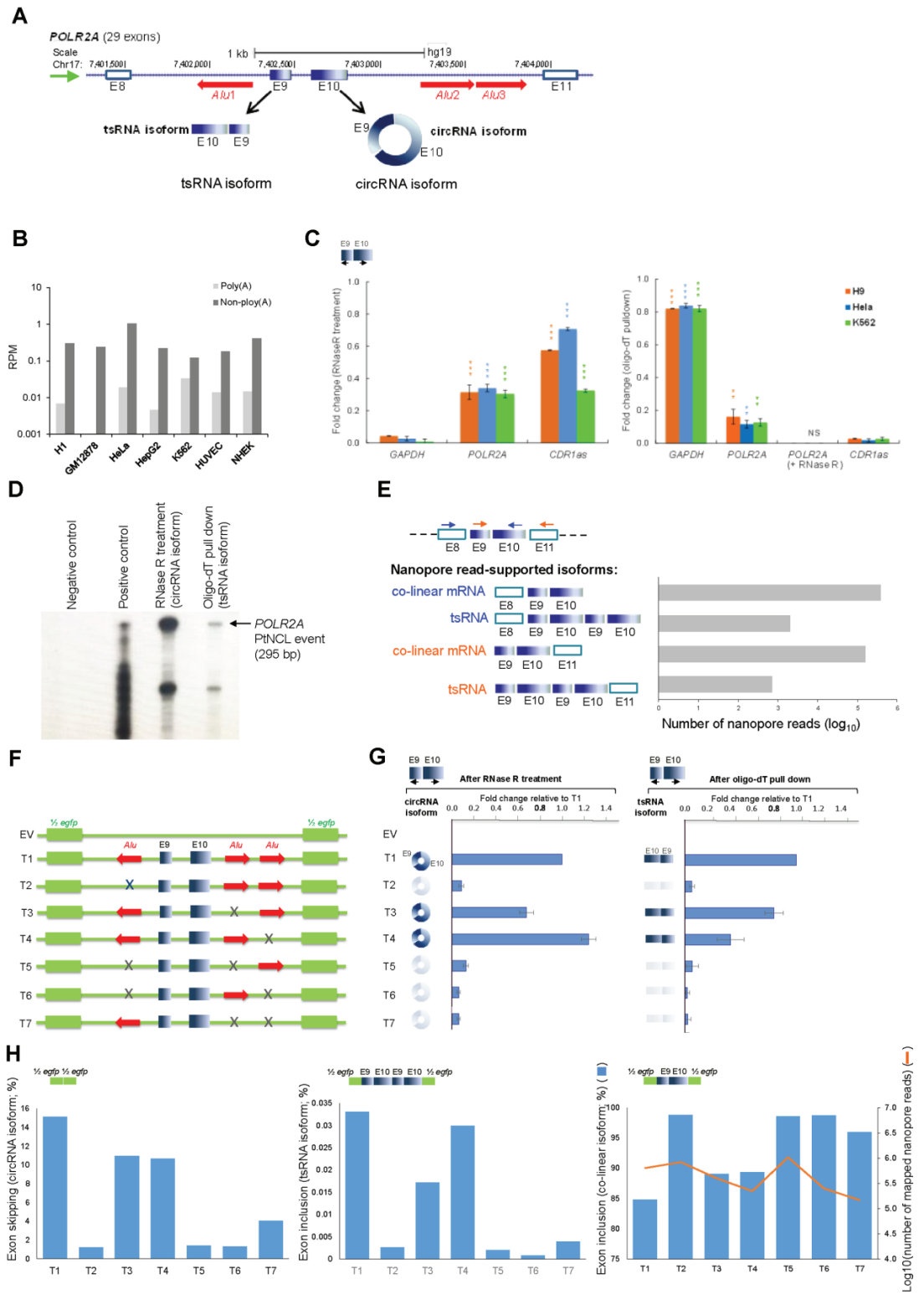
图12 tsRNA和circRNA的形成均依赖反向互补Alu元件(来自[1])
5. 相对于circRNA形式,tsRNA表达与常规线性产物表达量相关性更强,但丰度更低,稳定性也较差
两种NCL形式与母基因的表达关系是怎样的呢?作者基于Spearman’s秩相关系数分析,结果表明它们有微弱的相关性(P ≈ 0.2),进一步,利用偏相关分析的方法分别分析两种NCL形式与对应母基因的表达量相关性变化。当将tsRNA设为控制变量时,相关性明显降低,而当将circRNA设置为控制变量时,相关性明显增强。这说明tsRNA和circRNA的形成过程没有太多的相关性,也就是说两种形式的分子是独立形成的。而相对于circRNA,tsRNA的表达量与与母基因表达量的更有相关性。细胞类型特异性的分析表明circRNA形式的NCL丰度远高于TS-only形式,在TS-circRNA类的NCL中也是以circRNA形式的占绝大多数。稳定性分析表明tsRNA更不稳定。
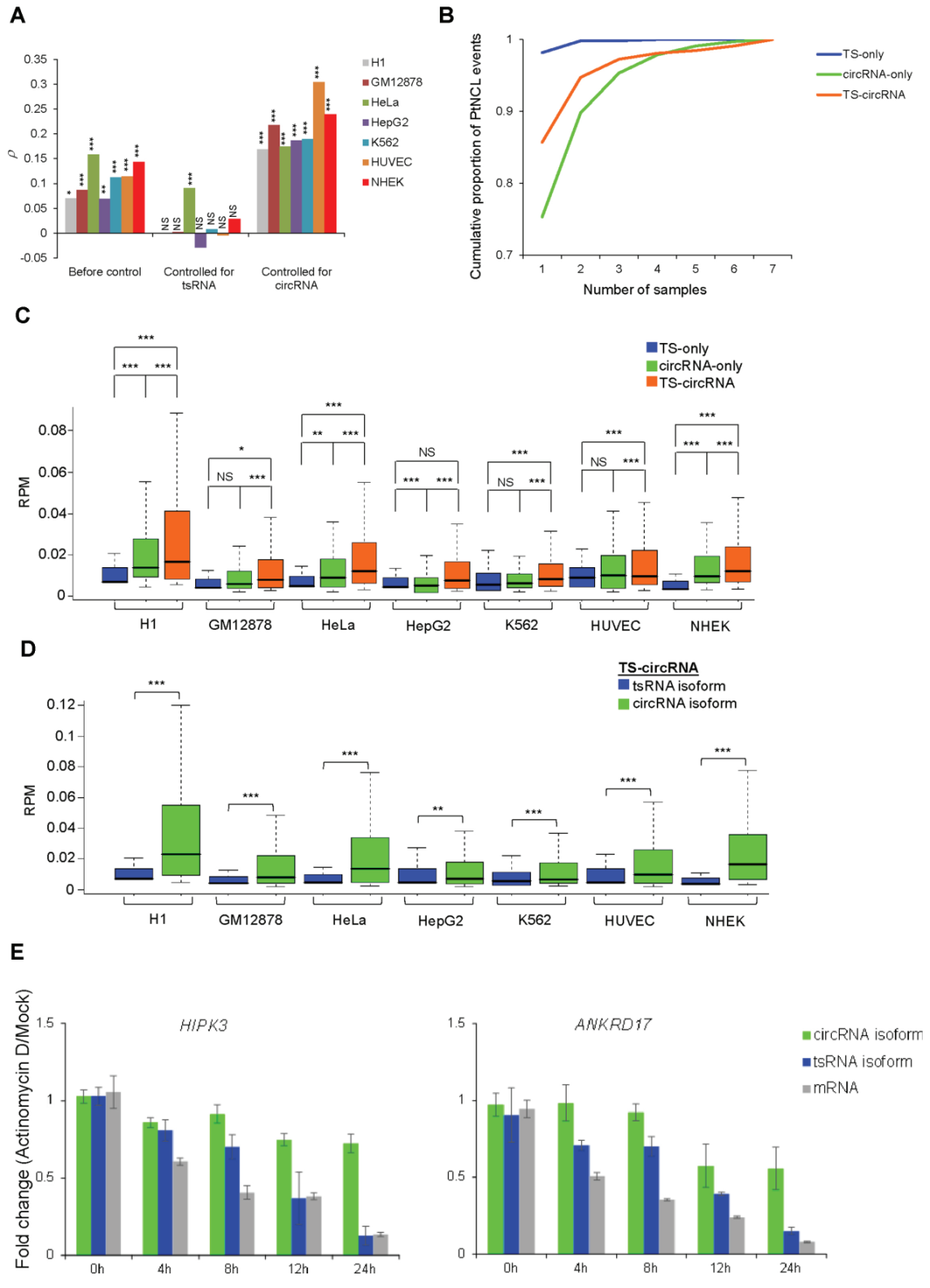
图13 tsRNA的表达量稳定性分析(来自[1])
6. tsRNA与circRNA的亚细胞定位有一定的差别
通过比较细胞质与细胞核测序的结果表明tsRNA和circRNA亚细胞定位存在一定的差异。进一步基于亚细胞组分分离和RNase R消化等实验进一步分析验证了两种RNA分子的亚细胞定位情况。FISH实验也进一步验证了相关的结果。最终表明tsRNA与circRNA的亚细胞定位不完全一致,暗示两类分子可能有不同的功能。
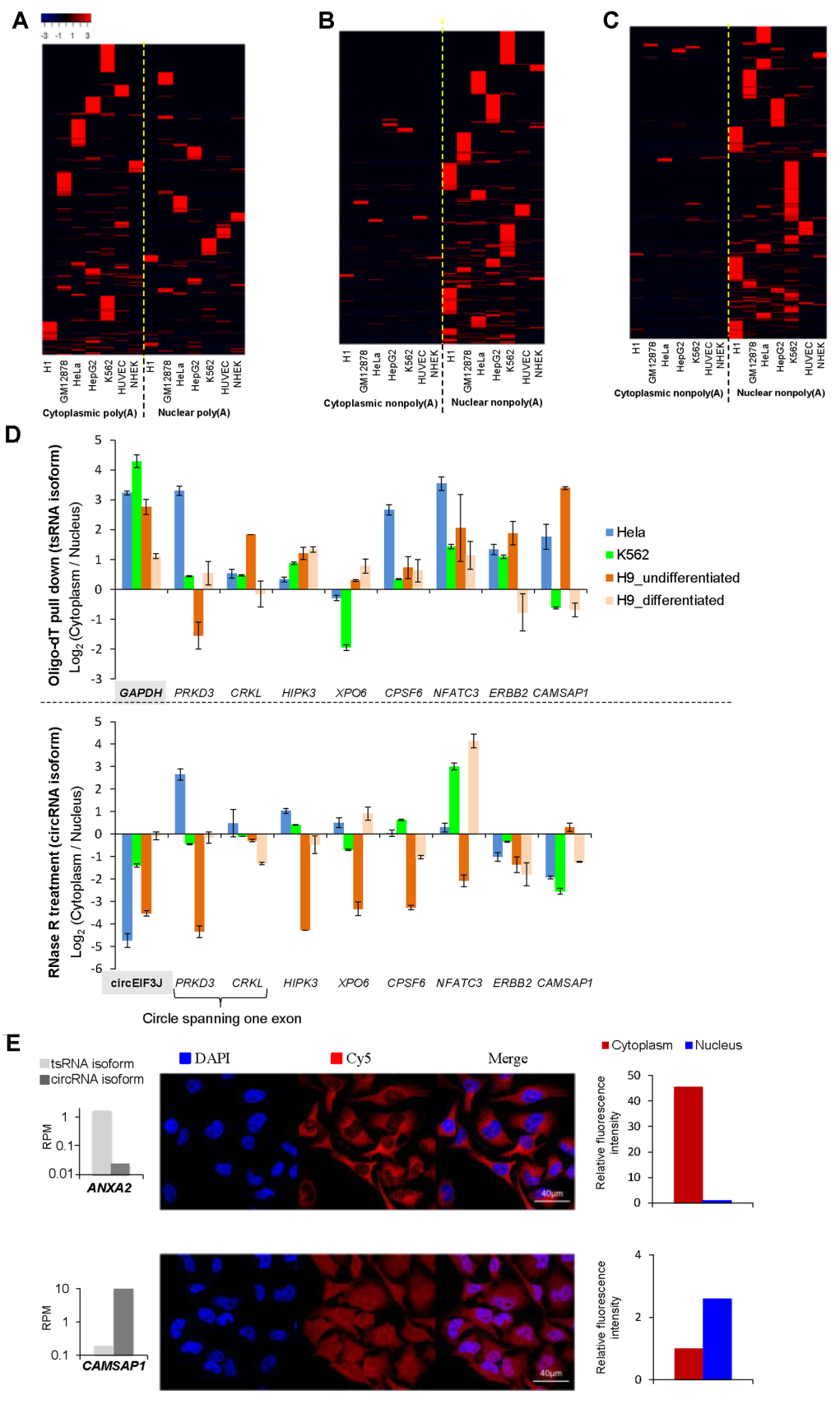
图14 tsRNA与circRNA的亚细胞定位有一定的差别(来自[1])
同位点tsRNA的发现对circRNA研究带来什么启发?
tsRNA并非本文首次报道,但将tsRNA的表达与circRNA的表达进行比较和分析具有非凡的意义,对circRNA的研究有重要的启发价值。区分tsRNA和circRNA其实是很简单的,tsRNA是依然是线性的RNA分子,带有Poly(A)尾,可以通过oligo-dT进行富集和反转录验证,而circRNA理论上耐受RNase R的消化,可以通过RNase R消化加以区分。
从本文的结果来看,tsRNA的丰度普遍不高,且往往与circRNA伴生,在研究特定circRNA的时候需要增加分析是否存在tsRNA的步骤,如果证明的确有tsRNA,则需要进一步分析tsRNA和circRNA的表达丰度,结合RT-PCR后Sanger测序等技术的手段弄清楚tsRNA和circRNA的序列,在此基础上设计针对不同分子形式的siRNA,这对于解释相关的机制非常重要。
参考文献:
1. Trees-Juen Chuang, Y.-J.C., Chia-Ying Chen, Te-Lun Mai, Yi-Da Wang, Chung-Shu Yeh, Min-Yu Yang, Yu-Ting Hsiao, Tien-Hsien Chang, Tzu-Chien Kuo, Hsin-Hua Cho, Chia-Ning Shen, Hung-Chih Kuo, Mei-Yeh Lu, Yi-Hua Chen, Shan-Chi Hsieh and Tai-Wei Chiang, Integrative transcriptome sequencing reveals extensive alternative trans-splicing and cis-backsplicing in human cells. Nucleic Acids Research, 2018.
2. Chuang, T.J., et al., NCLscan: accurate identification of non-co-linear transcripts (fusion, trans-splicing and circular RNA) with a good balance between sensitivity and precision. Nucleic Acids Res, 2016. 44(3): p. e29.
3. Lindblad-Toh, K., Garber,M., Zuk,O., Lin,M.F., Parker,B.J., Washietl,S., Kheradpour,P., Ernst,J., Jordan,G., Mauceli,E. et al., A high-resolution map of human evolutionary constraint using 29 mammals. Nature, 2011. 478: p. 476–482.
.png)