环状DNA是自然界普遍存在的一种DNA分子形式,例如细菌或酵母等微生物的基因组DNA、细菌质粒、线粒体DNA(mitochondrial DNA, mtDNA)等等都是环状DNA分子。真核生物中还有一类特殊的环状DNA分子,它们是从正常基因组中分离或脱落下来,游离于染色体基因组之外,以特殊的方式参与生理或病理过程。由于它们是在染色体之外独立存在的DNA分子,因此统称为染色体外DNA,又常常是环状的,因此称其为染色体外环状DNA(extrachromosomal circular DNA, eccDNA)。染色体外DNA在很多物种中均存在,包括酵母、线虫、果蝇、哺乳动物、植物等等([1])。eccDNA往往在肿瘤和衰老过程中富集,以特殊的方式参与肿瘤和衰老的发生发展进程([1]),因此备受关注。本文对eccDNA的发现历程、主要特征、功能机制和、研究方法进行汇总。
1. eccDNA的发现历程
eccDNA并不是最近才发现的,最早可追溯到1965年。当时Hotta, Y.等从麦胚和猪精子中发现了染色体外环状DNA分子([2])。同一年,Cox, D.等报道从人的肿瘤标本细胞中发现了染色体外的DNA分子,它们具体经常是成对出现的,因此当时称其为双微体(double-minutes)([3])。此后陆续在很多物种中都发现了染色体外环状DNA分子。随着分子克隆技术的发展和成熟,人们开始对双微体的分子功能展开了研究。1978年,Alt, F. W.等报道发现氨甲喋呤耐药的小鼠细胞存在携带DHFR基因的双微体,这些双微体的存在导致了DHFR基因的扩增,介导了小鼠细胞对氨甲喋呤的耐药([4])。这是首次报道双微体(eccDNA)的作用机制。1983年,Kohl, N. E.报道神经母细胞瘤中存在携带MYCN的双微体([5])。此后多项研究发现双微体可携带癌基因,包括EGFR和c-Myc等等([1])。多项研究也表明这种染色体外DNA分子常以环状DNA的形式存在,进一步的研究发现人类染色体外DNA其实只有大约30%的是以双微体的形式存在的,因此上面提到的双微体概念渐渐被染色体外环状DNA的表述替代了([1])。Episome是eccDNA的另一种表述形式,目前认为episome是eccDNA的前体分子形式([1])。

图1人类肿瘤细胞中存在双微体(eccDNA)([3])
人类基因组计划的完成和二代测序技术的兴起带动了eccDNA的发展。2012年,Shibata, Y.等报道从小鼠和人的细胞中发现了一类小的染色体外环状DNA分子,它们大小在200-400bp不等的染色体外环状DNA,但这些DNA分子与上述的eccDNA并不一致,他们称其为microDNA([6])。2014年,Paul S. Mischel团队发现胶质瘤细胞中存在携带EGFRvIII突变型基因的eccDNA,介导了EGFR抑制剂耐药([7])。2017年,Paul S. Mischel团队对17种肿瘤的2572种细胞系的全基因组测序分析,发现超过一半的人类肿瘤中存在eccDNA,这些eccDNA分子经常携带肿瘤驱动基因([8])。2018年,Moller, H. D.等报道从健康人的肌肉和血液细胞中分离到超过十万种的eccDNA分子,它们绝大部分都携带基因或基因的片段,长度往往小于25kb([9])。这一报道表明eccDNA可能普遍存在。2019年,Paul S. Mischel团队报道eccDNA存在显著的染色体开放状态和更远距离相互作用的形式,提高了对eccDNA的作用机制认识([10])。同期,Jeremy N. Rich和Peter C. Scacheri等对eccDNA中染色质结构和增强子状态进行了分析,表明eccDNA中非编码区的增强子功能也在eccDNA所携带的癌基因表达调控中发挥功能,也进一步表明了eccDNA特殊的染色质结构及其功能([11])。eccDNA研究过程中一些重要的发现可用下图的时间轴展示:
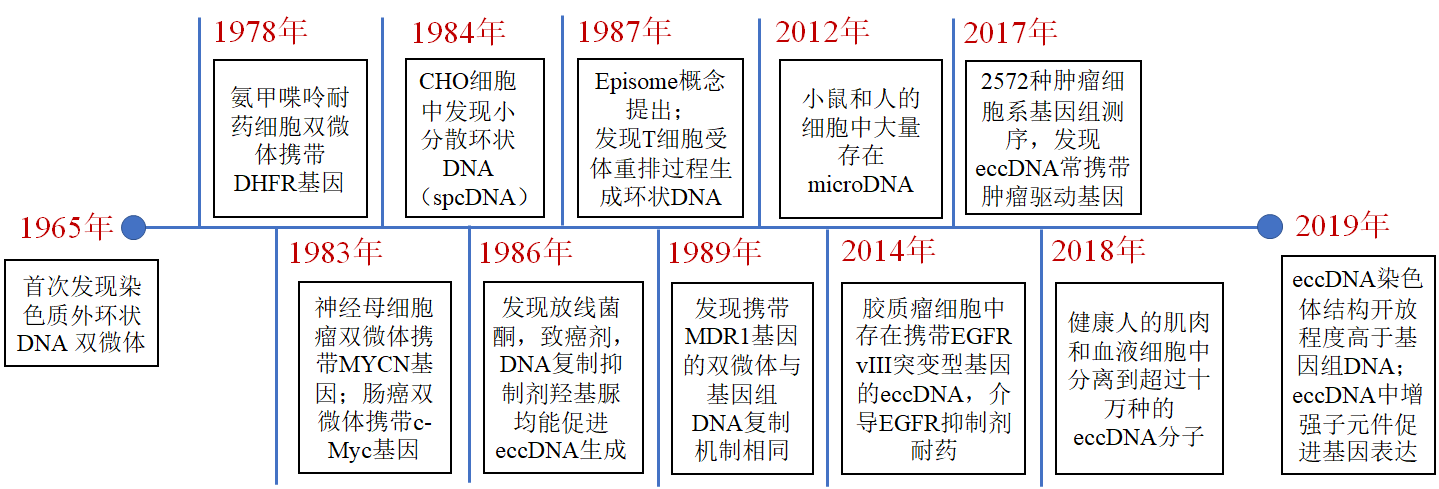
图2 eccDNA的主要研究历程
2. eccDNA的主要特征
2.1 染色体外环状DNA的大小和分类
染色体外DNA是泛指所有染色体基因组之外的DNA物质,也包含了线粒体DNA,一些病毒基因组环状DNA等。除了这些与核基因组序列不同的DNA分子,其余的根据分子大小大致可分为三类:(1)小于1kB的microDNA分子,包括小分散环状DNA(small polydispersed circular DNA,spcDNA),B-细胞或T-细胞成熟中形成的小环状DNA,端粒序列形成的小环状DNA分子,转座子元件形成的环状DNA分子等等,这类分子通常小于500bp,往往不携带完整的基因序列([1])。(2)从几kb到几Mb范围的eccDNA分子,目前对eccDNA的大小还没有定论,主要认为分布在1kb~5Mb范围内([10, 12]),但不同的文献报道中表述略有差别。它们往往是一些特殊的基因区域形成的,可携带完整的基因和特殊的调控元件,能独立完成复制过程,但不携带着丝粒和端粒结构。本文中若不特殊说明,所有的eccDNA均代表这一类的分子。(3)带有着丝粒和端粒结构的迷你染色体(minichromosomes)([1])。
2.2 eccDNA的结构特点
早期对eccDNA或双微体结构认识比较局限,主要发现它们是环状DNA分子,并且往往是一些基因扩增的产物,其中也包含着DNA复制起始点,因此可以独立完成复制作用。2019年,Paul S. Mischel团队的Nature文章以及Peter C. Scacheri与Jeremy N. Rich联合发表的Cell文章对eccDNA的结构有了较为深刻的探索分析,eccDNA不仅携带了完整的基因,还携带了基因上游的启动子和增强子元件([10, 11])。eccDNA的染色质结构更加松散,H3K27ac状态更高([11]),其中的电镜照片也清楚的显示eccDNA为游离在染色体之外的环状DNA分子([10])。
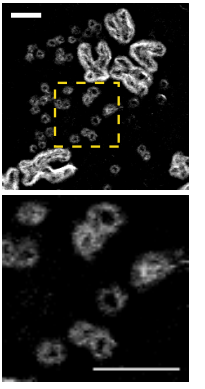
图3 eccDNA是一种环状的DNA分子 ([10])
2.3 eccDNA的形成机制
早期的研究就发现eccDNA是基因扩增的产物,是基因组不稳定性的一种表现形式。肿瘤基因扩增往往以两种方式,一种是在均质染色区域(homogeneously staining regions,HSR)进行基因的扩增,另一种则是形成eccDNA(双微体)([13])。目前关于肿瘤中HSR区和eccDNA基因扩增现象的分子机制还没有定论,一种可能的机制是基因组结构的一次“灾难性”事件(chromothripsis)有关([14])。早期的研究认为短重复序列是介导eccDNA形成的因素,主要通过基因重组机制实现,也有一些模型认为是在DNA复制过程中通过loop切除或在DNA复制暂停的时候复制泡中断裂的DNA片段连接形成([15])。早期的理论认为DNA复制是形成eccDNA所必需的,但有的研究中却发现DNA复制的抑制剂会促进eccDNA的形成,也有研究发现eccDNA的形成过程完全不依赖DNA复制作用([12])。有研究发现线菌酮,致癌剂或者DNA复制抑制剂(羟基脲)会促进eccDNA的生成([16]),范可尼贫血病人eccDNA显著增多,长度也更长([17]),这也说明eccDNA的形成可能是多种机制作用的结果。有趣的是c-Myc基因能促进eccDNA的生成([18])。Anindya Dutta等在他们的一篇综述文章中用示意图展示了各种可能形成eccDNA的分子模型,如下图所示:
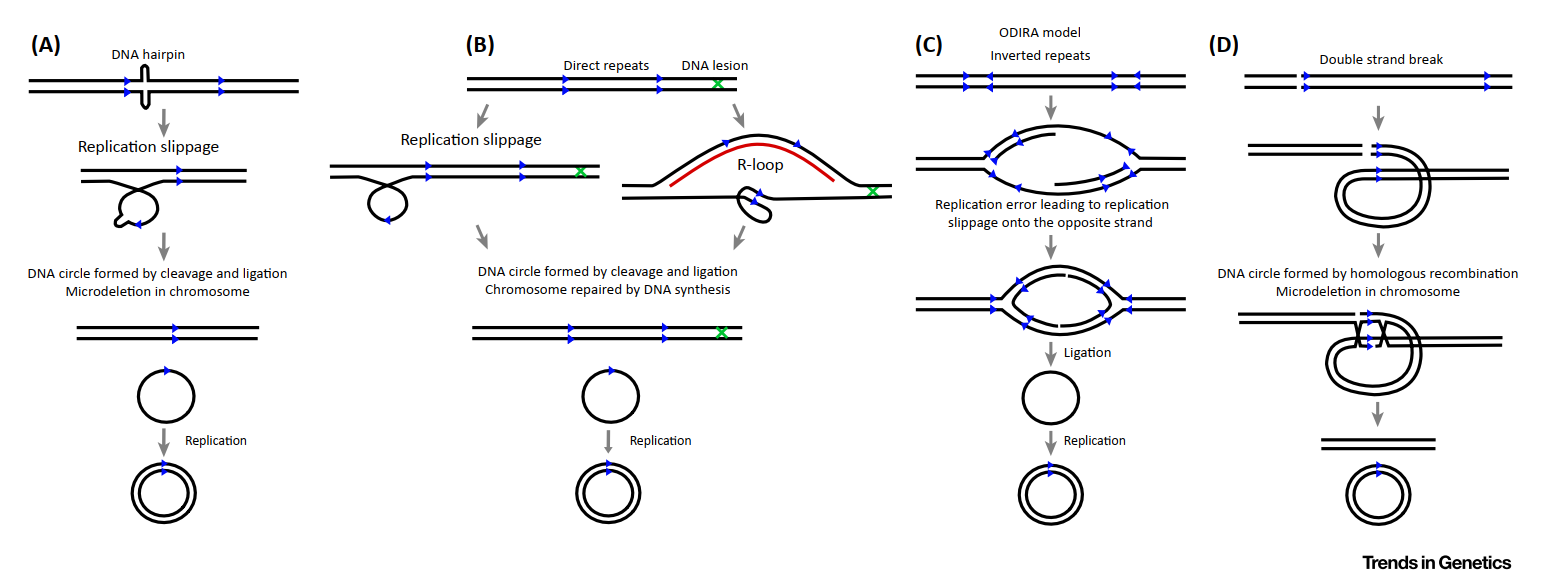
图4 eccDNA形成机制示意图([12])
2.4 eccDNA的序列来源
早期的研究就曾发现eccDNA所携带的序列不是单一基因组位点来源的,而是同时携带了多个位点的序列。2014年,Tiziana Storlazzi等在Nucleic Acids Research的文章结合了高通量SNP测序技术,FISH杂交和PCR技术分析了不同细胞中携带Myc基因的eccDNA,发现即使同一细胞样品中也会存在携带不同染色质序列的eccDNA分子,呈现高度的异质性。作者还利用他们的生物信息学工具分析了所研究样本中HSR和eccDNA的序列特征,排除了这些基因扩增现象是由chromothripsis驱动形成的可能,他们认为HSR和eccDNA的形成是一个持续不断进行的事情([19])。因此eccDNA并不是简单的基因组序列脱落后自连的产物,是经历过很多步骤后形成的。
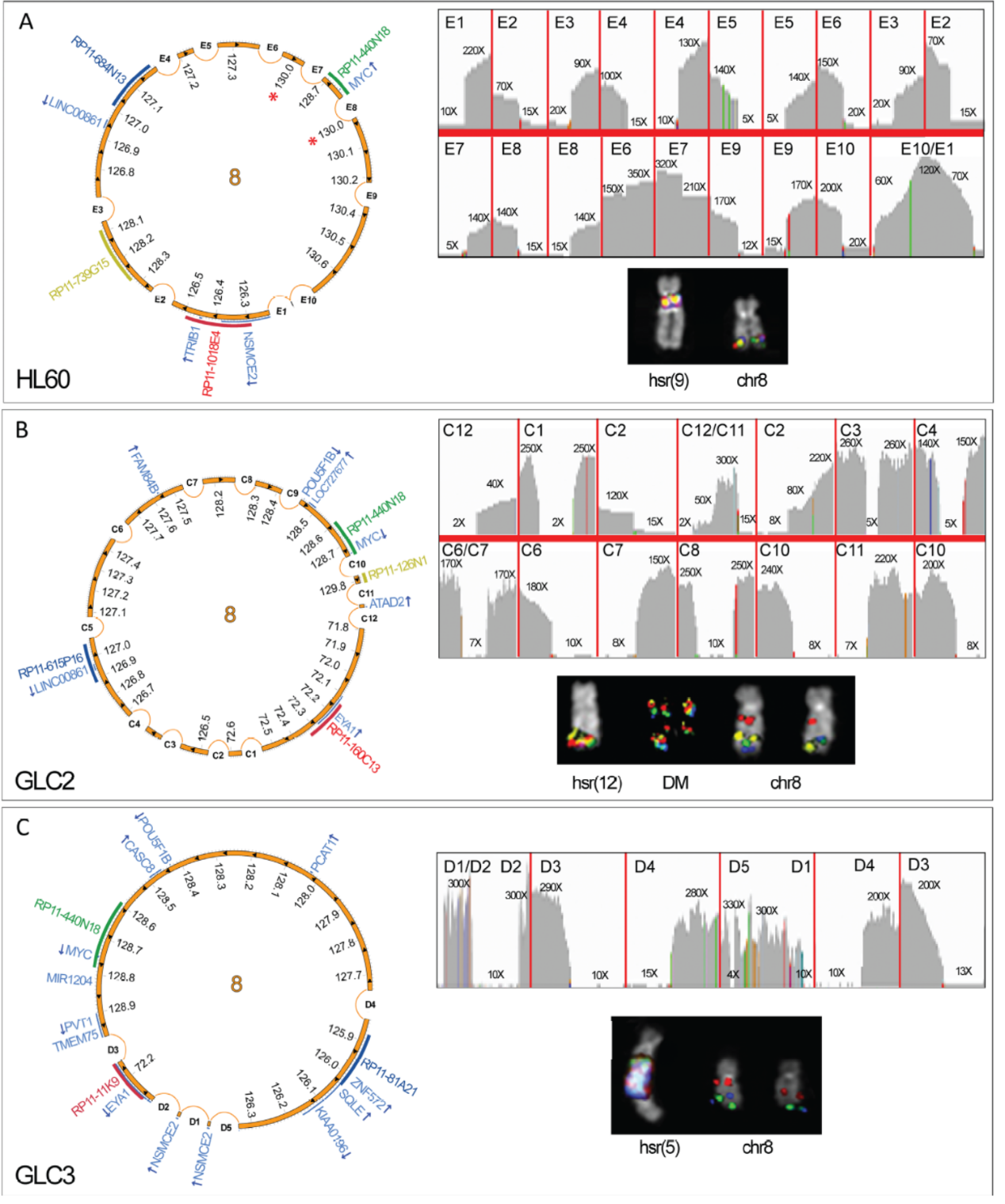
图5 eccDNA携带基因组多个位置的序列 ([19])
2.5 eccDNA基因的表达特征
eccDNA可以携带完整的基因,尤其是肿瘤中经常携带致癌的驱动基因,通过不受控制的表达这些基因,最终导致肿瘤不受调控的恶性增长。eccDNA是基因扩增的一种形式,对癌基因的表达增高的有重要贡献。早期的研究聚焦于发现eccDNA的存在,并且是癌基因扩增的重要形式,认为这对癌基因表达的贡献主要是由于基因拷贝数的增加所致。2019年,Paul S. Mischel团队的Nature文章发现,eccDNA对癌基因的表达增高不仅仅由于基因拷贝数的升高,还包括eccDNA中基因过度表达的因素,表现为即使用基因拷贝数进行归一化处理,eccDNA来源的基因表达数目也远高于基因组中的表达状况([10])。因此,eccDNA对癌基因的过度表达既有拷贝数增加的因素,更有eccDNA本身高度转录活性的贡献。这一特征也能从Peter C. Scacheri与Jeremy N. Rich联合发表的Cell文章中得到支持。
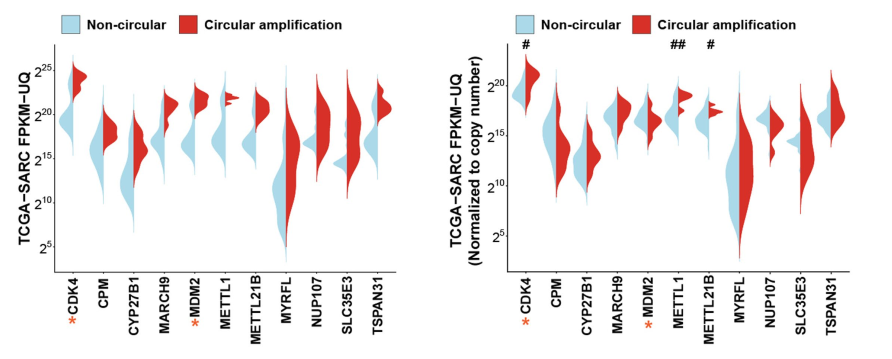
图6 eccDNA通过拷贝数增加和过度转录促进癌基因表达 ([10])
【珍藏版下集】 染色体外环状DNA的研究历程、功能机制和研究方法
聿筠
3. eccDNA的功能机制
3.1 eccDNA的常见功能
eccDNA作为独立的DNA分子,在细胞中发挥功能的方式和机制是非常重要的问题。目前对eccDNA的功能有一定的研究,但相信随着研究的不断深入,未来一定会有更多新的功能被发现。Anindya Dutta等在他们的综述文章中系统的汇总了eccDNA的主要功能,早期的研究就发现eccDNA可以介导细胞耐药作用,此外在癌基因扩增,肿瘤异质性调控,肿瘤进化等方面均起了非常重要的作用([12])。eccDNA的主要功能模型如下图所示,左侧的部分是作者预测的可能的作用机制,右侧是已经有实验证据支持的功能模型。
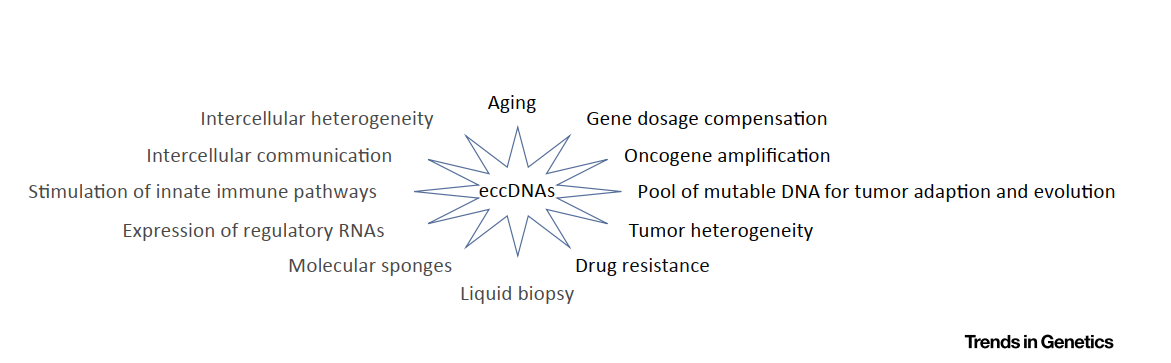
图7 常见eccDNA功能([12])
3.2 常见eccDNA携带基因
eccDNA可以携带完整的基因,一种模型认为eccDNA形成的早期是可以从基因组的大部分区域形成,但如果所携带的基因组片段没有给细胞带来生存优势,这些DNA片段将会随着细胞分裂过程而逐渐稀释和丢失,而当所携带的基因对细胞的生存有力,则可能被保留下来,并在细胞增殖分裂过程中不断被富集([20])。肿瘤中eccDNA常携带癌基因,常见的包括c-Myc,EGFR,MYCN,PDGFRA,MET等等([20]),在一些药物诱导生成的eccDNA中往往携带耐药相关的基因,比如氨甲喋呤耐药的小鼠细胞携带DHFR基因等等([4])。
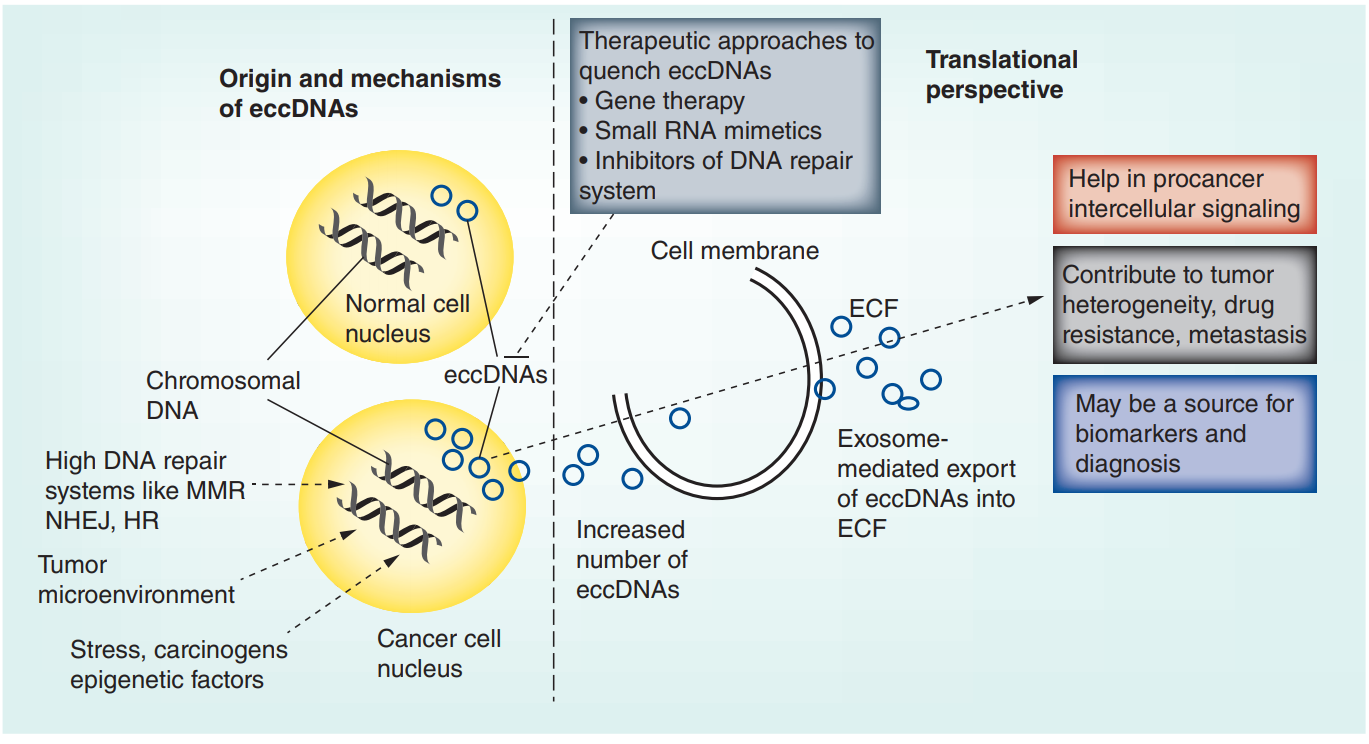
图8 eccDNA的形成和作用机制 ([21])
3.2 eccDNA遗传机制
eccDNA不带着丝粒元件,因此不能通过有丝分裂的方式均匀分配到分裂后的细胞中。但eccDNA可以通过不对称分布,在肿瘤细胞中实现定向富集。尤其是携带了癌基因的肿瘤细胞,会因为癌基因拷贝数的增高而获得生长速度的优势,不对称分裂过程中携带越多的eccDNA,细胞就会获得越强的生存优势,因此这种基于不对称分布状态的遗传机制能在快速增殖的细胞中定向富集([20])。
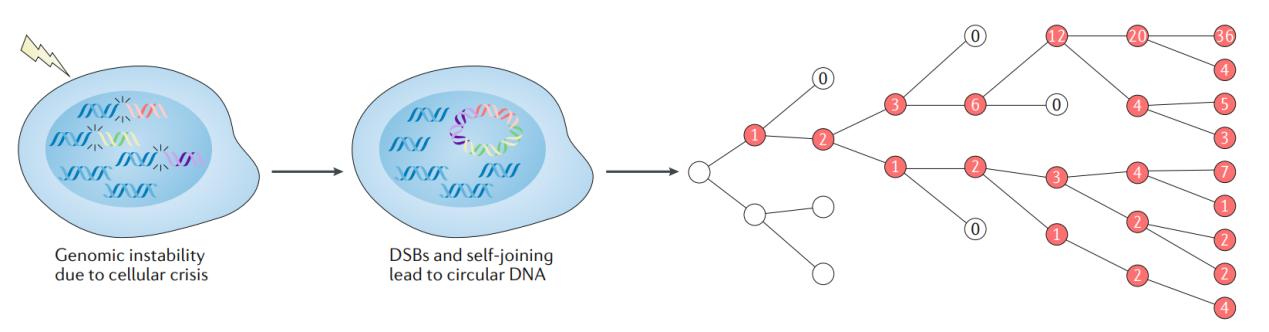
图9 eccDNA不均等分布遗传作用模型 ([20])
4. eccDNA主要研究方法
eccDNA的研究还处于不断发展的阶段,已报道的文章所使用的研究方法都值得我们学习借鉴,包括如何分析eccDNA,如何证明eccDNA是环状的,如何研究eccDNA的高级结构等。
4.1 如何分析eccDNA?
光学显微镜观察
eccDNA分子大小在1-5Mb,用普通的DNA染料和光学显微镜观察可以在视野中看到出于M期的细胞中存在的染色体外小DNA信号,其中一些就是eccDNA的信号。近几年发展起来的超高分辨率显微镜技术也可用于eccDNA的成像。Paul S. Mischel团队就曾用3D-SIM的超分辨成像技术进行eccDNA的成像分析([10])。
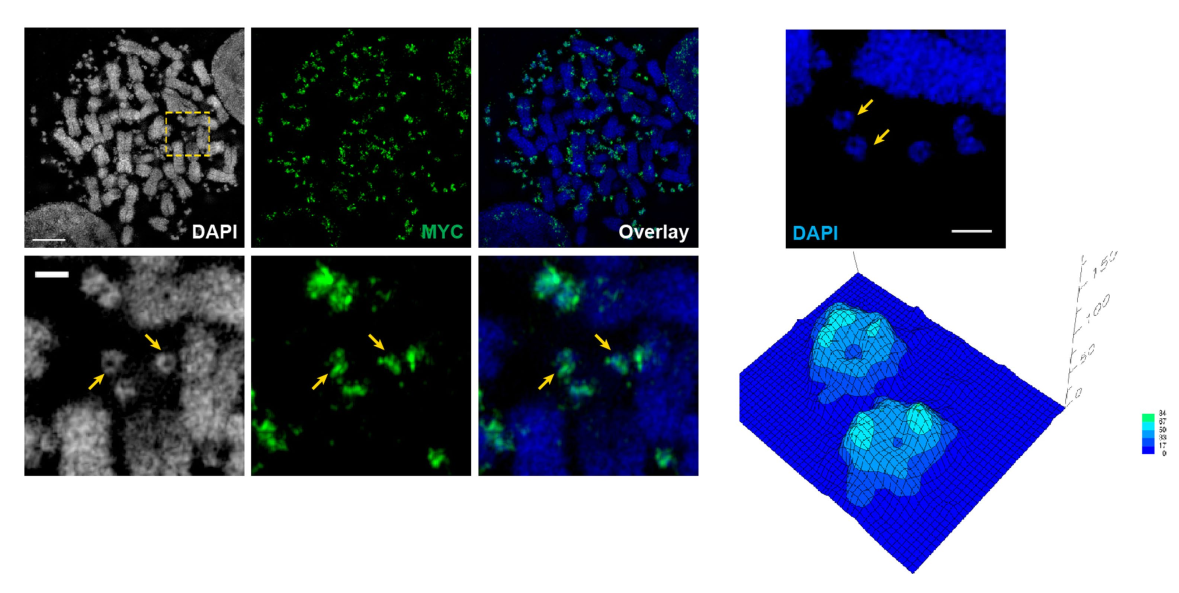
图10 3D-SIM超分辨显微镜观察eccDNA ([10])
电镜观察
光学显微镜的分辨率有限,没法实现对eccDNA更精细结构的观察和分析,电子显微镜可以解决这一问题。eccDNA的发现过程一直有电镜的贡献,最早的报道双微体结构的发现,直到最近的eccDNA研究工作都用到了电子显微镜。扫描电镜(SEM)和透射电镜(TEM)都可以用于eccDNA的成像观察。还有一种将同一视野的光学信号和电镜信号进行叠加的光电联用技术也可用于eccDNA的成像观察。
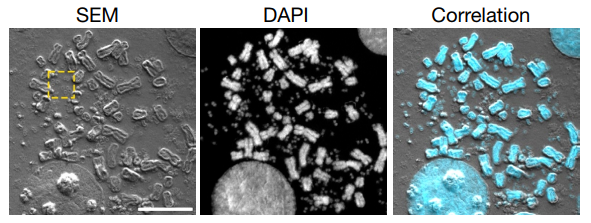
图11 光电联用观察eccDNA ([10])
密度梯度离心
氯化铯密度梯度离心是早期研究DNA的重要技术方法,可以分离出环状的DNA分子。但这种基于密度梯度离心的方法对细胞样品的需求量很大,尤其是研究eccDNA这样的低丰度物质,样品需求量会非常大,现在用的已经比较少了,但也依然是一个可选择的分离eccDNA的方法。
ATAC-see
Tn5转座子可以在裸露的基因组DNA区域随机插入,利用这一特点,可以实现对染色质开放性的检测。通过ATAC染色,将裸露的DNA区域利用Tn5转座酶将其染色,结合FISH和DAPI染色,可以实现对目标信号的定位观测。
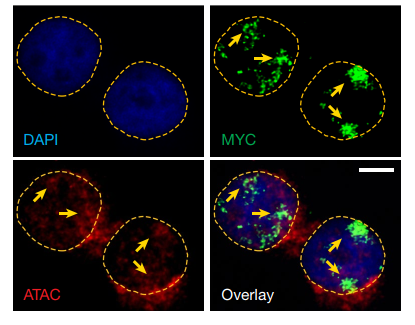
图12 ATAC-see观测疏松染色质 ([10])
Southern Blot
核酸杂交技术在DNA和RNA研究中应用非常广泛,针对eccDNA的探针进行Southern杂交也是可以用于eccDNA研究的方法之一。
荧光原位杂交(FISH)
利用eccDNA的荧光探针在细胞样品中检测eccDNA。FISH是比较有效的观察已知信息eccDNA的重要工具。
三代测序
二代测序的序列读长都比较短,一般小于500bp,可以用于分析eccDNA中的junction point信息,但eccDNA往往比较大,并且往往携带多个染色质来源的序列,因此仅用二代测序很难完整构建出eccDNA的全长序列。三代测序技术显著提高了测序中分子读长的问题,可以用于eccDNA的全长序列鉴定。
4.2 如何证明eccDNA是环状的?
上述观察eccDNA的技术方法中很多也可以用于证明它是环状分子,包括直接的电镜观察,三代测序等技术都可以用于证明eccDNA是环状分子。
双向电泳
环状DNA和线性DNA的结构有所不同,可以通过垂直方向的双向电泳将其分开。这种双向电泳技术还可用于DNA复制叉的研究。
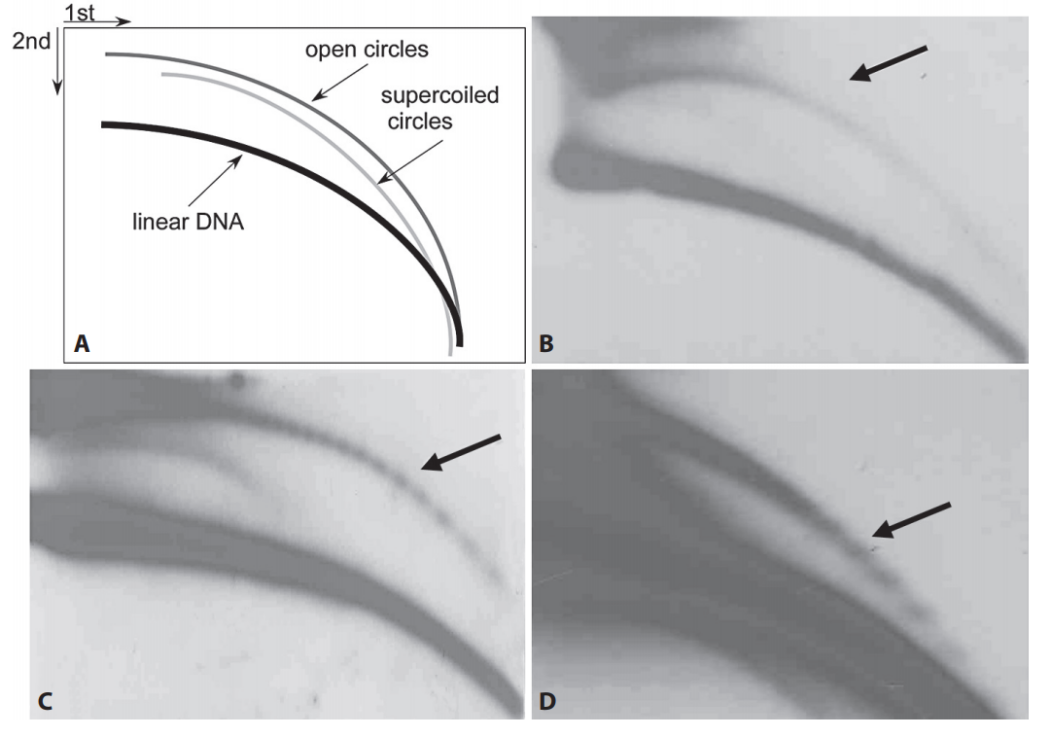
图13 双向电泳分析eccDNA的结构 ([22])
4.3 eccDNA的高级结构研究方法
ChIP-seq
eccDNA常伴随高度开放的染色质结构,也结合了组蛋白,且其中的H3K27ac修饰状态非常高,因此以H3K27ac等蛋白进行ChIP分析是研究eccDNA的重要手段。
4C-seq
是分析染色质高级结构的一种方法,主要原理是先用内切酶对基因组DNA做一次消化,然后进行连接,去除交联之后再做一次消化和链接,最后分析基因组相互作用的信息。4C-seq能更清晰的分析同一段染色质与多个染色质相互作用的情况([23])。
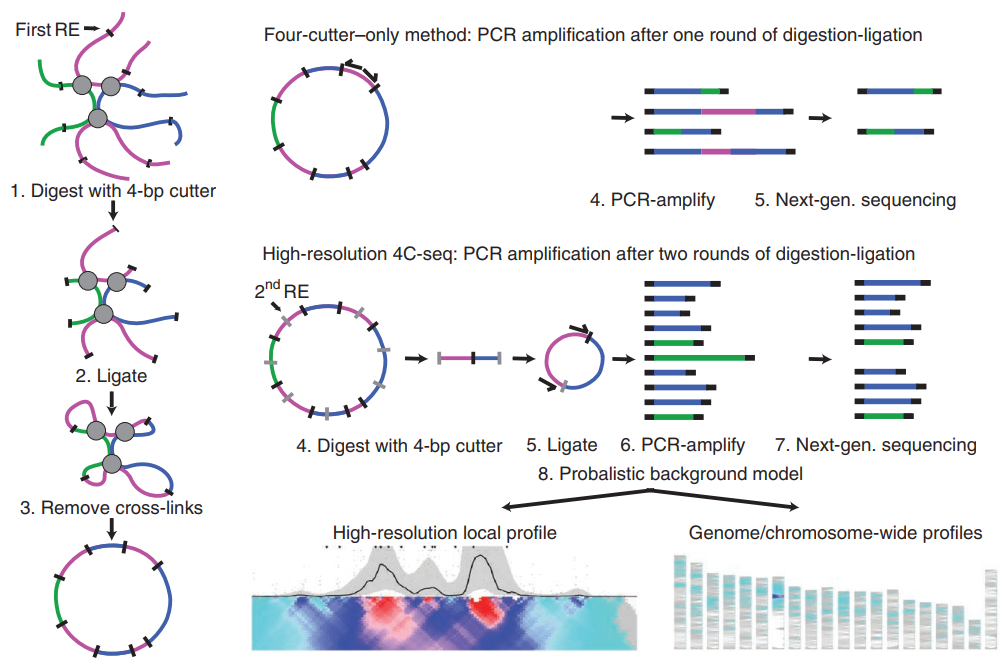
图14 4C-seq原理 ([23])
PLAC-seq
该技术主要用于分析染色质不同区域相互作用,样品先经过限制性内切酶处理,然后用连接酶将离得比较近的DNA片段连接在一起。如果不同区域的染色质空间上比较靠近,就会被连接在一起,这样经过ChIP等测序分析后,就可以找出远距离相互作用的染色质信息([24])。该技术的原理示意图如下:
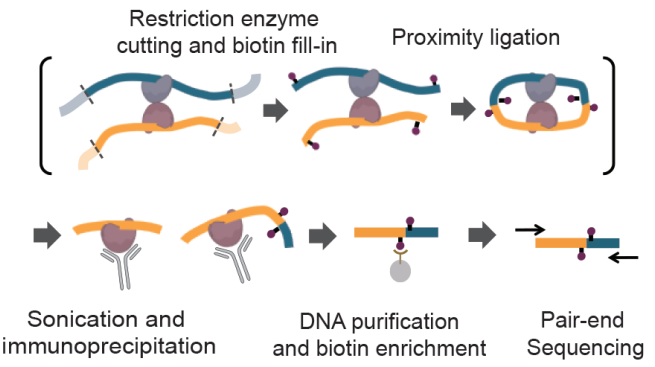
图15 PLAC-seq原理 ([24])
ATAC-seq
Tn5转座子可以在裸露的基因组DNA区域随机插入,利用这一特点,可以实现对染色质开放性的检测。
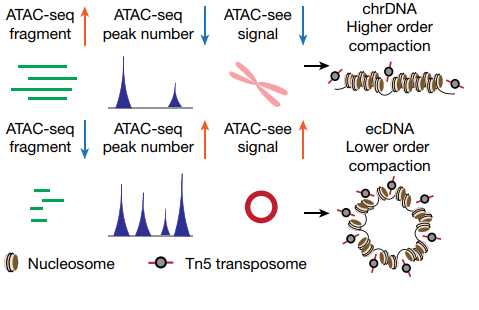
图16 ATAC-seq用于eccDNA的开放性分析 ([10])
4.4 几个分析工具及其主要工作原理
AmpliconArchitect
由Paul S. Mischel团队开发的专门用于寻找基因组来源的eccDNA,主要原理是基于全基因组测序的数据比对结果,自动检索基因组断裂点和其他基因组扩增子的区域,结合CNV和SV的结果,形成eccDNA完整序列的报告([25])。工具的下载地址:
https://github.com/virajbdeshpande/AmpliconArchitect
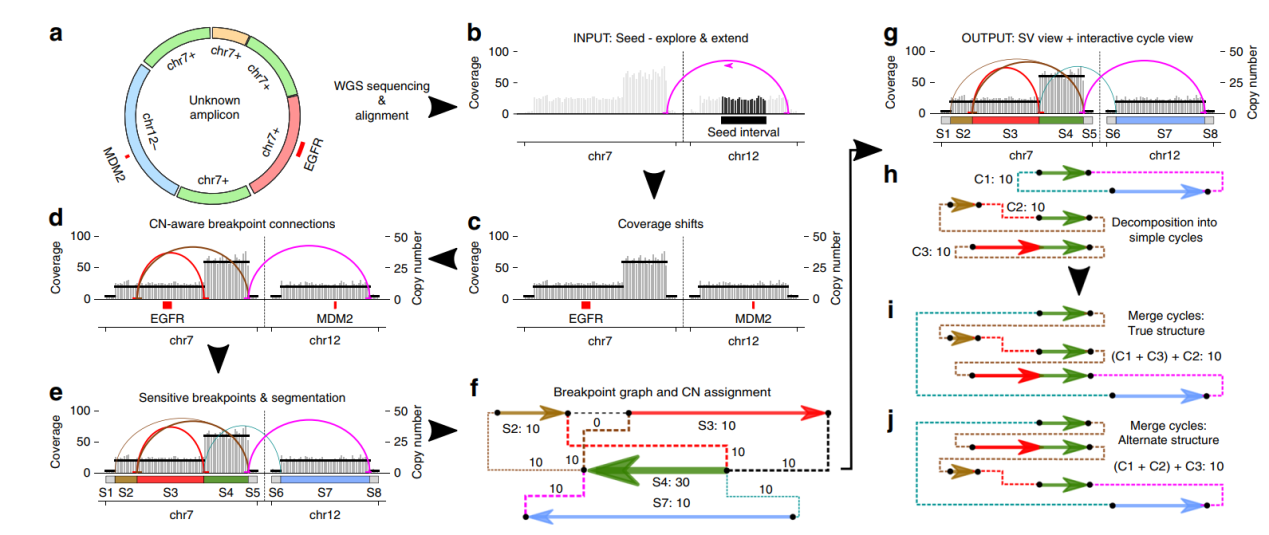
图17 AmpliconArchitect工具工作原理 ([25])
AmpliconReconstructor
Paul S. Mischel团队在AmpliconArchitect的基础上整合成像学信息进行eccDNA识别的工具([10])。工具的下载地址:
https://github.com/jluebeck/AmpliconReconstructor
5. 有关eccDNA几个问题的思考
5.1 eccDNA与circRNA有关系吗?
eccDNA和circRNA都是环状的核酸分子,有没有可能eccDNA也可以促进circRNA的产生呢?我个人认为完全有可能。eccDNA是来源于基因组的携带完整基因的环状DNA分子,其中还有基因上游的增强子元件,甚至会经过多次重组整合不同染色质来源的序列,这为eccDNA所携带的基因表达状态提供了全新的序列条件和调控方式,从Paul S. Mischel团队的Nature文章和Peter C. Scacheri与Jeremy N. Rich联合发表的Cell文章的结果来看,eccDNA一般处于高度开放的状态,基因的转录异常活跃([10, 11]),这也为circRNA的形成创造了条件。eccDNA还可以携带不同染色质的序列,有可能会引入不同于染色质中原基因位点的新序列元件,这也为circRNA的形成提供了新的可能。但不管怎样,eccDNA中基因转录,RNA编辑的体系应该是和基因组DNA一致的,如果希望探索这方面的问题,建议首先从鉴定eccDNA全长序列入手,看有没有特殊的序列进入eccDNA,然后再设计实验分析其中circRNA的形成。
5.2 为什么大部分染色体外DNA都是环状的?
DNA复制过程存在一种称为5’-末端缩隐的问题,就是DNA复制其实必须在前体RNA的后面延长,不能从零开始DNA的复制,因此在被复制的DNA模板的5’端是没法直接通过DNA复制过程将其补齐的,如果这样经过多轮的DNA复制之后DNA模板会变得越来越短,这就是5’-末端缩隐问题。生物体解决这个问题有两种方法,一种是在末端增加非基因序列来保护基因序列不会丢失,真核生物染色末端的端粒就是起这样的作用。另一种策略是让DNA变成环形,这样就可以通过滚环复制的方式实现全序列的复制。细菌,病毒和酵母等低等生物的基因组呈现环形就是基于这个原因。回到我们的主角,如果基因组因为种种原因产生了一些DNA片段,细胞内也存在将这些片段互连或自连的机制(同源重组或微同源序列介导的末端连接等等)([12]),因此一些DNA片段就会最终形成环状DNA分子。随着细胞增殖和分裂,那些没有成功成环的序列会因为5’-末端缩隐问题而越来越短, 最终消失。而顺利成环的分子就会正常复制,假如刚好携带了癌基因等对细胞生长有利的基因,就会通过不对称分布实现定向富集。
eccDNA虽然很早就发现和报道了,但只有到近期通过结合各种高通量测序技术之后才对它们进行了深入的分析研究。目前还有很多重要的科学问题没有得到很好的解答,包括eccDNA的形成机制,是否存在形成eccDNA的拼接热点?eccDNA对基因组DNA的影响和调控方式,eccDNA是否存在细胞间传递机制?肿瘤发生发展过程中eccDNA如何实现富集或丢失的?等等,相信随着研究工作的不断推进,这些问题可以陆续得到解答。
参考文献
1. Barreto, S.C., M. Uppalapati, and A. Ray, Small Circular DNAs in Human Pathology. Malays J Med Sci, 2014. 21(3): p. 4-18.
2. Hotta, Y. and A. Bassel, Molecular Size and Circularity of DNA in Cells of Mammals and Higher Plants. Proc Natl Acad Sci U S A, 1965. 53: p. 356-62.
3. Cox, D., C. Yuncken, and A.I. Spriggs, Minute Chromatin Bodies in Malignant Tumours of Childhood. Lancet, 1965. 1(7402): p. 55-8.
4. Alt, F.W., et al., Selective multiplication of dihydrofolate reductase genes in methotrexate-resistant variants of cultured murine cells. J Biol Chem, 1978. 253(5): p. 1357-70.
5. Kohl, N.E., et al., Transposition and amplification of oncogene-related sequences in human neuroblastomas. Cell, 1983. 35(2 Pt 1): p. 359-67.
6. Shibata, Y., et al., Extrachromosomal microDNAs and chromosomal microdeletions in normal tissues. Science, 2012. 336(6077): p. 82-6.
7. Nathanson, D.A., et al., Targeted therapy resistance mediated by dynamic regulation of extrachromosomal mutant EGFR DNA. Science, 2014. 343(6166): p. 72-6.
8. Turner, K.M., et al., Extrachromosomal oncogene amplification drives tumour evolution and genetic heterogeneity. Nature, 2017. 543(7643): p. 122-125.
9. Moller, H.D., et al., Circular DNA elements of chromosomal origin are common in healthy human somatic tissue. Nat Commun, 2018. 9(1): p. 1069.
10. Wu, S., et al., Circular ecDNA promotes accessible chromatin and high oncogene expression. Nature, 2019.
11. Morton, A.R., et al., Functional Enhancers Shape Extrachromosomal Oncogene Amplifications. Cell, 2019.
12. Paulsen, T., et al., Discoveries of Extrachromosomal Circles of DNA in Normal and Tumor Cells. Trends Genet, 2018. 34(4): p. 270-278.
13. Schimke, R.T., Gene amplification in cultured animal cells. Cell, 1984. 37(3): p. 705-13.
14. Stephens, P.J., et al., Massive genomic rearrangement acquired in a single catastrophic event during cancer development. Cell, 2011. 144(1): p. 27-40.
15. Vogt, N., et al., Molecular structure of double-minute chromosomes bearing amplified copies of the epidermal growth factor receptor gene in gliomas. Proc Natl Acad Sci U S A, 2004. 101(31): p. 11368-73.
16. Sunnerhagen, P., et al., Molecular cloning and characterization of small polydisperse circular DNA from mouse 3T6 cells. Nucleic Acids Res, 1986. 14(20): p. 7823-38.
17. Motejlek, K., et al., Increased amount and contour length distribution of small polydisperse circular DNA (spcDNA) in Fanconi anemia. Mutat Res, 1993. 293(3): p. 205-14.
18. Smith, G., et al., c-Myc-induced extrachromosomal elements carry active chromatin. Neoplasia, 2003. 5(2): p. 110-20.
19. L’Abbate, A., et al., Genomic organization and evolution of double minutes/homogeneously staining regions with MYC amplification in human cancer. Nucleic Acids Res, 2014. 42(14): p. 9131-45.
20. Verhaak, R.G.W., V. Bafna, and P.S. Mischel, Extrachromosomal oncogene amplification in tumour pathogenesis and evolution. Nat Rev Cancer, 2019. 19(5): p. 283-288.
21. Tandon, I., et al., Extrachromosomal circular DNAs: an extra piece of evidence to depict tumor heterogeneity. Future Sci OA, 2019. 5(6): p. FSO390.
22. Cohen, S. and D. Segal, Extrachromosomal circular DNA in eukaryotes: possible involvement in the plasticity of tandem repeats. Cytogenet Genome Res, 2009. 124(3-4): p. 327-38.
23. van de Werken, H.J., et al., Robust 4C-seq data analysis to screen for regulatory DNA interactions. Nat Methods, 2012. 9(10): p. 969-72.
24. Fang, R., et al., Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Res, 2016. 26(12): p. 1345-1348.
25. Deshpande, V., et al., Exploring the landscape of focal amplifications in cancer using AmpliconArchitect. Nat Commun, 2019. 10(1): p. 392.
.png)