十月已过去一半,在十月的上半个月,circRNA研究相关报道相对保持稳定,主要包括Nucleic Acids Rsearch杂志公布了两个circRNA相关的数据库,发表了RNase R作用的结构生物学机制等等。详情如下:
1. Nucleic Acids Research杂志连续发布两个circRNA相关数据库:
CSCD:肿瘤特异性circRNA数据库
9月28日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了武汉大学何春江教授和UT health Science Center韩冷教授为共同通讯作者的文章,介绍发布一个全新的肿瘤特异性circRNA数据库:CSCD(cancer-specific circRNA database,网址:http://gb.whu.edu.cn/CSCD)[1]。

CSCD数据库从ENCODE中收集了19种肿瘤类型的87种细胞系及141种正常细胞的circRNA数据,构建了该数据库。总共汇总得到了272152种肿瘤特异性的circRNAs,950962种正常细胞特异的circRNAs,还有170909种为肿瘤和正常样本共有的。共收集了1394023种circRNA分子,其中外显子来源759039种,内含子来源436668种,基因间序列来源122806种。来自mRNA的1153542种,来lncRNA的42165种。预测到了MRE 76439955个,RNA结合蛋白结合位点103927037个,ORF 3462097个。
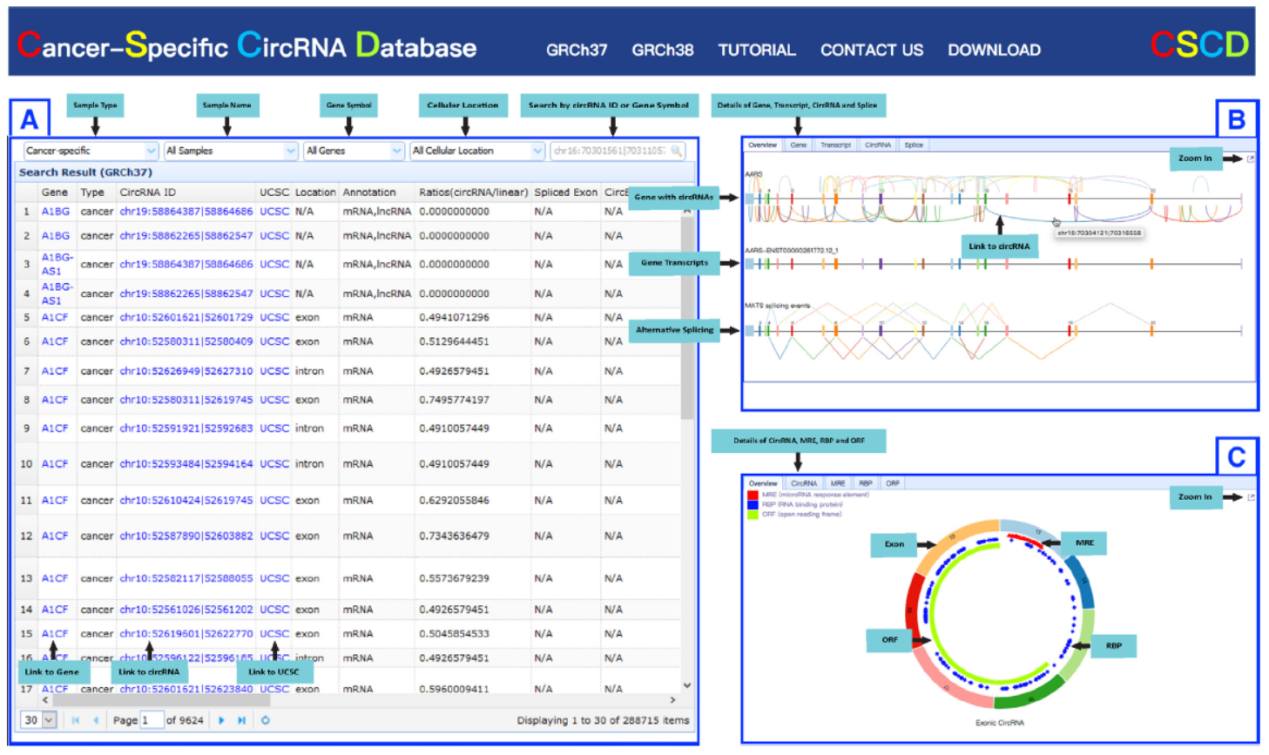
图1数据库概览 (来自[1])
exoRBase:外泌体长链RNA数据库
10月9日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了复旦大学医学院上海肿瘤研究所黄胜林教授为通讯作者的文章,公布他们开发的专门收录外泌体中各种长链RNA(mRNA, lncRNA及circRNA)的数据库:exoRBase [2]。数据库连接:http://www.exoRBase.org

exoRBase数据库共收录了58330种circRNAs,15501种lncRNAs,18333种mRNAs,它们来自92份血清外泌体RNA-seq测序样本。作者还依据GTEx project获得组织特异性RNA表达特征,分析了相关RNA分子的组织来源。数据库收录的样本包括健康对照者32例,冠状动脉心脏疾病(CHD)6例,结肠癌(CRC)12例,肝细胞癌(HCC)21例,胰腺腺癌(PAAD)14例,乳腺癌2例。

图2 exoRBase数据库概览 (来自[2])
2. RNase R作用的结构生物学机制
10月3日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了台湾中央研究院分子生物学研究所Hanna S. Yuan为通讯作者的文章,介绍解析了RNase R作用的结构生物学机制[3]。

RNase R是常用的3’→5’外切核糖核酸酶,在circRNA研究中经常作为消化线性RNA的工具酶。本文作者解析了来自大肠杆菌的截短型RNase R(87-725 aa)的1.85 Å分辨率的晶体结构,揭示了RNase R同步发挥RNA解链和消化作用的结构生物学机制。
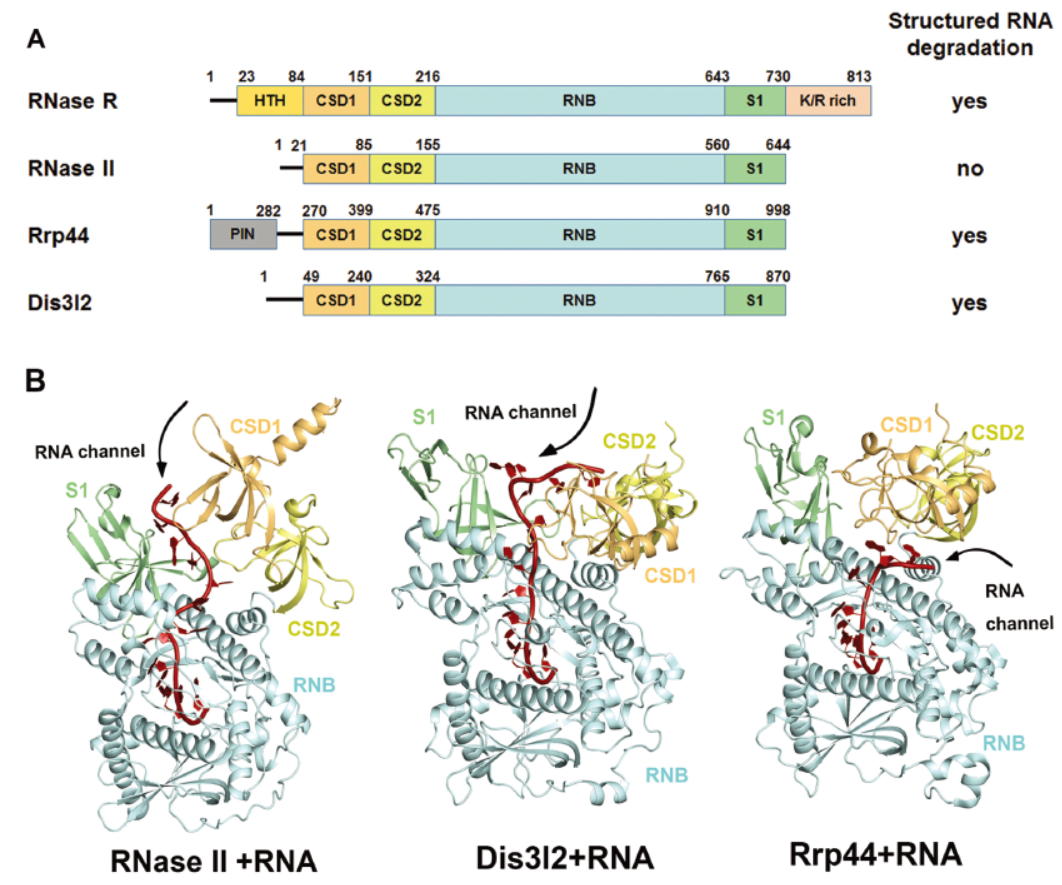
图3 大肠杆菌RNase R的结构生物学机制 (来自[3])
3. circNPM1在急性髓系白血病(AML)中的作用
9月29日,著名血液学杂志Haematologica(IF 2016= 7.702)在线发表了德国乌尔姆大学医学院Lars Bullinger教授为通讯作者的文章,介绍发现circNPM1在急性髓系白血病(AML)中发挥重要作用[4]。

作者发现来自NPM1基因的circRNA hsa_circ_0075001与AML疾病存在相关性。circ_0075001表达状态差异对应于不同的基因表达状态,如高表达的患者TLR通路基因表现为低表达等等。
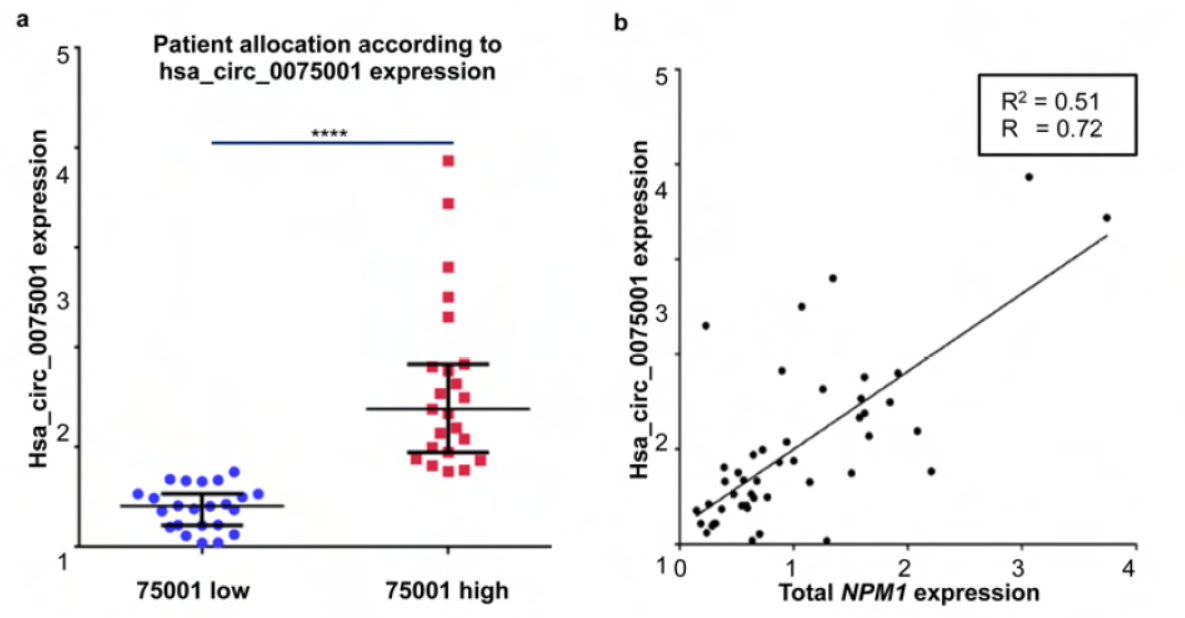
图4 AML病人circ_0075001表达特征 (来自[4])
4. HPV诱发宫颈癌中lncRNA和circRNA表达特征
9月19日,Frontiers in Microbiology杂志(IF 2016= 4.076)在线发表了中科院武汉病毒所Chen Mingyue和崔杰研究员为共同通讯作者的文章,介绍在HPV(人乳头状瘤病毒)感染诱发的宫颈癌中发现疾病相关lncRNA和circRNA[5]。

作者分析了三利HPV16感染诱发的宫颈癌和对应癌旁组织的全转录组,共发现了差异表达的19种lncRNAs,99种circRNAs,28 种miRNAs和304种mRNAs。作者也构建了这些基因的相互作用网络图[5]。
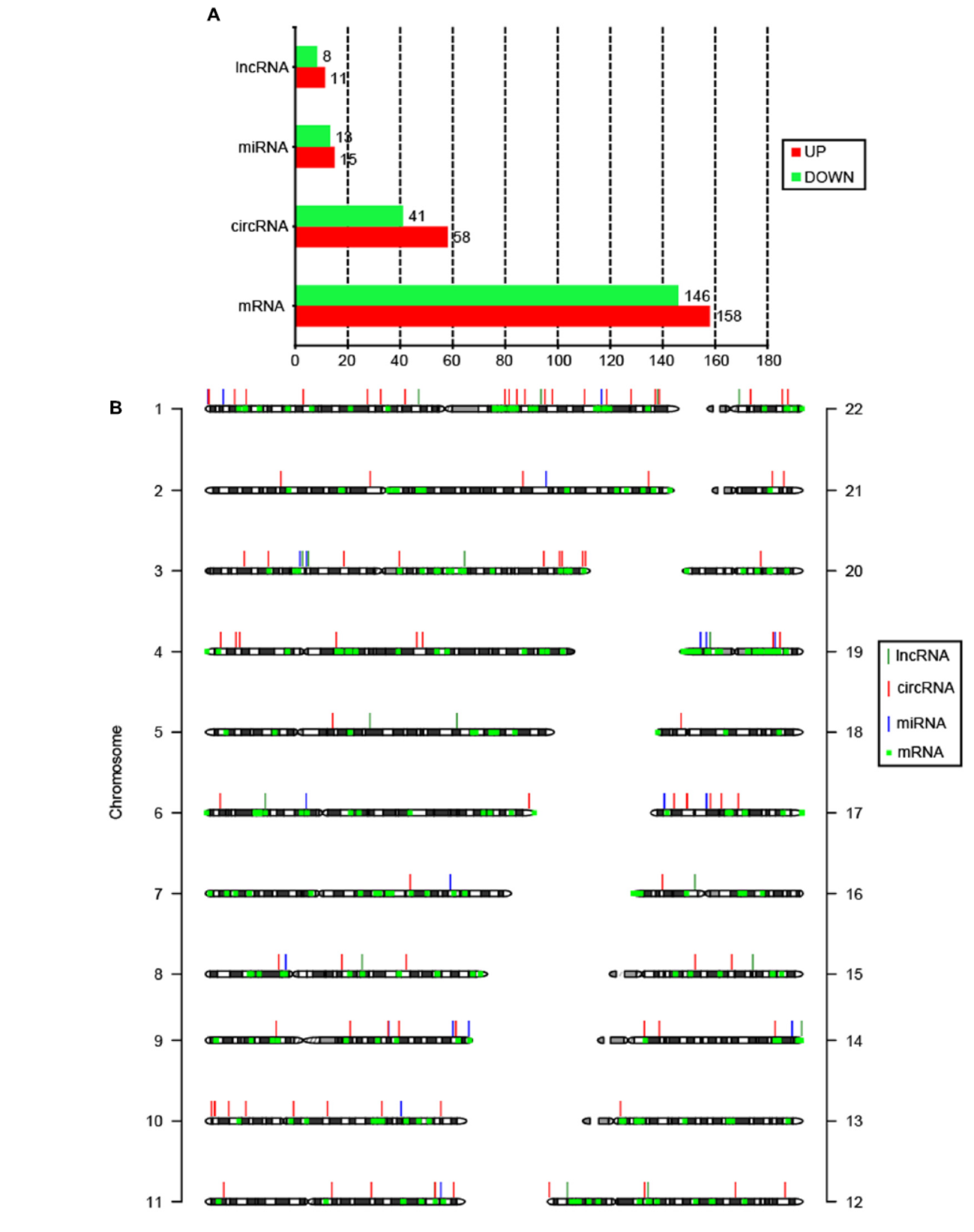
图5 HPV感染宫颈癌中差异表达基因(来自[5])
5. 组织特异性circRNA表达特征分析
10月3日,BMC Genomics杂志(IF 2016= 3.729)发表了美国德州大学休斯敦健康科学中心赵中明教授与南京航空航天大学宋晓峰教授为共同通讯作者的文章,介绍基于RNA-seq数据分析组织特异性circRNA表达特征的工作[6]。

文中共收集了来自成年人和胎儿的六种不同组织(结肠,心脏,肾脏,肝脏,肺和胃)来源的RNA-Seq数据,还收集了四种腺体组织(肾上腺,乳腺,胰腺和甲状腺)的数据。这些数据主要来源于NCBI和ENCODE。以大于2个有效Reads为标准,作者在成年,胎儿和腺体组织中分别分析到了8120,25933 和14433种circRNAs分子。这些circRNAs分子大部分为组织特异性表达的,比例占到36.97–50.04%。
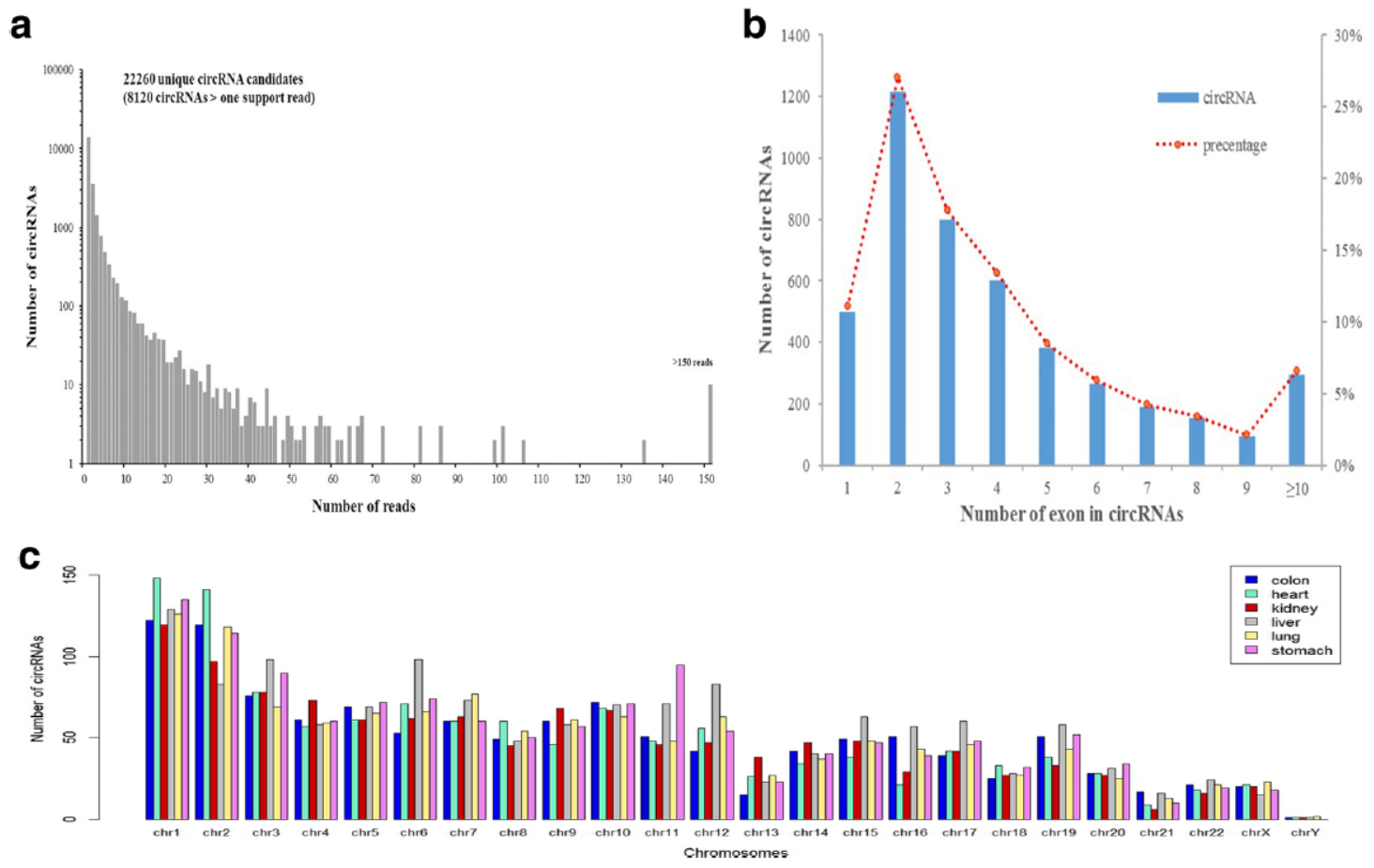
图6 组织特异性circRNAs分子总体分布特征 (来自[6])
6. 胃癌中发现具有癌基因特征的circRNA
10月5日,Cell Cycle杂志(IF 2016= 3.53)在线发表了北大人民医院王杉主任为通讯作者的文章,介绍在胃癌中发现了一组具有癌基因特征的circRNAs分子[7]。

作者利用芯片法分析了胃癌中circRNA和mRNA的表达特征,发现了三个高表达的circRNA分子:hsa_circ_0047905、hsa_circ_0138960 和has-circRNA7690-15。RNA干扰后细胞增殖和迁移能力下降。
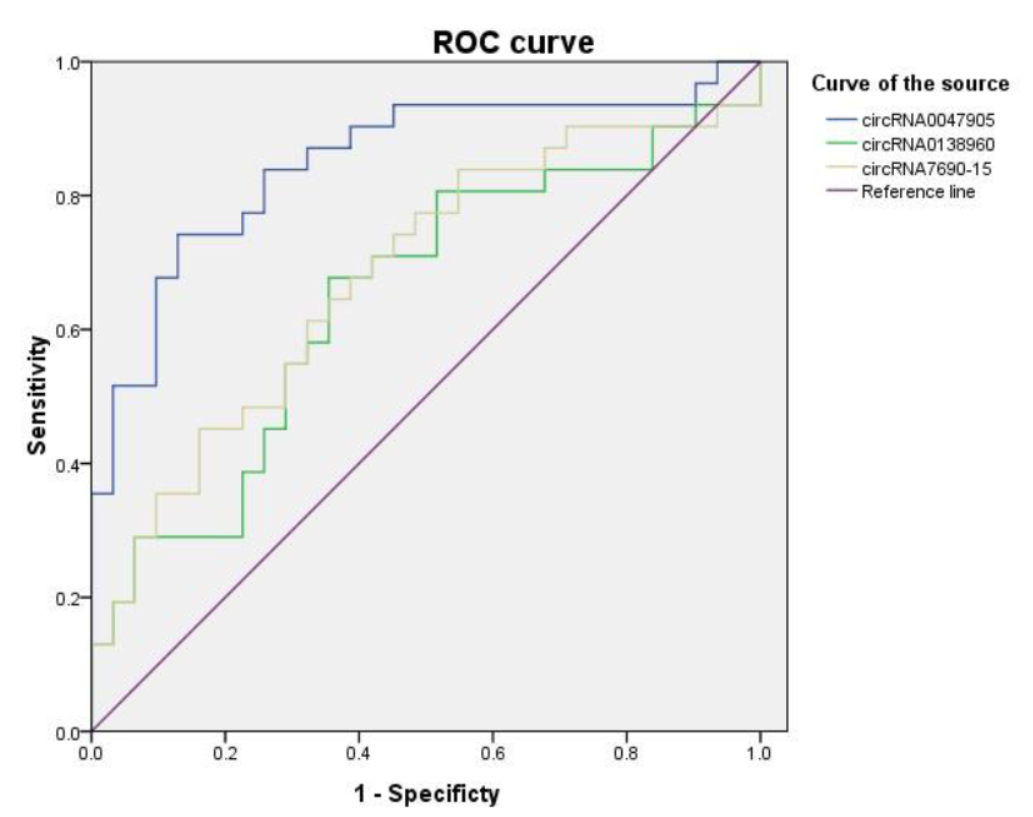
图7 胃癌中癌基因性circRNA的ROC曲线 (来自[7])
7. circRNA作为胃癌标志物
9月14日,world journal of gastroenterology杂志(IF 2016= 3.365)在线发表了安徽医科大学附属安徽省立医院朱志强主任为通讯作者的文章,介绍发现hsa_circ_0000745可作为胃癌诊断标志物 [8]。

作者在20例胃癌和癌旁组织中通过QPCR分析了三种circRNA分子的表达情况,基于通路预测和竞争性结合miRNA的分析,作者将研究聚焦在hsa_circ_0000745上。在60例临床标本的验证中表明hsa_circ_0000745在胃癌中低表达。ROC曲线分析表明将hsa_circ_00 00745与CEA联合检测可获得AUC值0.775。
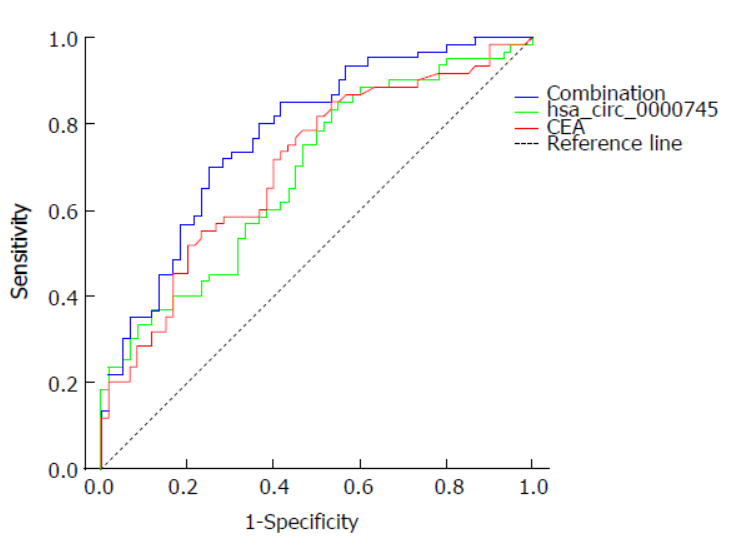
图8 hsa_circ_0000745在胃癌中的ROC曲线 (来自[8])
8. 人类主动脉瓣中circRNA表达分析
10月7日,Gene杂志(IF 2016= 2.4149)在线发表了南京医科大学附属第一医院邵永丰主任为通讯作者的文章,分析了人主动脉瓣中circRNA的表达特征[9]。

本文共分析到5476种circRNA分子,其中1412种为人类主动脉瓣特有的。作者也针对这些circRNA进行了通路分析,RBP结合情况预测等信息学分析。
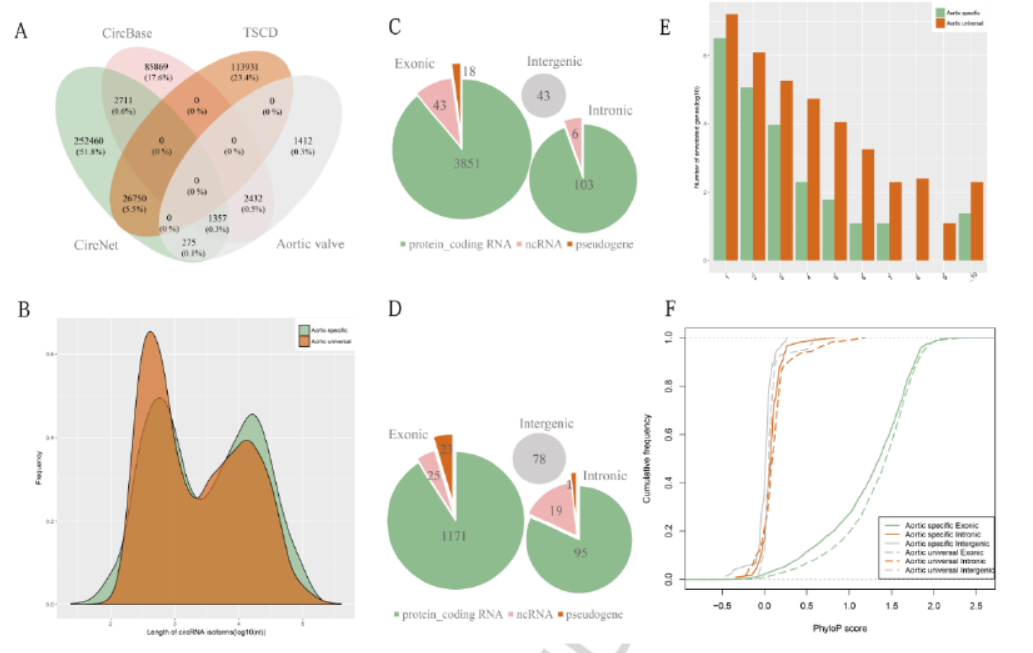
图9人类主动脉瓣中circRNA表达分析 (来自[9])
9. Moyamoya 病中circRNA表达分析
10月15日,Journal of the Neurological Sciences杂志(IF 2016=2.295)北京天坛医院神经外科赵继宗主任为通讯作者的文章,介绍分析了Moyamoya病中circRNA的表达特征[10]。

作者利用芯片法分析了各5例病人与健康对照样品,共得到146种差异显著的circRNA分子,其中29种为上调。文中也针对这些circRNA进行了通路分析。
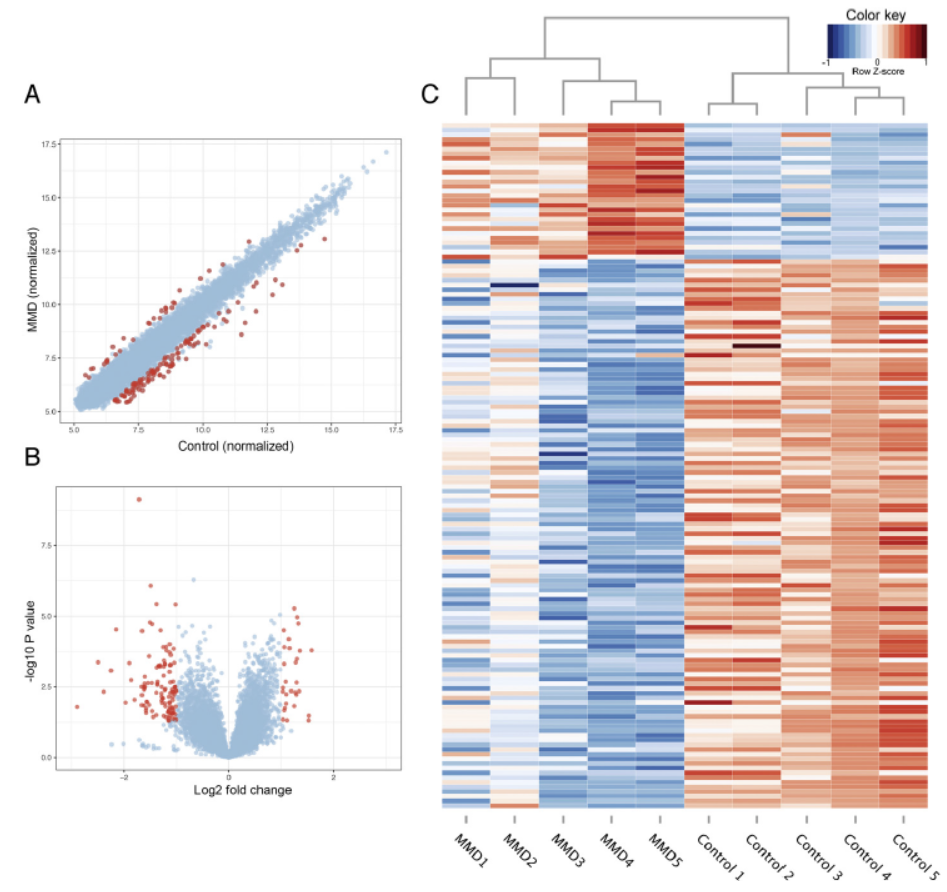
图10 Moyamoya 病中circRNA表达 (来自[10])
10. 类风湿性关节炎PBMC中circRNA表达分析
9月27日,Molecular Medicine Reports杂志(IF 2016= 1.692)在线发表了深圳市人民医院(暨南大学第二临床医院)戴勇主任为通讯作者的文章,分析了类风湿性关节炎患者外周血单个核细胞(PBMC)中circRNA表达特征[11]。

本文利用芯片法进行分析了各10例类风湿性关节炎患者和健康对照者的外周血单个核细胞样品,共得到了584种差异表达的circRNA分子,其中255种为上调。
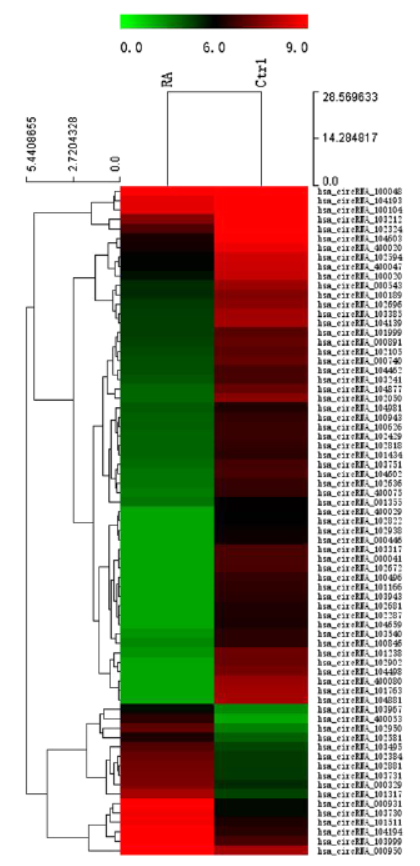
图11 类风湿性关节炎PBMC中circRNA差异表达 (来自[11])
11. 植物相关circRNA研究进展
拟南芥全基因组circRNA分析
9月27日,Frontiers in Plant Science杂志(IF 2016= 4.2986)在线发表了扬州大学农学院金飚为通讯作者的文章,介绍将拟南芥中circRNA进行全基因组定位分析[12]。

作者利用链特异性测序分析了不同发育阶段,应激条件下circRNA的表达情况,做成了拟南芥基因组中circRNA的全基因组定位图。共得到5861种circRNA的信息,其中1275种为新发现的。
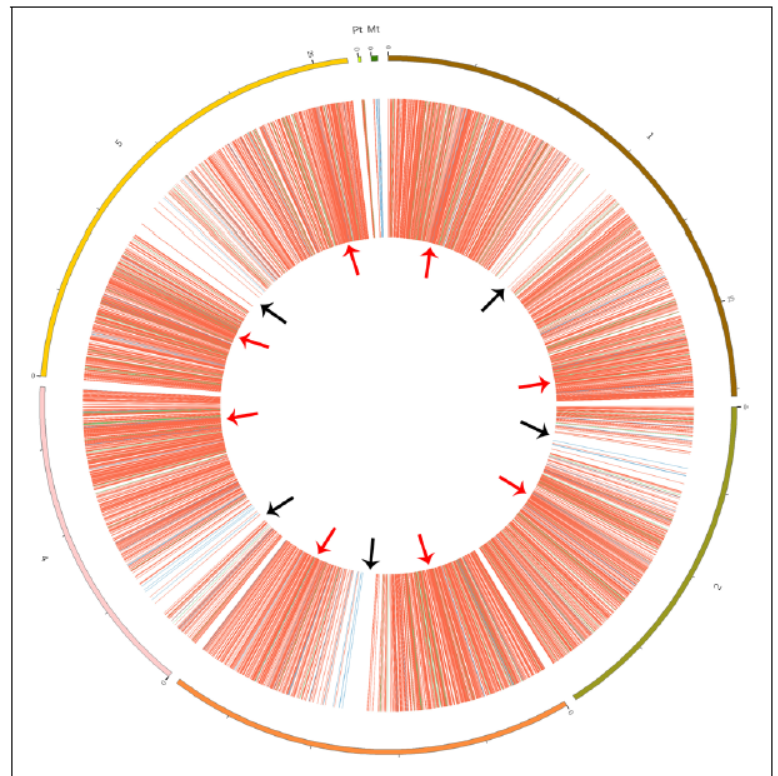
图12 拟南芥circRNA的全基因组定位 (来自[12])
棉属多倍体物种及其祖先中保守circRNA表达特征分析
10月3日,FEBS Letters杂志(IF 2016= 3.519 )在线发表了南京农业大学关雪莹和周宝良为共同通讯作者的文章,介绍在棉属多倍体物种及其祖先中保守性circRNA的表达特征分析[13]。

本文主要利用去rRNA后建库分析,RNA-seq后分析得到相关circRNA信息,其中发现了23种circRNA为非GT/AG剪切形成的,并且只有约10%的外显子来源的circRNA在两侧内含子中存在反向互补元件,这些特征说明在植物中可能存在与动物不同的circRNA形成机制。
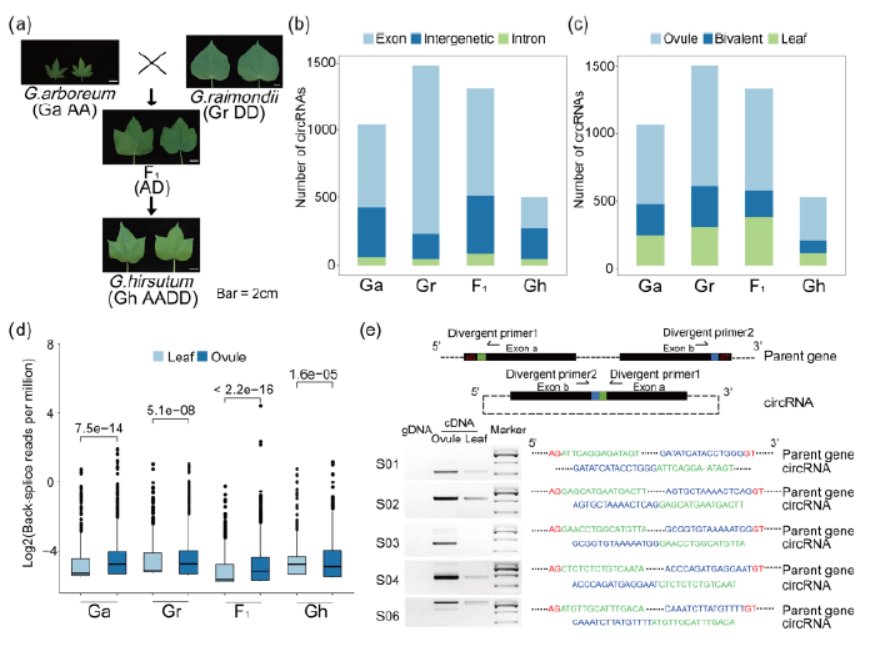
图13 棉属多倍体物种及其祖先中保守circRNA表达特征 (来自[13])
12. circRNA相关综述与观点
Oncogene发表重要circRNA综述
10月9日,Oncogene杂志(IF 2016= 7.519)在线发表了丹麦奥胡斯大学Lasse Sommer Kristensen 教授为通讯作者的综述文章,著名RNA研究专家Jørgen Kjems教授也是本文的共同作者之一[14]。
本文中作者系统汇总了目前关于circRNA研究的主要进展,重点汇总了circRNA作为肿瘤标志物的进展,还针对circRNA的功能研究,将可能具有抑癌基因特征和癌基因特征的circRNA分子进行了简单的分类。关于今后circRNA研究需要解决的问题,文章给出了非常中肯的建议,其中包括circRNA的标准化命名问题,肿瘤特异性circRNA表达特征形成的机制,circRNA的修饰等问题。提高RNA-seq的样品制备质量和分析精度也是需要继续改进的方向。
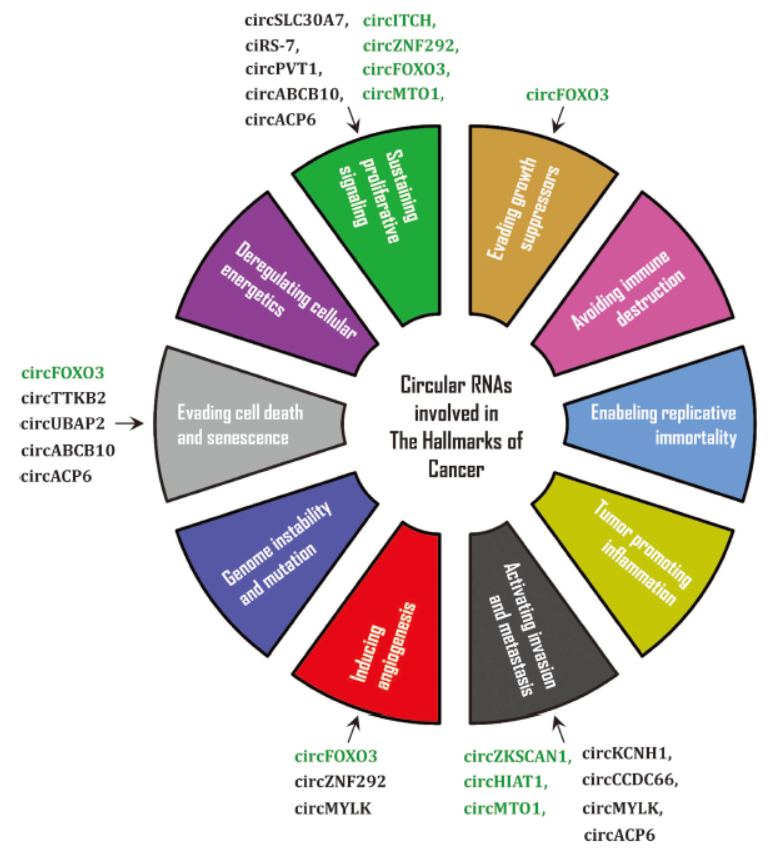
图12 可作为肿瘤标志物的circRNA分子 (来自[14])
Jørgen Kjems教授发表circRNA研究综述
10月6日,RNA Biology杂志(IF 2016= 3.9 )在线发表了知名RNA研究专家Jørgen Kjems教授为通讯作者的综述文章。本文主要针对即将出版的关于circRNA的专业书作的序(文中未提及书是谁撰写的,书题目是什么,估计不久将会公开)。在这篇序中,Jørgen Kjems教授教授大致分析了目前circRNA研究的热点和焦点问题,其中包括竞争性结合miRNA或蛋白的功能模型,还特别提到了今年新发现的circRNA可翻译蛋白的机制,也提到了包括circRNA形成机制,从细胞核中转运至胞浆的机制等目前circRNA研究普遍关注的重要问题。文章最后还提到circRNA作为药物用于临床的可能性 [15]。

OncoTarget发表circRNA综述
9月8日,Oncotarget杂志(IF 2016= 5.1657)发表了circRNA竞争性结合miRNA相关进展的综述,文章的通讯作者为郑州大学附属第一医院Duan Zhenfeng。

胃肠癌中circRNA综述
10月3日,Oncology Letters杂志(IF 2016= 1.3899)在线发表了北京协和医院由磊和赵玉沛为共同通讯作者的综述,汇总了胃肠癌中circRNA的相关研究进展[16]。

参考文献:
1. Siyu Xia, J.F., Ke Chen, Yanbing Ma, Jing Gong, Fangfang Cai, Yuxuan Jin, Yang Gao, Linjian Xia, Hong Chang, Lei Wei, Leng Han and Chunjiang He, CSCD: a database for cancer-specific circular RNAs. Nucleic Acids Research, 2017.
2. Shengli Li, Y.L., Bing Chen, Jingjing Zhao, Shulin Yu, Yan Tang, Qiupeng Zheng, Yan Li, Peng Wang, Xianghuo He and Shenglin Huang, exoRBase: a database of circRNA, lncRNA and mRNA in human blood exosomes. Nucleic Acids Research, 2017.
3. Lee-Ya Chu, T.-J.H., Bagher Golzarroshan, Yi-Ping Chen, Sashank Agrawal and Hanna S. Yuan, Structural insights into RNA unwinding and degradation by RNase R. Nucleic Acids Research, 2017.
4. Hirsch, S., et al., Circular RNAs of the nucleophosmin (NPM1) gene in acute myeloid leukemia. Haematologica, 2017.
5. Wang, H., et al., Identification of Novel Long Non-coding and Circular RNAs in Human Papillomavirus-Mediated Cervical Cancer. Front Microbiol, 2017. 8: p. 1720.
6. Tianyi Xu, J.W., Ping Han, Zhongming Zhao and Xiaofeng Song, Circular RNA expression profiles and features in human tissues: a study using RNA-seq data. BMC Genomics, 2017.
7. Lai, Z.Y., et al., Analysis of co-expression networks for circular RNAs and mRNAs reveals that circular RNAs hsa_circ_0047905, hsa_circ_0138960 and has-circRNA7690-15 are candidate oncogenes in gastric cancer. Cell Cycle, 2017: p. 0.
8. Huang, M., et al., Circular RNA hsa_circ_0000745 may serve as a diagnostic marker for gastric cancer. World J Gastroenterol, 2017. 23(34): p. 6330-6338.
9. Chen, J., et al., Identification of circular RNAs in human aortic valves. Gene, 2017.
10. Zhao, M., et al., Altered expression of circular RNAs in Moyamoya disease. J Neurol Sci, 2017. 381: p. 25-31.
11. Zheng, F., et al., Circular RNA expression profiles of peripheral blood mononuclear cells in rheumatoid arthritis patients, based on microarray chip technology. Mol Med Rep, 2017.
12. Chen, G., et al., Genome-Wide Identification of Circular RNAs in Arabidopsis thaliana. Front Plant Sci, 2017. 8: p. 1678.
13. Zhao, T., et al., Characterization of conserved circular RNA in polyploid Gossypium species and their ancestors. FEBS Lett, 2017.
14. Kristensen, L.S., et al., Circular RNAs in cancer: opportunities and challenges in the field. Oncogene, 2017.
15. Hansen, T.B. and J. Kjems, The invasion of circRNAs. RNA Biol, 2017: p. 0.
16. XIAOXIA REN, Y.D., LEI YOU and YUPEI ZHAO, Potential functions and implications of circular RNA in gastrointestinal cancer. Oncology Letters, 2017.
十月已过去一半,在十月的上半个月,circRNA研究相关报道相对保持稳定,主要包括Nucleic Acids Rsearch杂志公布了两个circRNA相关的数据库,发表了RNase R作用的结构生物学机制等等。详情如下:
1. Nucleic Acids Research杂志连续发布两个circRNA相关数据库:
CSCD:肿瘤特异性circRNA数据库
9月28日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了武汉大学何春江教授和UT health Science Center韩冷教授为共同通讯作者的文章,介绍发布一个全新的肿瘤特异性circRNA数据库:CSCD(cancer-specific circRNA database,网址:http://gb.whu.edu.cn/CSCD)[1]。

CSCD数据库从ENCODE中收集了19种肿瘤类型的87种细胞系及141种正常细胞的circRNA数据,构建了该数据库。总共汇总得到了272152种肿瘤特异性的circRNAs,950962种正常细胞特异的circRNAs,还有170909种为肿瘤和正常样本共有的。共收集了1394023种circRNA分子,其中外显子来源759039种,内含子来源436668种,基因间序列来源122806种。来自mRNA的1153542种,来lncRNA的42165种。预测到了MRE 76439955个,RNA结合蛋白结合位点103927037个,ORF 3462097个。
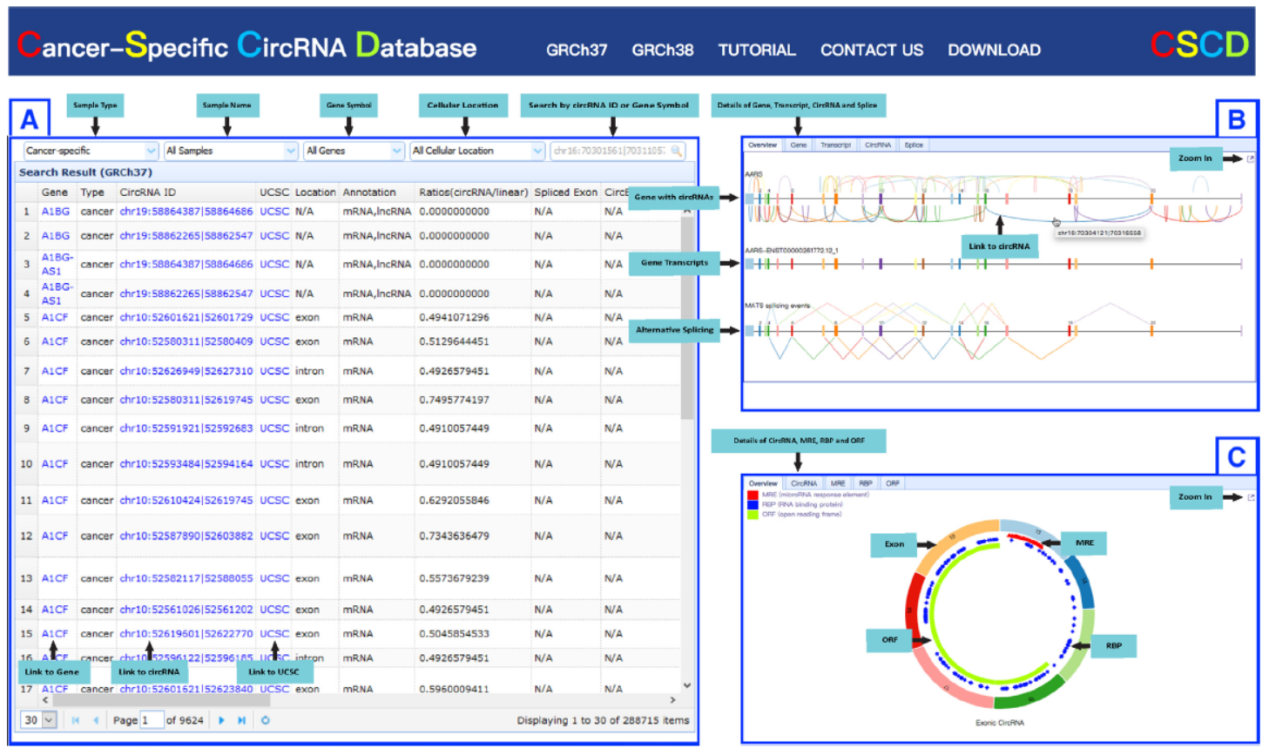
图1数据库概览 (来自[1])
exoRBase:外泌体长链RNA数据库
10月9日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了复旦大学医学院上海肿瘤研究所黄胜林教授为通讯作者的文章,公布他们开发的专门收录外泌体中各种长链RNA(mRNA, lncRNA及circRNA)的数据库:exoRBase [2]。数据库连接:http://www.exoRBase.org

exoRBase数据库共收录了58330种circRNAs,15501种lncRNAs,18333种mRNAs,它们来自92份血清外泌体RNA-seq测序样本。作者还依据GTEx project获得组织特异性RNA表达特征,分析了相关RNA分子的组织来源。数据库收录的样本包括健康对照者32例,冠状动脉心脏疾病(CHD)6例,结肠癌(CRC)12例,肝细胞癌(HCC)21例,胰腺腺癌(PAAD)14例,乳腺癌2例。

图2 exoRBase数据库概览 (来自[2])
2. RNase R作用的结构生物学机制
10月3日,Nucleic Acids Research杂志(IF 2016= 10.16)在线发表了台湾中央研究院分子生物学研究所Hanna S. Yuan为通讯作者的文章,介绍解析了RNase R作用的结构生物学机制[3]。

RNase R是常用的3’→5’外切核糖核酸酶,在circRNA研究中经常作为消化线性RNA的工具酶。本文作者解析了来自大肠杆菌的截短型RNase R(87-725 aa)的1.85 Å分辨率的晶体结构,揭示了RNase R同步发挥RNA解链和消化作用的结构生物学机制。
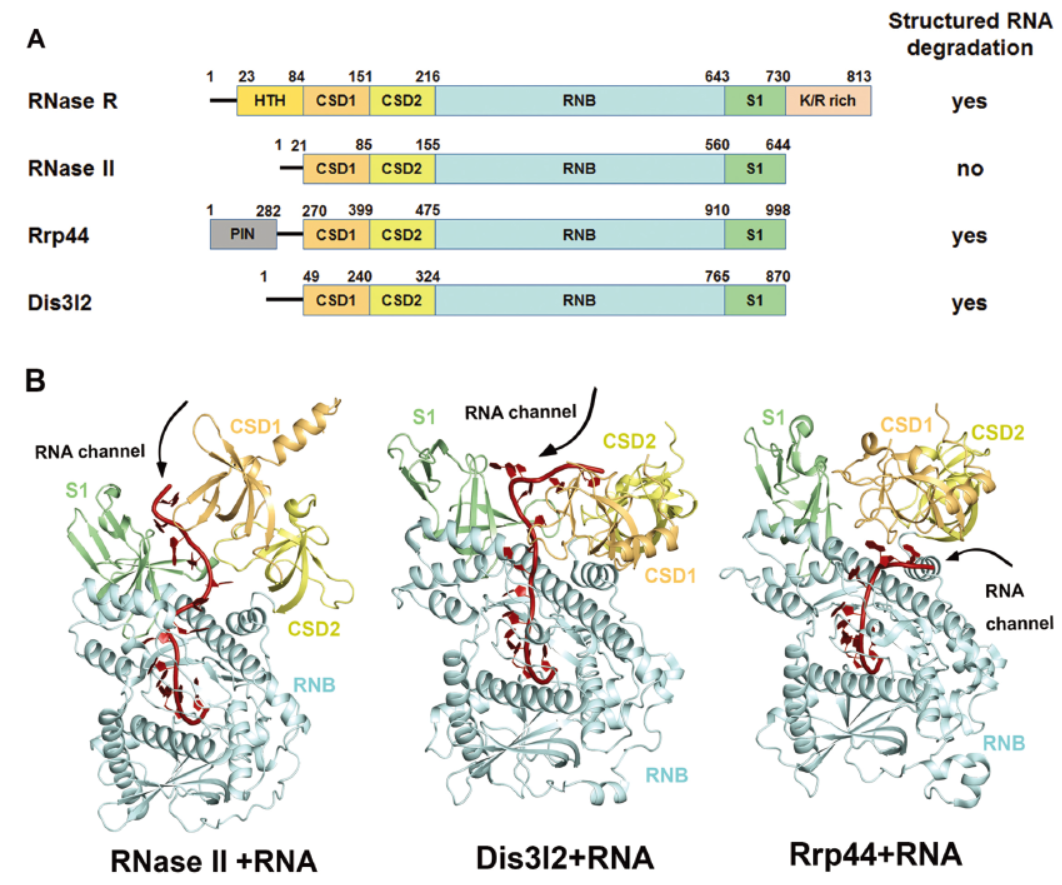
图3 大肠杆菌RNase R的结构生物学机制 (来自[3])
3. circNPM1在急性髓系白血病(AML)中的作用
9月29日,著名血液学杂志Haematologica(IF 2016= 7.702)在线发表了德国乌尔姆大学医学院Lars Bullinger教授为通讯作者的文章,介绍发现circNPM1在急性髓系白血病(AML)中发挥重要作用[4]。

作者发现来自NPM1基因的circRNA hsa_circ_0075001与AML疾病存在相关性。circ_0075001表达状态差异对应于不同的基因表达状态,如高表达的患者TLR通路基因表现为低表达等等。
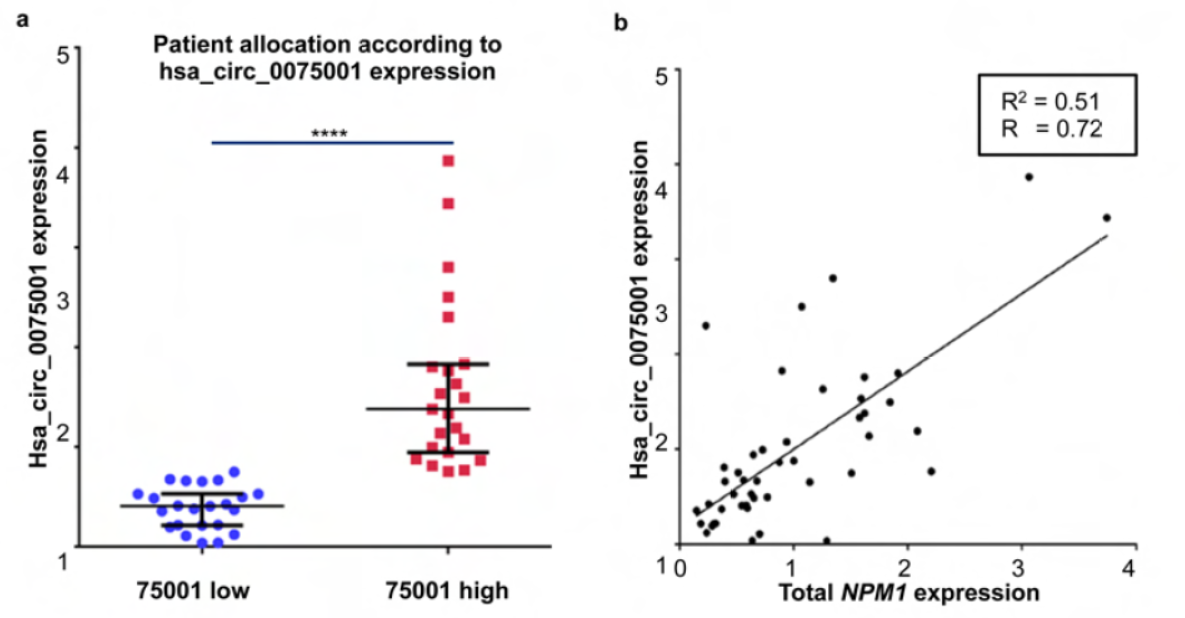
图4 AML病人circ_0075001表达特征 (来自[4])
4. HPV诱发宫颈癌中lncRNA和circRNA表达特征
9月19日,Frontiers in Microbiology杂志(IF 2016= 4.076)在线发表了中科院武汉病毒所Chen Mingyue和崔杰研究员为共同通讯作者的文章,介绍在HPV(人乳头状瘤病毒)感染诱发的宫颈癌中发现疾病相关lncRNA和circRNA[5]。

作者分析了三利HPV16感染诱发的宫颈癌和对应癌旁组织的全转录组,共发现了差异表达的19种lncRNAs,99种circRNAs,28 种miRNAs和304种mRNAs。作者也构建了这些基因的相互作用网络图[5]。
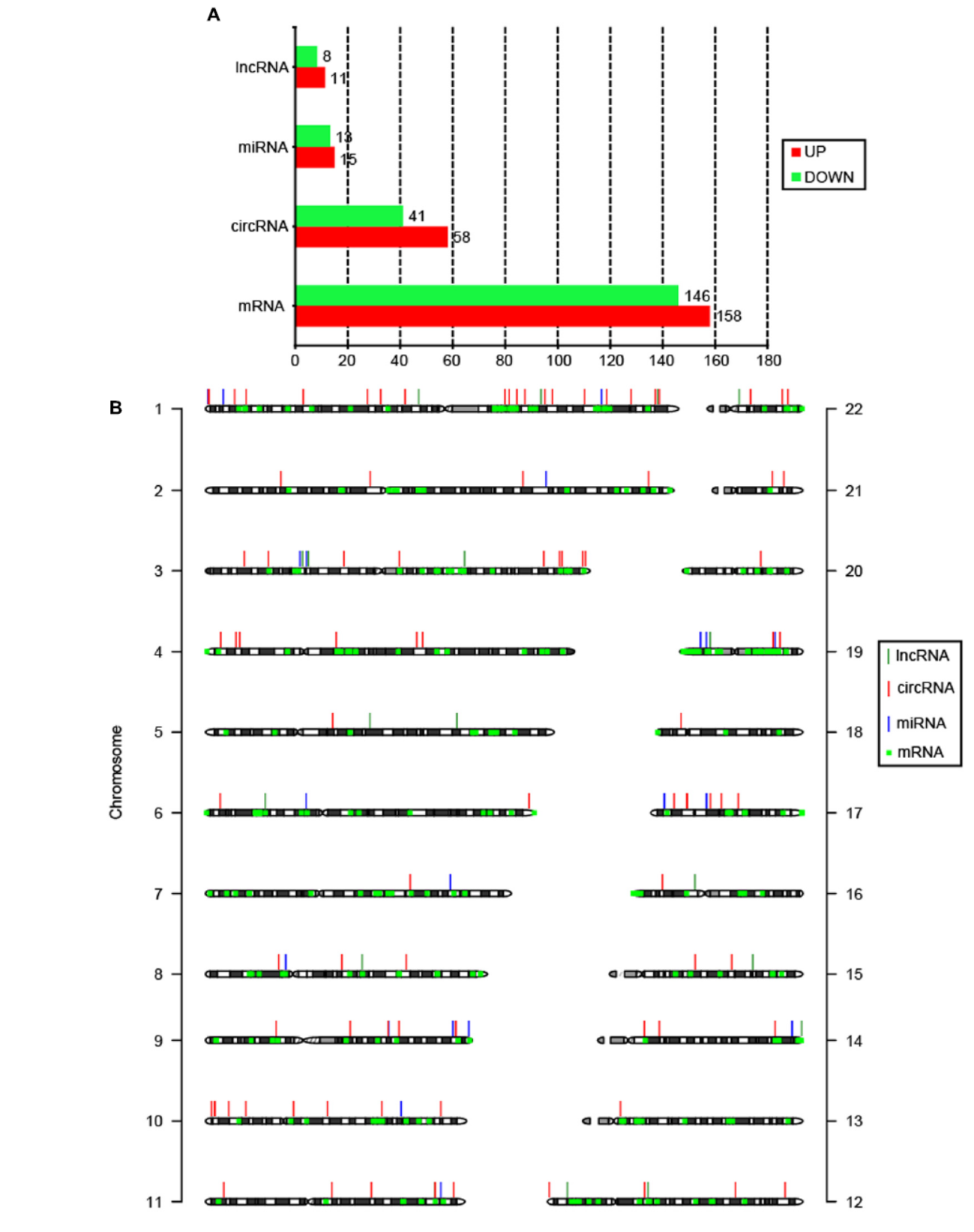
图5 HPV感染宫颈癌中差异表达基因(来自[5])
5. 组织特异性circRNA表达特征分析
10月3日,BMC Genomics杂志(IF 2016= 3.729)发表了美国德州大学休斯敦健康科学中心赵中明教授与南京航空航天大学宋晓峰教授为共同通讯作者的文章,介绍基于RNA-seq数据分析组织特异性circRNA表达特征的工作[6]。

文中共收集了来自成年人和胎儿的六种不同组织(结肠,心脏,肾脏,肝脏,肺和胃)来源的RNA-Seq数据,还收集了四种腺体组织(肾上腺,乳腺,胰腺和甲状腺)的数据。这些数据主要来源于NCBI和ENCODE。以大于2个有效Reads为标准,作者在成年,胎儿和腺体组织中分别分析到了8120,25933 和14433种circRNAs分子。这些circRNAs分子大部分为组织特异性表达的,比例占到36.97–50.04%。
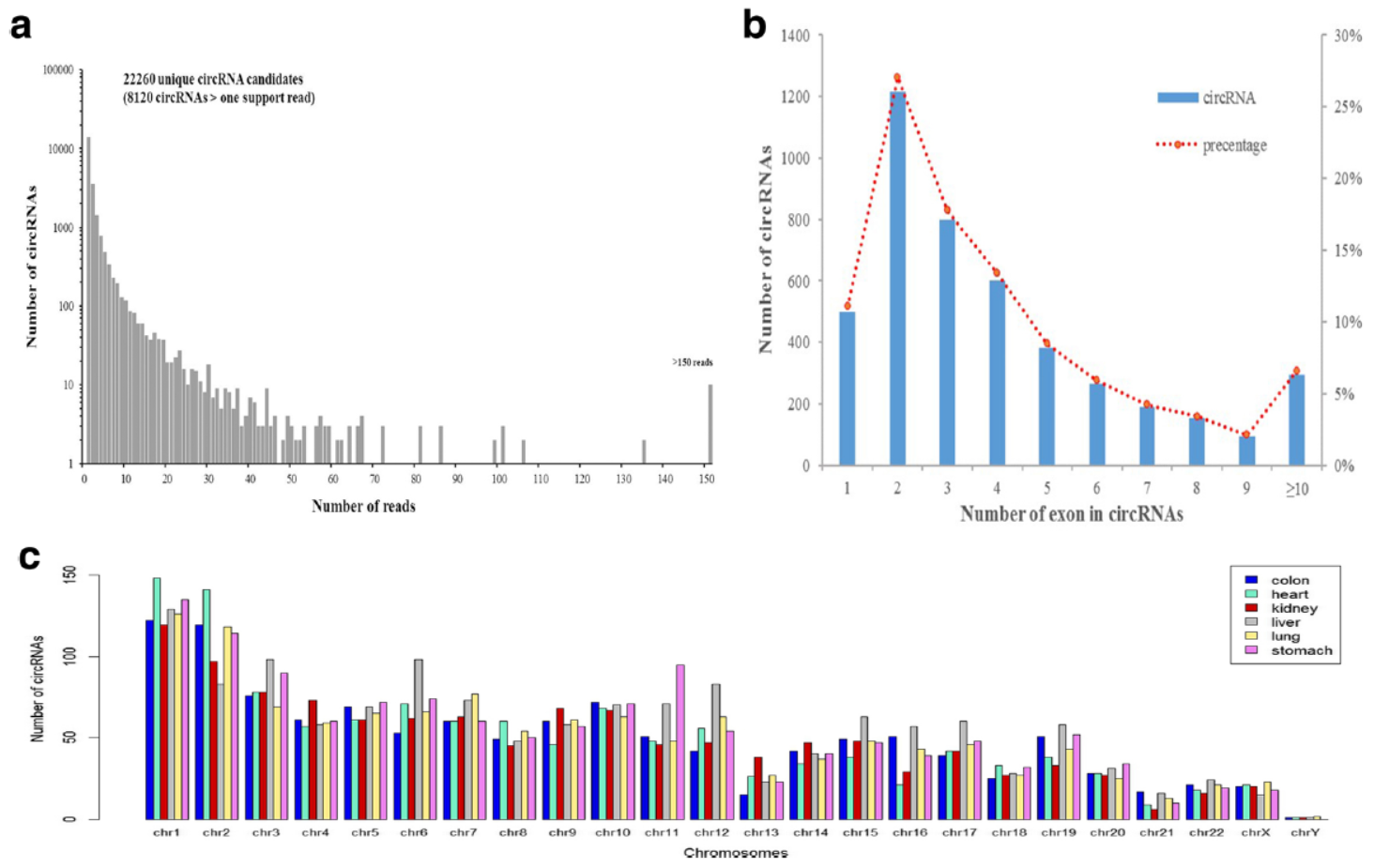
图6 组织特异性circRNAs分子总体分布特征 (来自[6])
6. 胃癌中发现具有癌基因特征的circRNA
10月5日,Cell Cycle杂志(IF 2016= 3.53)在线发表了北大人民医院王杉主任为通讯作者的文章,介绍在胃癌中发现了一组具有癌基因特征的circRNAs分子[7]。

作者利用芯片法分析了胃癌中circRNA和mRNA的表达特征,发现了三个高表达的circRNA分子:hsa_circ_0047905、hsa_circ_0138960 和has-circRNA7690-15。RNA干扰后细胞增殖和迁移能力下降。
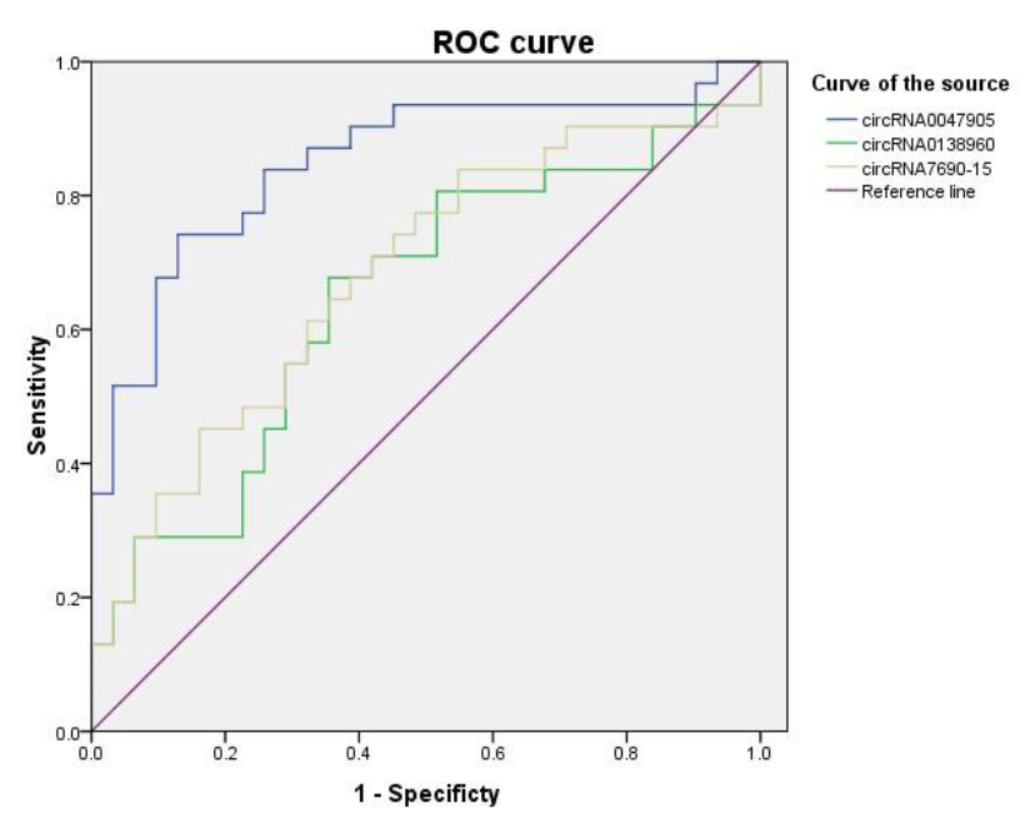
图7 胃癌中癌基因性circRNA的ROC曲线 (来自[7])
7. circRNA作为胃癌标志物
9月14日,world journal of gastroenterology杂志(IF 2016= 3.365)在线发表了安徽医科大学附属安徽省立医院朱志强主任为通讯作者的文章,介绍发现hsa_circ_0000745可作为胃癌诊断标志物 [8]。

作者在20例胃癌和癌旁组织中通过QPCR分析了三种circRNA分子的表达情况,基于通路预测和竞争性结合miRNA的分析,作者将研究聚焦在hsa_circ_0000745上。在60例临床标本的验证中表明hsa_circ_0000745在胃癌中低表达。ROC曲线分析表明将hsa_circ_00 00745与CEA联合检测可获得AUC值0.775。
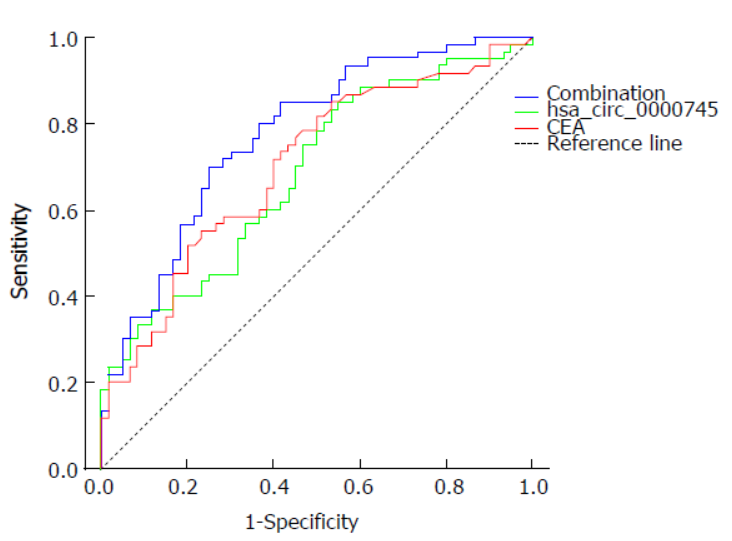
图8 hsa_circ_0000745在胃癌中的ROC曲线 (来自[8])
8. 人类主动脉瓣中circRNA表达分析
10月7日,Gene杂志(IF 2016= 2.4149)在线发表了南京医科大学附属第一医院邵永丰主任为通讯作者的文章,分析了人主动脉瓣中circRNA的表达特征[9]。

本文共分析到5476种circRNA分子,其中1412种为人类主动脉瓣特有的。作者也针对这些circRNA进行了通路分析,RBP结合情况预测等信息学分析。
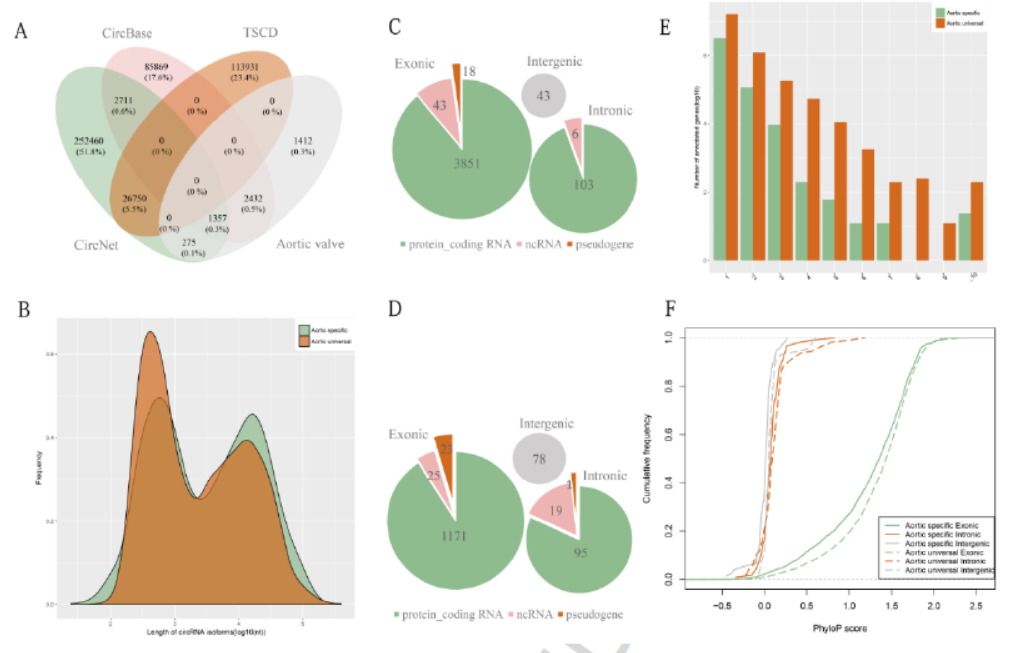
图9人类主动脉瓣中circRNA表达分析 (来自[9])
9. Moyamoya 病中circRNA表达分析
10月15日,Journal of the Neurological Sciences杂志(IF 2016=2.295)北京天坛医院神经外科赵继宗主任为通讯作者的文章,介绍分析了Moyamoya病中circRNA的表达特征[10]。

作者利用芯片法分析了各5例病人与健康对照样品,共得到146种差异显著的circRNA分子,其中29种为上调。文中也针对这些circRNA进行了通路分析。
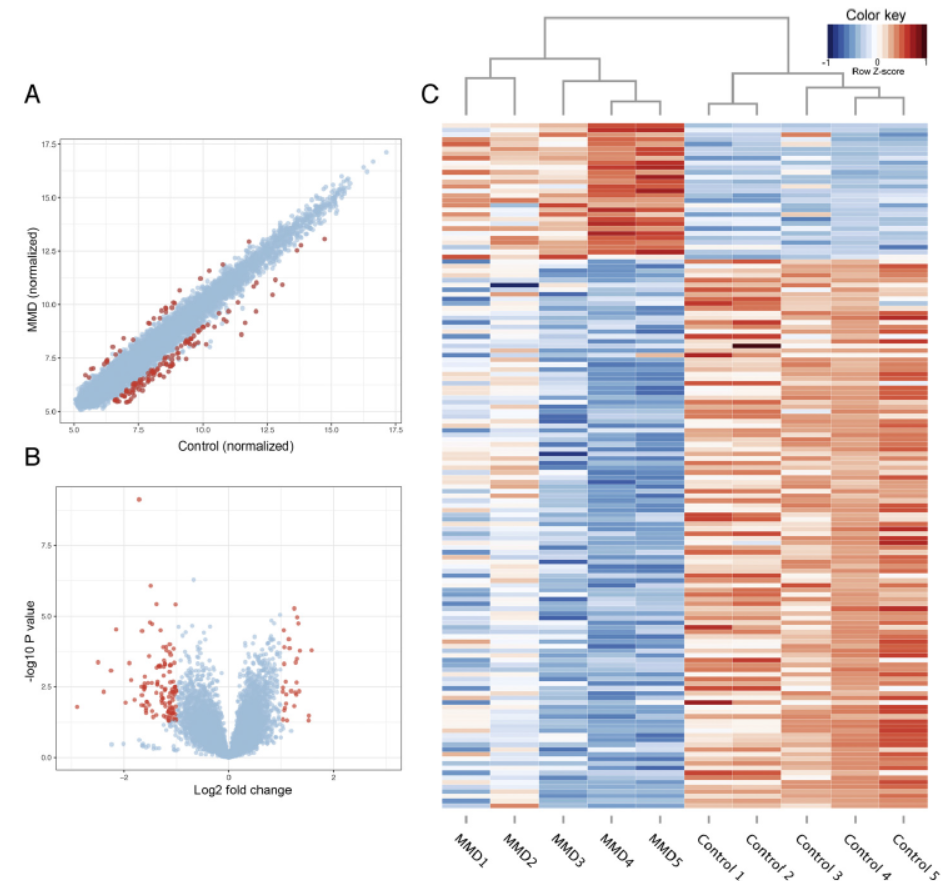
图10 Moyamoya 病中circRNA表达 (来自[10])
10. 类风湿性关节炎PBMC中circRNA表达分析
9月27日,Molecular Medicine Reports杂志(IF 2016= 1.692)在线发表了深圳市人民医院(暨南大学第二临床医院)戴勇主任为通讯作者的文章,分析了类风湿性关节炎患者外周血单个核细胞(PBMC)中circRNA表达特征[11]。

本文利用芯片法进行分析了各10例类风湿性关节炎患者和健康对照者的外周血单个核细胞样品,共得到了584种差异表达的circRNA分子,其中255种为上调。
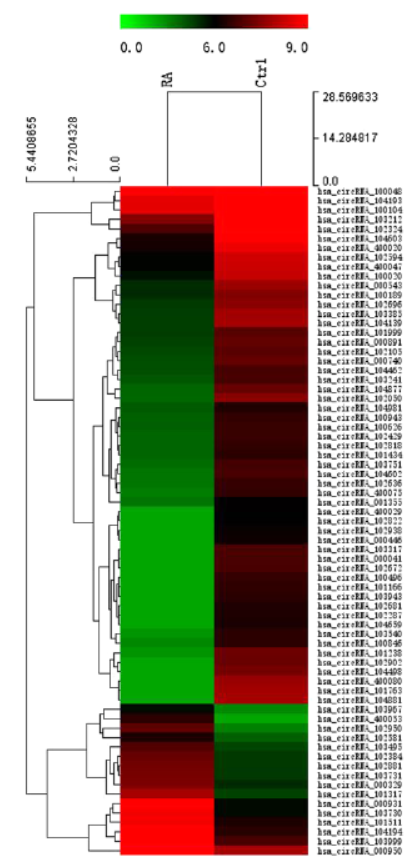
图11 类风湿性关节炎PBMC中circRNA差异表达 (来自[11])
11. 植物相关circRNA研究进展
拟南芥全基因组circRNA分析
9月27日,Frontiers in Plant Science杂志(IF 2016= 4.2986)在线发表了扬州大学农学院金飚为通讯作者的文章,介绍将拟南芥中circRNA进行全基因组定位分析[12]。

作者利用链特异性测序分析了不同发育阶段,应激条件下circRNA的表达情况,做成了拟南芥基因组中circRNA的全基因组定位图。共得到5861种circRNA的信息,其中1275种为新发现的。
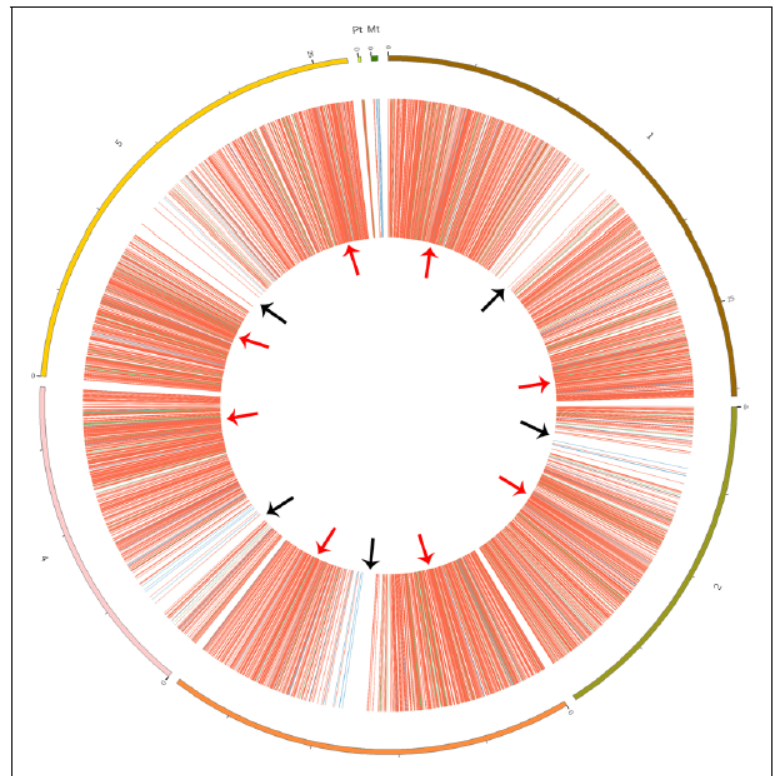
图12 拟南芥circRNA的全基因组定位 (来自[12])
棉属多倍体物种及其祖先中保守circRNA表达特征分析
10月3日,FEBS Letters杂志(IF 2016= 3.519 )在线发表了南京农业大学关雪莹和周宝良为共同通讯作者的文章,介绍在棉属多倍体物种及其祖先中保守性circRNA的表达特征分析[13]。

本文主要利用去rRNA后建库分析,RNA-seq后分析得到相关circRNA信息,其中发现了23种circRNA为非GT/AG剪切形成的,并且只有约10%的外显子来源的circRNA在两侧内含子中存在反向互补元件,这些特征说明在植物中可能存在与动物不同的circRNA形成机制。
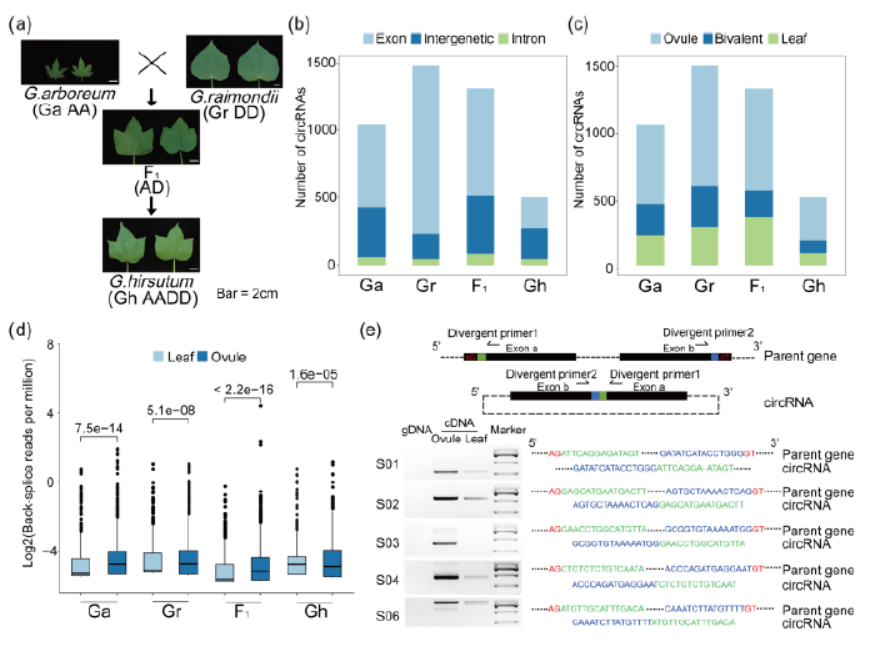
图13 棉属多倍体物种及其祖先中保守circRNA表达特征 (来自[13])
12. circRNA相关综述与观点
Oncogene发表重要circRNA综述
10月9日,Oncogene杂志(IF 2016= 7.519)在线发表了丹麦奥胡斯大学Lasse Sommer Kristensen 教授为通讯作者的综述文章,著名RNA研究专家Jørgen Kjems教授也是本文的共同作者之一[14]。
本文中作者系统汇总了目前关于circRNA研究的主要进展,重点汇总了circRNA作为肿瘤标志物的进展,还针对circRNA的功能研究,将可能具有抑癌基因特征和癌基因特征的circRNA分子进行了简单的分类。关于今后circRNA研究需要解决的问题,文章给出了非常中肯的建议,其中包括circRNA的标准化命名问题,肿瘤特异性circRNA表达特征形成的机制,circRNA的修饰等问题。提高RNA-seq的样品制备质量和分析精度也是需要继续改进的方向。
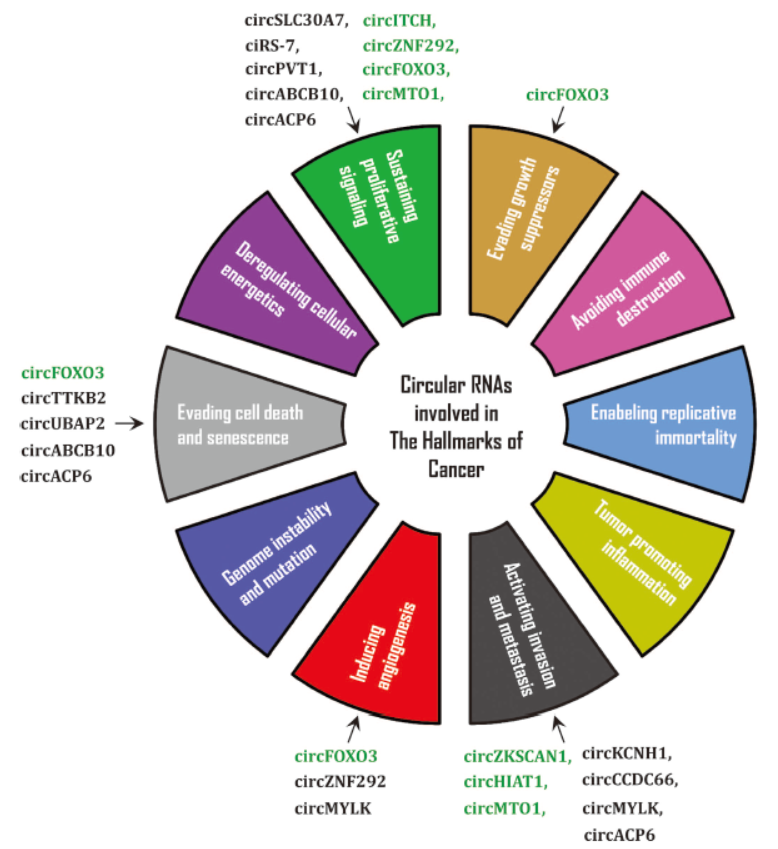
图12 可作为肿瘤标志物的circRNA分子 (来自[14])
Jørgen Kjems教授发表circRNA研究综述
10月6日,RNA Biology杂志(IF 2016= 3.9 )在线发表了知名RNA研究专家Jørgen Kjems教授为通讯作者的综述文章。本文主要针对即将出版的关于circRNA的专业书作的序(文中未提及书是谁撰写的,书题目是什么,估计不久将会公开)。在这篇序中,Jørgen Kjems教授教授大致分析了目前circRNA研究的热点和焦点问题,其中包括竞争性结合miRNA或蛋白的功能模型,还特别提到了今年新发现的circRNA可翻译蛋白的机制,也提到了包括circRNA形成机制,从细胞核中转运至胞浆的机制等目前circRNA研究普遍关注的重要问题。文章最后还提到circRNA作为药物用于临床的可能性 [15]。

OncoTarget发表circRNA综述
9月8日,Oncotarget杂志(IF 2016= 5.1657)发表了circRNA竞争性结合miRNA相关进展的综述,文章的通讯作者为郑州大学附属第一医院Duan Zhenfeng。

胃肠癌中circRNA综述
10月3日,Oncology Letters杂志(IF 2016= 1.3899)在线发表了北京协和医院由磊和赵玉沛为共同通讯作者的综述,汇总了胃肠癌中circRNA的相关研究进展[16]。

参考文献:
1. Siyu Xia, J.F., Ke Chen, Yanbing Ma, Jing Gong, Fangfang Cai, Yuxuan Jin, Yang Gao, Linjian Xia, Hong Chang, Lei Wei, Leng Han and Chunjiang He, CSCD: a database for cancer-specific circular RNAs. Nucleic Acids Research, 2017.
2. Shengli Li, Y.L., Bing Chen, Jingjing Zhao, Shulin Yu, Yan Tang, Qiupeng Zheng, Yan Li, Peng Wang, Xianghuo He and Shenglin Huang, exoRBase: a database of circRNA, lncRNA and mRNA in human blood exosomes. Nucleic Acids Research, 2017.
3. Lee-Ya Chu, T.-J.H., Bagher Golzarroshan, Yi-Ping Chen, Sashank Agrawal and Hanna S. Yuan, Structural insights into RNA unwinding and degradation by RNase R. Nucleic Acids Research, 2017.
4. Hirsch, S., et al., Circular RNAs of the nucleophosmin (NPM1) gene in acute myeloid leukemia. Haematologica, 2017.
5. Wang, H., et al., Identification of Novel Long Non-coding and Circular RNAs in Human Papillomavirus-Mediated Cervical Cancer. Front Microbiol, 2017. 8: p. 1720.
6. Tianyi Xu, J.W., Ping Han, Zhongming Zhao and Xiaofeng Song, Circular RNA expression profiles and features in human tissues: a study using RNA-seq data. BMC Genomics, 2017.
7. Lai, Z.Y., et al., Analysis of co-expression networks for circular RNAs and mRNAs reveals that circular RNAs hsa_circ_0047905, hsa_circ_0138960 and has-circRNA7690-15 are candidate oncogenes in gastric cancer. Cell Cycle, 2017: p. 0.
8. Huang, M., et al., Circular RNA hsa_circ_0000745 may serve as a diagnostic marker for gastric cancer. World J Gastroenterol, 2017. 23(34): p. 6330-6338.
9. Chen, J., et al., Identification of circular RNAs in human aortic valves. Gene, 2017.
10. Zhao, M., et al., Altered expression of circular RNAs in Moyamoya disease. J Neurol Sci, 2017. 381: p. 25-31.
11. Zheng, F., et al., Circular RNA expression profiles of peripheral blood mononuclear cells in rheumatoid arthritis patients, based on microarray chip technology. Mol Med Rep, 2017.
12. Chen, G., et al., Genome-Wide Identification of Circular RNAs in Arabidopsis thaliana. Front Plant Sci, 2017. 8: p. 1678.
13. Zhao, T., et al., Characterization of conserved circular RNA in polyploid Gossypium species and their ancestors. FEBS Lett, 2017.
14. Kristensen, L.S., et al., Circular RNAs in cancer: opportunities and challenges in the field. Oncogene, 2017.
15. Hansen, T.B. and J. Kjems, The invasion of circRNAs. RNA Biol, 2017: p. 0.
16. XIAOXIA REN, Y.D., LEI YOU and YUPEI ZHAO, Potential functions and implications of circular RNA in gastrointestinal cancer. Oncology Letters, 2017.
.png)