Circular RNA (circRNA)自大约1979年被发现,之后的30年里几乎无人问津,直到近几年出现了爆发性的研究增长,研究热点也已从开始的大规模筛选鉴定逐渐转向特定基因的功能和机制研究。
除了CDR1as(ciRS-7)和circ-FOXO3等少数几个基因有多篇文章报道其功能和作用机制外,大量cricRNAs的功能研究还是空白,这其中过表达(功能获得性研究,gain of function)自是避不开的重中之重。不同于mRNA,circRNA由于其形成的复杂性和表达的多态性,对其过表达有更大的难度,大部分同学也都碰到了各种各样的问题。
综合文章和其他人的反馈来看,circRNA过表达主要三个难点:1)成环效率:不同的外源成环框架,对于不同的基因的过表达效率也有差异。部分基因特别是序列偏长或偏短,二级结构复杂的circRNA有可能无法成环或成环效率低;2)成环准确率:使用外源的成环框架容易,但保证准确剪切环化,不把酶切位点或载体序列环化进去却成了最大的难点;3)特殊基因:特殊的序列来源或非AG-GT剪切环化模式的circRNA用多种方法都不能实现准确高效的过表达。
湖人今天就为大家支几招,介绍具体怎么构建circRNA的过表达载体,明天还将介绍怎么严谨地验证是否过表达成功。
第一招:拿来主义
“你能想到的,别人早都想到了,而且还发了文章。”研究生几年里听到最多的大概就是这句话了,每每兴奋有个奇思妙想,最后总是碰一鼻子灰,毕竟研究既要novel又要有mechanism可一点都不容易,这时候拿来主义就显得比较有用了。我做不好,总有人做的专业呀!
目前国内能提供circRNA过表达业务的公司很多,都有现成的方案和空载体,直接拿来用就行了。这里我以自己用过的某公司产品pLCDH-ciR举例,据介绍该载体基于模拟内源性的环化模式开发,由两个通用的成环框架介导环化和剪切,框架序列包括一段反向重复(Alu)序列和一段效应QKI蛋白的序列,以及一段人工修改的剪切介导序列,整体框架可使插入的环状RNA序列高效率环化,并精确地在AG-GT位点严格剪切。应该就是和内源的侧翼Alu序列介导环化一个原理,不过特殊设计了序列,以使对不同的插入序列都能介导环化,并且可以准确剪切,逻辑思路还是很清晰的。
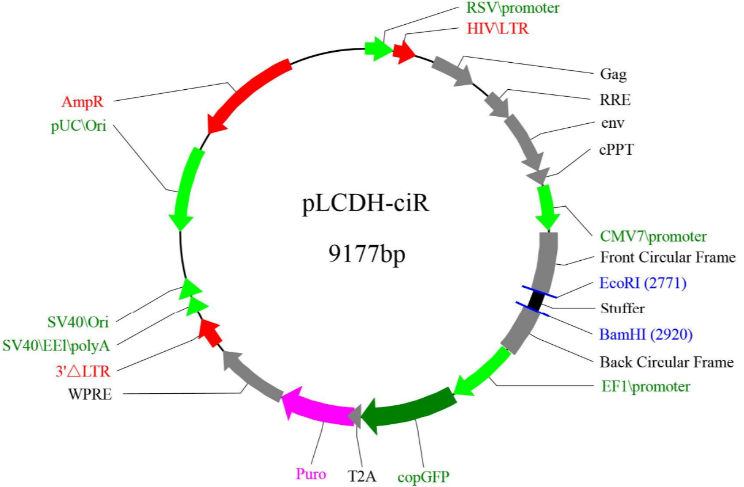
该载体使用需要先扩增目的circRNA序列,在5’和3’端加上一段介导序列(听说可以调控准确剪切)和两个酶切位点EcoRI/BamHI,然后做酶切连接。这和其他载体构建是一样的流程,几天就可以构建完成,还是很简便的,重点是通用性高,随便挑个基因都可以这样做,很适合同时做多个基因,拿来用之,省时省力呀!
第二招:他山之石
读文献,读文献,读文献,重要的事情说三遍。这里要介绍的第二招就是看文章来的,在Short intronic repeat sequences facilitate circular RNA production(Genes Dev. 2014;28:2233–2247)这篇文章里,作者分析了circ-ZKSCAN1的侧翼序列和Alu元件,首先克隆ZKSCAN1的exon2/3和侧翼序列共2232bp来构建载体,成功过表达了circ-ZKSCAN1。接着对侧翼序列具体分析,一段一段地删除序列再检测基因过表达情况,以得到真正决定成环的侧翼序列区,最终保留5’端侧翼序列87bp,3’端侧翼序列59bp,并将ZKSCAN1的exon2/3替换成一段多克隆酶切位点,构建了一个空载体pcDNA3.1(+) CircRNA Mini Vector。
pcDNA3.1(+) CircRNA Mini Vector示意图:
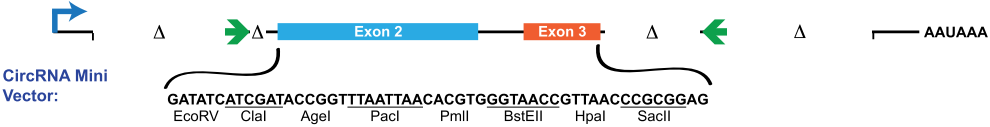
该载体使用也是以酶切位点连接目的circRNA序列,但可能由于侧翼的成环序列比较短,介导成环能力有限。我做过的几个基因过表达倍数普遍不高,还有错误成环,把框架序列也加到成环产物里了的,这样的恐怕不能往下做。想使用这个载体的同学,建议去掉酶切位点,直接把成环序列加到你circRNA的两端,这样应该可以保证成环准确。
第三招:回本溯源
前面两招都是使用通用的载体,其成环序列是模拟和替换内源性的模式,这里第三招回本溯源就要介绍使用基因天然的侧翼序列构建载体了。很明显几乎所有circRNA成环都和本身的侧翼序列直接相关,那我们把基因和其本身的侧翼序列一起克隆出来,放到载体上自然也应该可以过表达。
对于侧翼序列的选择,最理想的情况是上下游1kb内都有明显的重复元件(Alu),克隆到的侧翼序列含两个或以上重复元件分布在circRNA的上下游,可以促使成环和剪切,这和体内天然的形成模式是一致的。

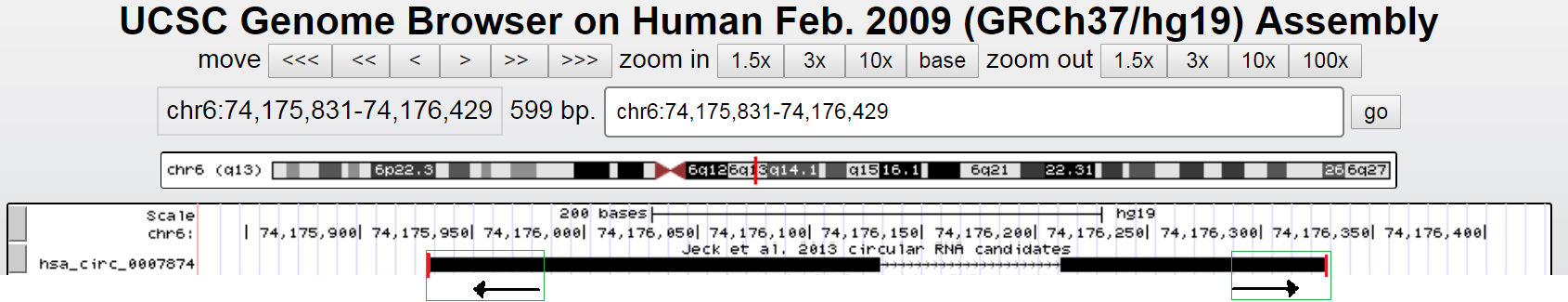
这里的has_circ_0007874由两个外显子组成,长度318bp,我向上下游各延伸1kb,在Repeat Element处可以看到都有明显的重复元件,下一步导出序列,只截取上下游各延长550bp即可,过长的序列可能导致克隆难度增加,这里550bp内有重复元件,肯定够用了。序列里下划线标记的就是延伸的侧翼序列,其中小写代表重复元件,红色标记的AG-GT是剪切位点。可以PCR法或基因合成克隆这一段1418bp序列到载体上(如pcDNA3.1)就可以过表达has_circ_0007874了。
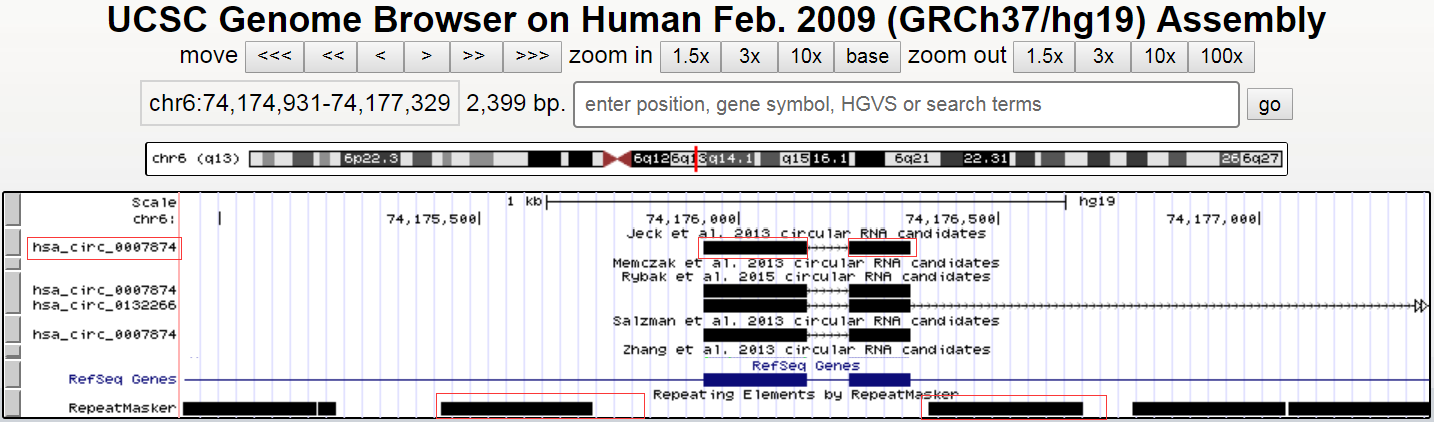
>hg19_dna range= chr6:74175382-74176879 5’pad=0 3’pad=0 strand=+ repeatMasking=lower
AAACAGTAAAAATATGCATCAAGGAAGAGATTCAAAAGAAAGTTCAggccgggcgcggtggctcacggctgtaatcccagcactctgggaggccgaggtgggcagatcacctgaggtcaggagtttgagaccagcctggtcgacatggtgaaacccgtctaccaaaaatacaaaaattagccaggcgtggtgccggacacctgtaatcccggctactcgggatgctgaggcaggagaatcgcttgaacccaggaggcagaggttgcagtgagccgagattgtaccactgcactccagcctgggtgacagagtgagactccatcttaaaaaaaacaaaaGGAAGTTCAGGAAGTGGTTTGTCATCCAGGTTGCCCTGGACACTTTTGCATCCCATTGGGTCTTTGGGATTTTCTTTGTAGTATATCTTTCACGTTTTCTATTTTTTATCATATGAGATGATTATAGTCACCTGTATAAATGTTTCCTTATTAAGTAGTCACTATGTGCAAGGTGCTCATATATTTATTTTGTTTATGTCTATTATCTTTAGGTCAGATGTCATGTAATCCTTCCTTTGGTGGCATCGGAAAGGGACATTTAATGAGGGAAGTAGATGCCTTGGATGGCCTGTGTTCTCGCATCTGTGACCAGTCTGGTGTACATTATAAAGTATTAAACCGGCGTAAGGGACCAGCTGTGTGGGGTCTGAGAGCTCAGATTGATAGGAAACTCTATAAACAGAACATGCAGAAAGAAATCTTGAATACACCACTGCTTACTGTTCAGGAGGGAGCTGTAGAAGATCTTATTCTTACAGAACCAGAGCCTGAACACACTGGGAAATGCCGTGTCAGTGGGGTTGTTTTGGGTACGTATTGGTTATAGATGGTGTATGATAACAGCatatcaaattccatgtgagaaatttattttagagcaaagtgtggagatatgttgcttattcaaggaacttgttgcattttctctacctggatgaggaaaactatataaataaatatgtttctcttttcatcttggcatttatgatgctcagtcaaattctaggctcatcagtaccatgttccatcttagtgtagaactttgtaatctgcatatttatgtctccctctctccccatctttctattgtaatttctgttttttctggttctgactttaattttttaaaaaattcatattttAACAGCTTATATATATTTTTATGTAAAAGAGTTATGTATGTTTTTTATCAAAATAACTCATGCATATAATTTTTTCTTAAAATATTAAAAGGGTctgggcatggtggctcatgcccgtaatcctagcactttgggaggccgaggtaggtggattgcttgagccccagagtttgagaccagcctcggcaagatggcaaaacctcatgtctacaaaaaa
PS:此处仅以has_circ_0007874为例说明分析和确定侧翼成环序列的思路,我自己没有实际做过这个基因。
第四招:移花接木
前面讲了先模拟再通用,又介绍了基因特异的成环方案,这里第四招移花接木要介绍将基因特异的成环方案换成一个通用的方案。对于cricRNA过表达,总是依赖侧翼序列促使成环来得方便,那我们是否可以将has_circ_0007874的侧翼序列拿去过表达其他基因呢?答案是肯定的。
具体做法也简单粗暴,把第三招里的全长序列复制下来,接着把内部AG-GT之间的has_circ_0007874序列换成你要研究的circRNA,也即替换中间的NNNN,然后构建载体,就可以成功过表达啦。
AAACAGTAAAAATATGCATCAAGGAAGAGATTCAAAAGAAAGTTCAggccgggcgcggtggctcacggctgtaatcccagcactctgggaggccgaggtgggcagatcacctgaggtcaggagtttgagaccagcctggtcgacatggtgaaacccgtctaccaaaaatacaaaaattagccaggcgtggtgccggacacctgtaatcccggctactcgggatgctgaggcaggagaatcgcttgaacccaggaggcagaggttgcagtgagccgagattgtaccactgcactccagcctgggtgacagagtgagactccatcttaaaaaaaacaaaaGGAAGTTCAGGAAGTGGTTTGTCATCCAGGTTGCCCTGGACACTTTTGCATCCCATTGGGTCTTTGGGATTTTCTTTGTAGTATATCTTTCACGTTTTCTATTTTTTATCATATGAGATGATTATAGTCACCTGTATAAATGTTTCCTTATTAAGTAGTCACTATGTGCAAGGTGCTCATATATTTATTTTGTTTATGTCTATTATCTTTAGNNNNGTACGTATTGGTTATAGATGGTGTATGATAACAGCatatcaaattccatgtgagaaatttattttagagcaaagtgtggagatatgttgcttattcaaggaacttgttgcattttctctacctggatgaggaaaactatataaataaatatgtttctcttttcatcttggcatttatgatgctcagtcaaattctaggctcatcagtaccatgttccatcttagtgtagaactttgtaatctgcatatttatgtctccctctctccccatctttctattgtaatttctgttttttctggttctgactttaattttttaaaaaattcatattttAACAGCTTATATATATTTTTATGTAAAAGAGTTATGTATGTTTTTTATCAAAATAACTCATGCATATAATTTTTTCTTAAAATATTAAAAGGGTctgggcatggtggctcatgcccgtaatcctagcactttgggaggccgaggtaggtggattgcttgagccccagagtttgagaccagcctcggcaagatggcaaaacctcatgtctacaaaaaa
第五招:无招胜有招
前面四招我相信基本可以覆盖所有circRNA了,一招不成功就换另一招。可能还有些同学饱受deadline的摧残,赶时间毕业或评职称,特别一些学临床的没有条件和资源做实验,给你们的第五招就是请外援,找公司!毕竟我们讲专业的事交给专业的人做,你没时间做,没实验条件,或者总是做不成功的,都可以尝试找公司做。我自己有接触过一两家,还是很靠谱的,需要做的就是提供基因信息,然后等结果,看似无招实则胜过所有招数啊。
好了,上面的五种招数应该够用,湖人也祝所有同学都可以成功。明天我将继续讲解怎么严谨地验证是否过表达成功,我们江湖再见!
看过这篇文章的人,做过表达都成功了(下)
古月湖人
昨天湖人为大家支了五招,讲怎么构建circRNA过表达载体,今天就接着介绍怎么严谨地验证是否过表达成功。
构建好载体之后,自然是验证能否成功(正确)过表达了,常用的思路是瞬时转染细胞系,24-48h后取样做RT-qPCR检测,以2-△△Ct法计算表达差异。而对于circRNA过表达来讲,因为其准确成环难度大,转录后还要经过剪切环化,仅仅做常规的RT-qPCR计算表达差异是不够的,而是一定要将PCR的产物进行电泳,检测是否单一条带,条带大小是否正确,并且进行sanger测序,比对PCR产物的序列是否和基因一致,确保环化位点处准确成环,没有错误添加进酶切位点或缺失序列。
此处还有一个难点,circRNA由于其反向剪切后环化位点(Splice Junction)在基因组上mapping是前后相反的,所以检测circRNA的特异引物一般设计为Divergent primers,这和用来检测线性基因的Convergent primers相对应。多数文章里描述Divergent primers是相反方向设计,上下游引物分别在环化位点的两端,使扩增产物内包含环化位点。但这样设计的引物具体位置和扩增特异性问题大都语焉不详,相信很多同学也发现这样设计引物总是有非特异条带或者扩增无条带的情况,稀里糊涂的做就不可取了。这里的重点就是,针对检测circRNA过表达,需要同时设计两种Divergent primers,产物要经电泳确认大小,并且进行sanger测序。
首先来看一下引物设计,第一种普通的Divergent primers可参考大部分文章(如环状RNA引物设计链接),一般设计成背对背,使环化位点包含在产物中间,除了需要将序列首尾拼凑成环化后的形式外,和普通的引物设计基本一样的思路。设计出来大概是这样的:
对应在外显子上是这样的,此时扩增产物是绿色框选部分,中间包含一个环化位点(红色标记)。
这种引物设计思路没有问题,但如果是下图的情况呢?
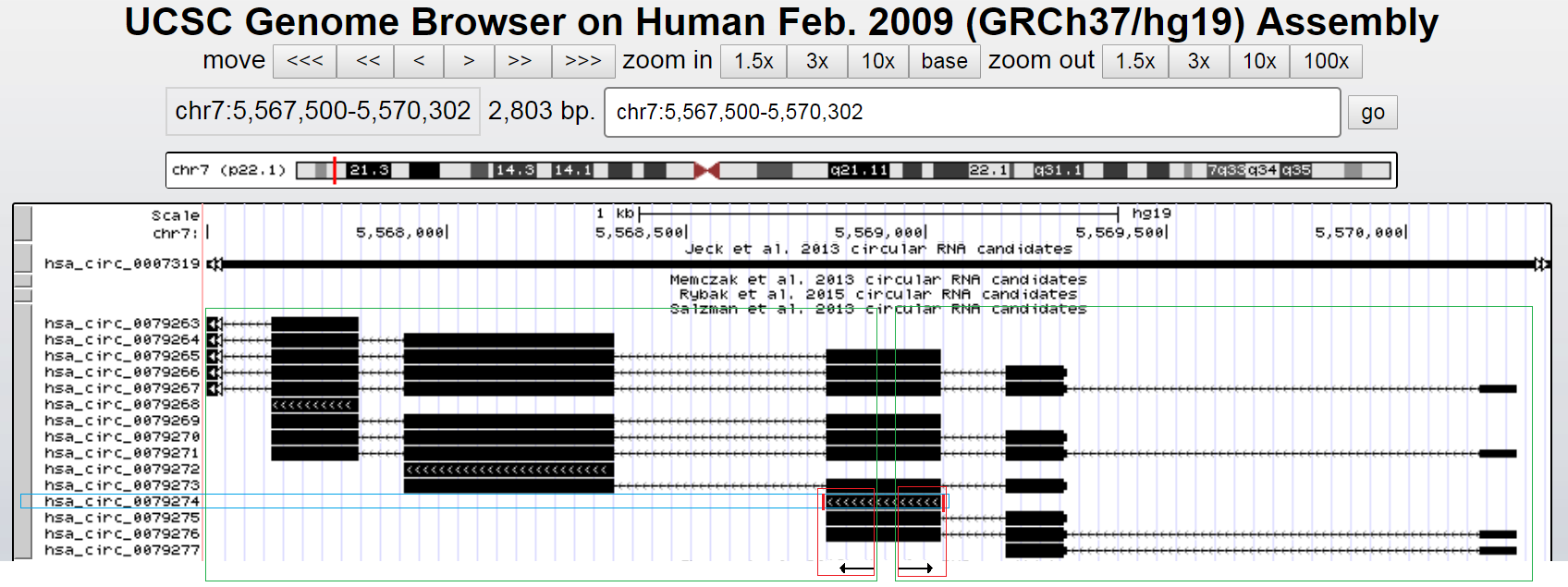
上面的情况是最常见的,因为circRNA的反向剪切有高度的可变性,可能是exon4单独成环,可能是exon4/5成环,也可能是exon3/4/5或者exon2/3/4/5/6成环,包含exon4的circRNA有多个,此时按上面的方法对has_circ_0079274设计引物,预期扩增红色框选部分,但实际上引物可以同时扩增到exon4/5、exon3/4/5或者exon2/3/4/5/6等成环产物(绿色框选),那么引物的特异性就没办法保证了,PCR产物电泳检测很可能是多条带,所以就需要第二种引物了。
第二种引物我自己叫做跨环化位点的引物(Splice Junction Overlapping Divergent Primers,Sjod Primers),主要基于两点考虑:1)不同circRNA的环化位点处序列肯定是不一样的;2)引物的3’端容错率低,有错配会直接影响扩增效率。Sjod Primers设计需要将环化位点放在一条引物的3’端,以使环化位点包含在引物上,而不是包含在PCR产物中,PCR时这条引物只有匹配目的circRNA才能正常扩增,而有错配时基本不能扩增,以此大大提高特异性。Sjod Primers设计出来大概是这样的:
实际设计时可根据具体序列将环化位点放在上游或下游引物的3’端皆可,另一条引物按一般方法设计,确保两条引物是背对背方向即可。Sjod Primers的3’端跨越环化位点几个碱基是一个关键的因素,一般设计跨越3-8个碱基效果最好,跨越碱基过少,可能PCR时被忽略错配差异,无法精确匹配环化位点;跨越碱基过多特别是超过10nt时,可能只由3’端部分序列主导PCR过程,此时引物的5’端相当于悬挂接头,不在PCR过程中匹配模板,最终扩增产物大部分是线性基因的序列,会导致假阳性结果。
再回去看看刚才的图,如果分别对exon4、exon3/4/5或exon2/3/4/5/6的成环产物设计Sjod Primers,即使基因有部分序列相同,也可保证每个circRNA的引物都不同,确保特异性。
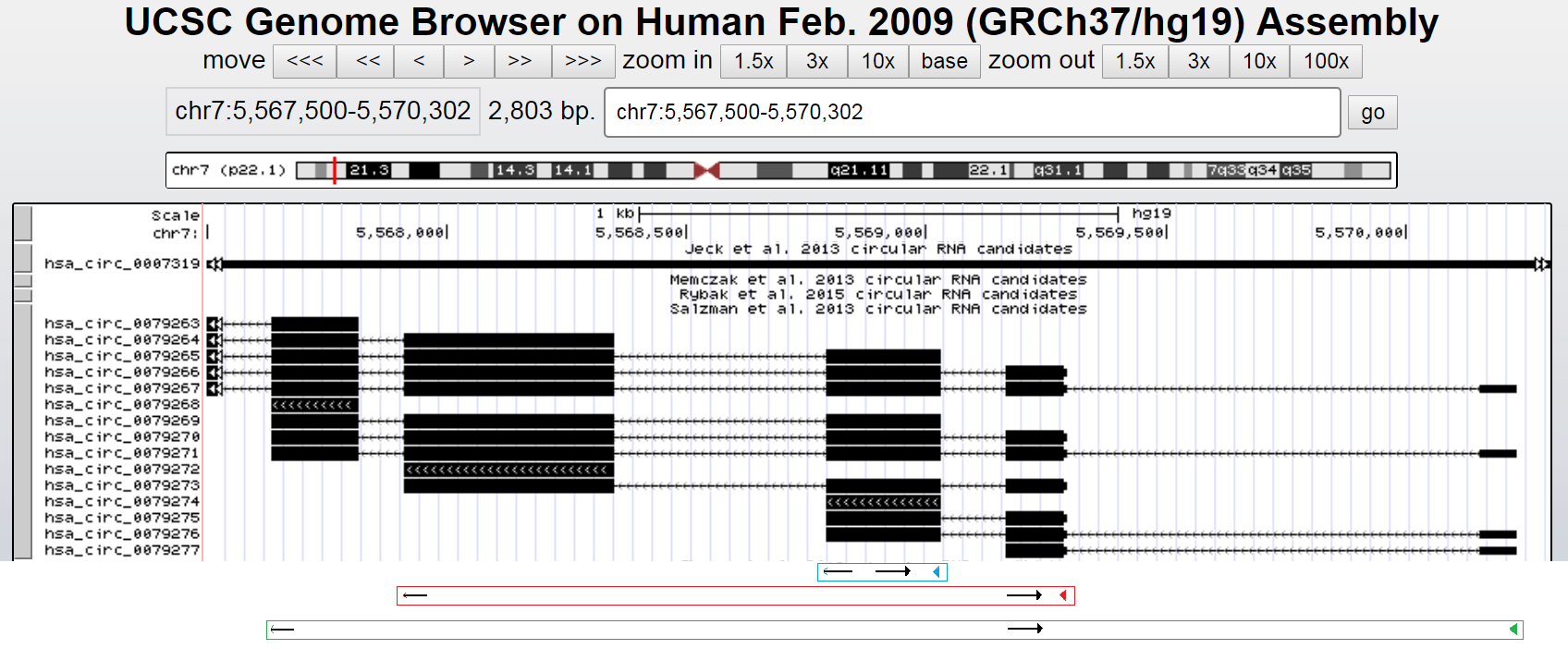
以上两种引物设计好后也应先用样品扩增验证一下,并最好对PCR产物进行sanger测序,以确认扩增产物是正确的。
接下来检测circRNA过表达是否成功,需要同时用到普通的Divergent Primers和Sjod Primers两对引物,其中1)普通的Divergent Primers扩增产物在过表达组样品中应该是单一条带且大小正确,在NC中可能不是单一条带;2)Sjod Primers扩增产物在过表达样品和NC组都应该是单一条带且大小正确。具体步骤如下:
- 同时用普通的Divergent Primers和Sjod Primers进行RT-qPCR,扩增曲线要正常,熔解曲线是单一峰,数据计算有过表达,一般50倍以上就是比较好的结果。无条件的也可以做半定量。
- 分别对Divergent Primers和Sjod Primers的PCR产物做电泳检测,确认产物是单一条带,并且大小正确。
- 过表达组样品使用Sjod Primers扩增的PCR产物进行sanger测序,以确认环化位点处是正确的。此时若有环化错误,在测序结果中是可以发现的。

先来看一个成功过表达的,使用Sjod Primers扩增条带单一且大小正确,使用普通Divergent Primers扩增条带同样单一且大小正确,PCR产物后经sanger测序确认环化位点处序列完全正确,电泳图也显示两对引物扩增产物都有过表达。
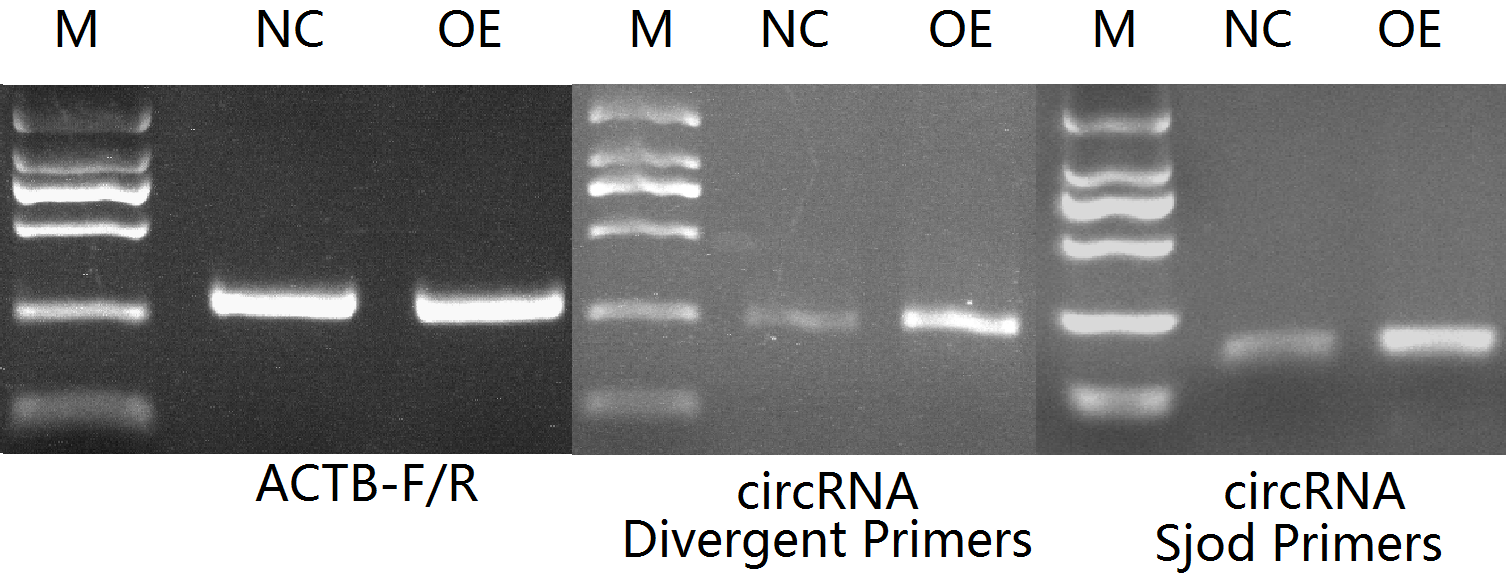
下面是一个错误环化的例子,使用Sjod Primers扩增条带单一且大小正确,但观察不到明显的过表达。使用普通Divergent Primers扩增条带显示NC样品中约250bp大小正确,但OE样品中条带明显偏大,后经sanger测序确认OE中将载体上含酶切位点的一段序列错误环化进去了。
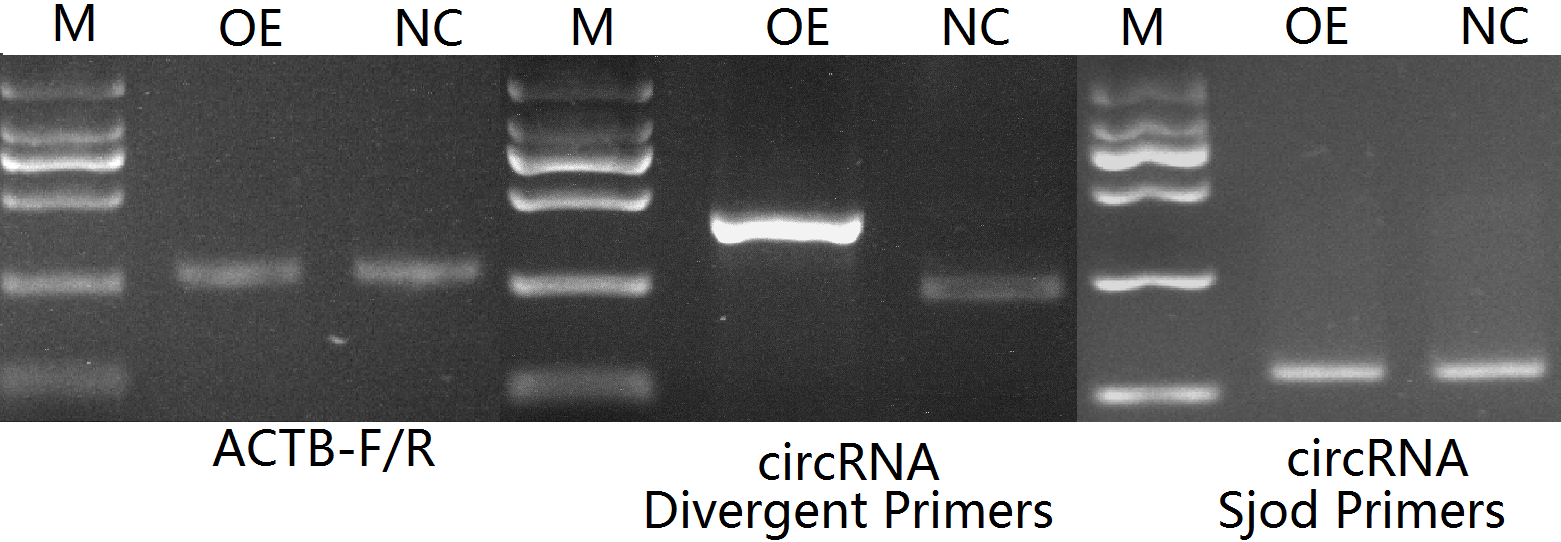
综合来讲,和检测其他线性基因过表达不同,对circRNA的过表达检测必须同时使用普通的Divergent Primers和Sjod Primers两对引物。普通的Divergent Primers特异性较差,但可以扩增到环化错误的条带(若有),适合扩增条带来做sanger测序。而Sjod Primers特异性高,适合做定量检测。另外扩增产物必须进行sanger测序,以确认过表达后产物是准确环化的,当然sanger测序得到环化位点处序列也是目前验证circRNA真实存在的金标准,这个测序结果自然是重中之重了。
好了,昨天湖人为大家支了五招来构建circRNA过表达载体,今天再介绍了相对严谨地验证是否过表达成功,相信对大家有所启发,也祝所有同学以后做circRNA过表达都可以成功,我们江湖再见!
.png)