RNase R ( Ribonuclease R ) 是一种来源于大肠杆菌RNR超家族的3’-5’核糖核酸外切酶,可从3’-5’方向将RNA逐步切割成二核苷酸和三核苷酸。RNase R能够消化几乎所有的线性RNA分子,但不易消化环形RNA,因此常用于circRNA的富集和鉴定实验。湖人今天将介绍RNase R在circRNA研究中的具体应用和常见问题。
1 RNase R的应用
1.1 circRNA高通量测序
高通量测序是大批量发现circRNA最快捷的方法,目前对于circRNA的高通量测序主要有两种策略[1]。第一种将total RNA去除rRNA(核糖体RNA)后直接进行建库测序,不做RNase R消化(RNase R-),也叫全转录组测序。第二种将total RNA去除rRNA后进行RNase R消化(RNase R+),以使circRNA相对富集,再进行建库测序,这种策略结果中circRNA的junction reads相对于RNase R-样本有5-10倍的富集,一般能发现几千到上万个circRNAs,差异表达的有几百到几千个。
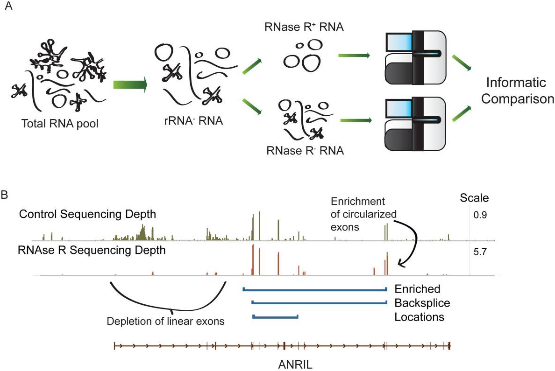
图1 RNase R+和RNase R- RNA测序示意图(Jeck WR, Sharpless NE, 2014)
1.2 Northern blot
Northern blot是一项特异性检测RNA的杂交技术,使用探针可对内源性的circRNA和/或mRNA同时/分别做定量和定性检测。
对于线性mRNA的探针,按照一般设计方法设计即可。而呈环形的circRNA需要针对其Splice Junction位置设计探针。
若同时使用mRNA探针和circRNA的Splice Junction探针进行检测,结果中RNase R-组中应该可以同时检测到mRNA和circRNA的条带。而在RNase R+组中只能检测到circRNA条带,检测不到mRNA的条带(或条带变淡)[2]。
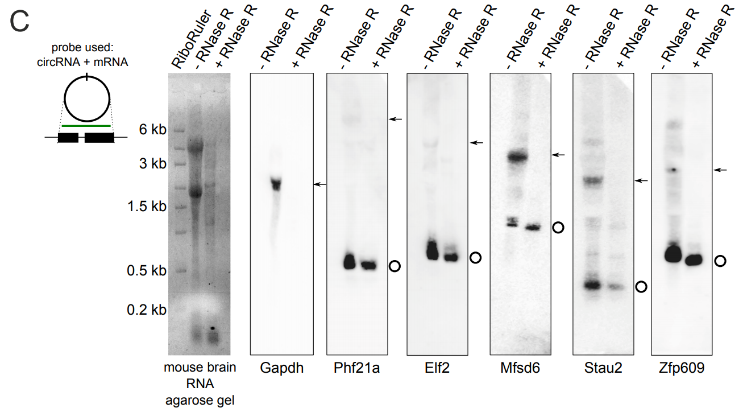
图2 Northern blot同时使用circRNA和mRNA探针(Rybak-Wolf, A. et al., 2015)
注:实验中也可对mRNA和circRNA的同源序列设计探针,用来同时检测mRNA和circRNA。
1.3 PCR检测
Total RNA经RNase R消化后可于70℃ 10 min使酶灭活后直接进行逆转录反应,确保在RNase R-和RNase R+组中使用等量的total RNA,统一以RNase R-组的内参(ACTB或GAPDH)作为计算标准。
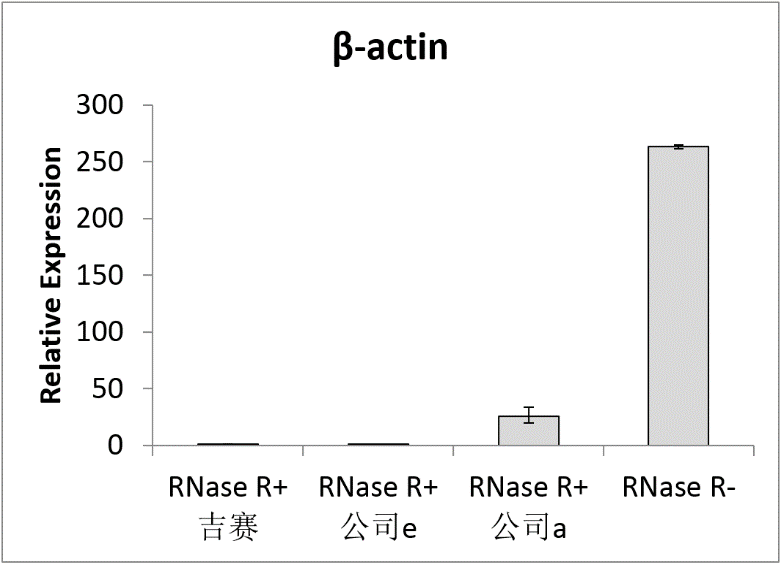
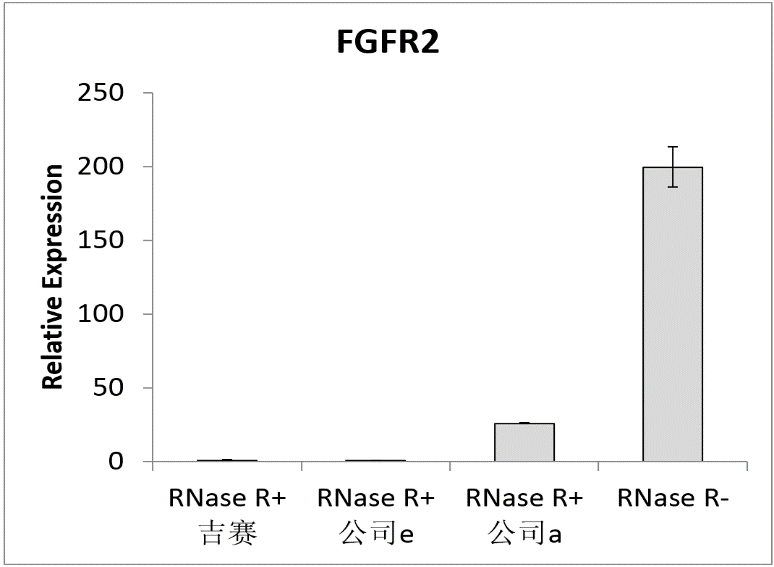
图3 RNase R消化后以qPCR检测β-actin和FGFR2
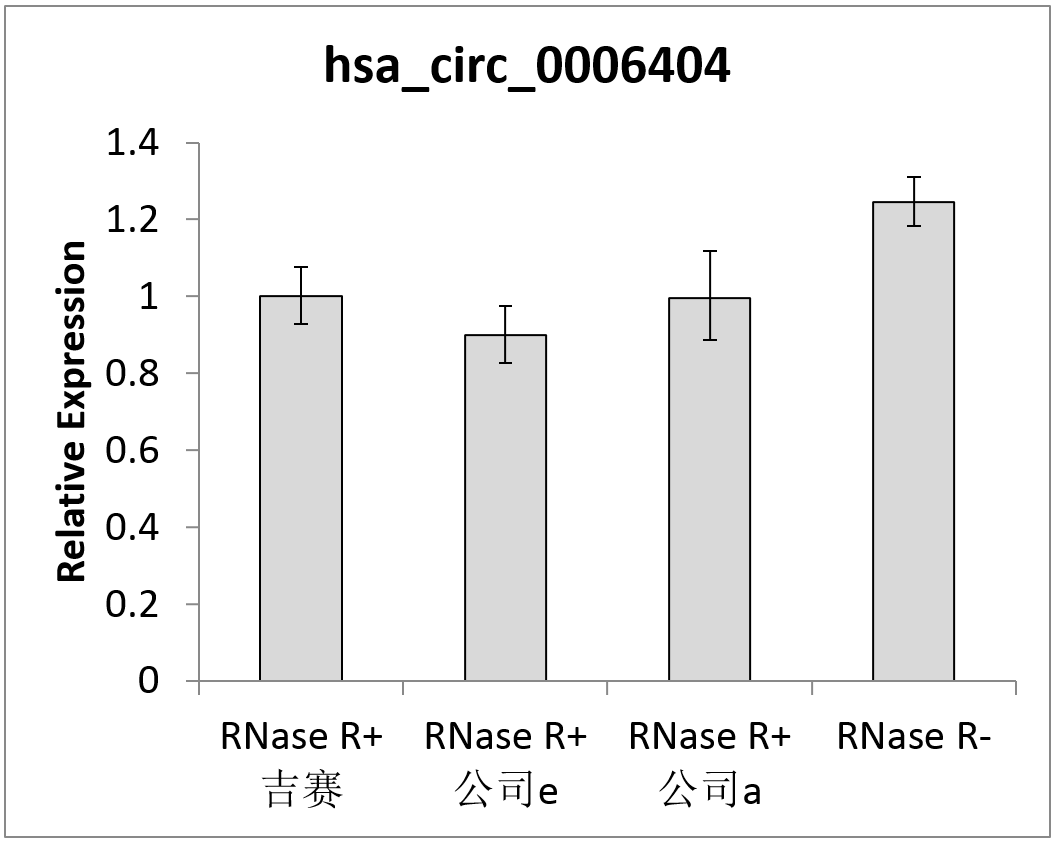
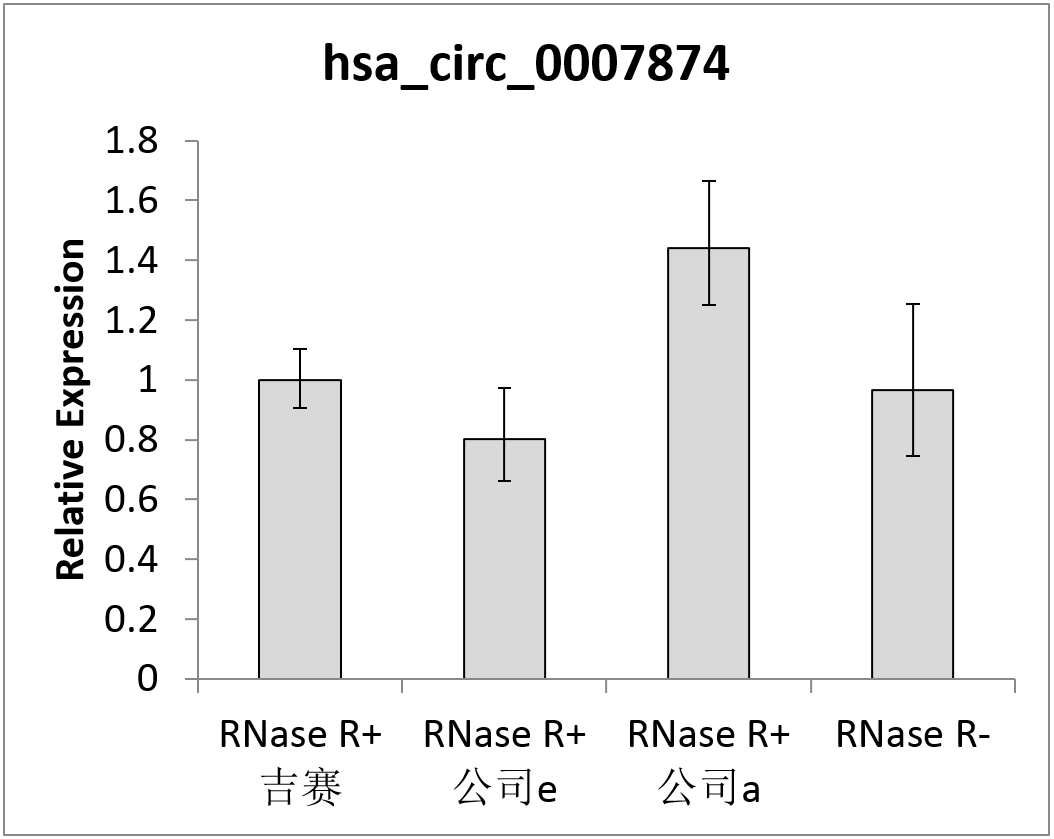
图4 RNase R消化后以qPCR检测hsa_circ_0006404和has_circ_0007874
文章里常见的一个结果如图5,分别以Divergent Primer和Convergent Primer检测cDNA和gDNA样品。图中gDNA中Divergent Primer无条带,Convergent Primer有条带;RNase R+组中Divergent Primer有条带,Convergent Primer无条带;RNase R-组中Divergent Primer和Convergent Primer都有条带,表明检测的基因呈环形,且耐受RNase R消化[3]。
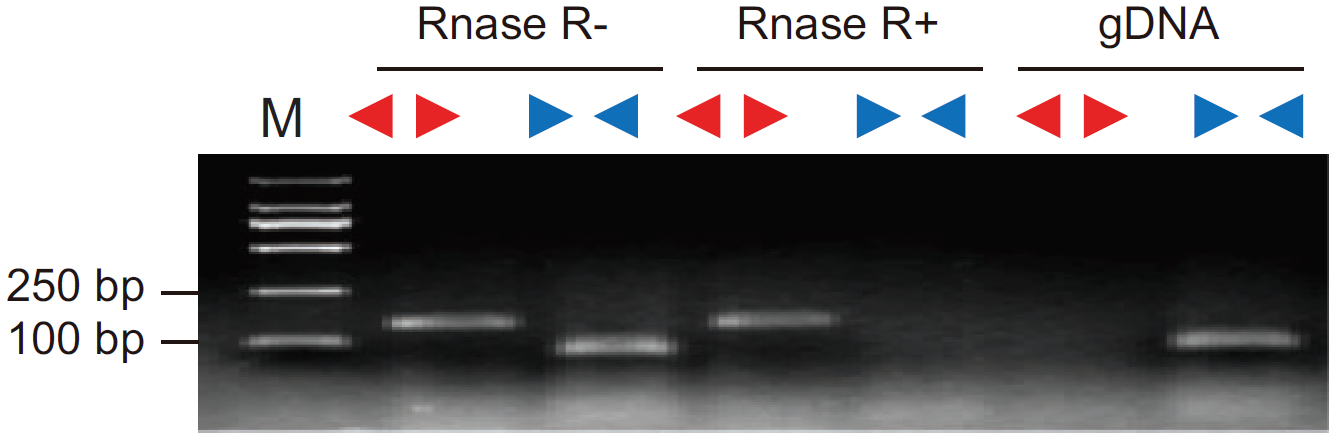
图5 RNase R消化后分别以Divergent Primer和Convergent Primer检测cDNA和gDNA样品(Yibing Y et al., 2018)
- RNase R消化实验步骤
2.1 反应体系
推荐按表1配置反应体系。
表1 RNase R消化反应体系
| RNA | < 5 μg | > 5 μg |
| 10X Reaction Buffer | 2 μL | 5 μL |
| RNase R(20U/μL) | 3-4 U/μg RNA | 3-4 U/μg RNA |
| RNase-Free Water | Up to 20 μL | Up to 50 μL |
注:3-4 U/μg RNA为参考文章适用于人或小鼠total RNA的比例,其他物种或条件可能需要适当调整。
-
- 反应条件
37℃10-30 min。
注:1)可随RNA增加适当延长消化时间,一般10-30 min即可消化掉大部分的线性RNA,PCR检测线性RNA丰度有几百倍的降低。消化1 h以上是非必要的,因为时间过长可能导致少数耐受力弱的circRNA被消化。2)孵育后可先纯化回收,或者70℃ 10 min使酶灭活后直接进行下游实验。
-
- 纯化回收
消化后的RNA可使用苯酚:氯仿:异戊醇(25: 24:1, V: V)溶液抽提,再使用乙醇沉淀回收;或者使用RNA纯化柱和磁珠进行纯化回收。
注:苯酚:氯仿:异戊醇(25: 24:1, V: V)溶液最好现配现用,没有试剂也可以Trizol Reagent代替。
3 常见问题
3.1 内参的选择
应用于RNase R消化前后或处理与不处理组的对比,PCR检测的常用内参ACTB或GAPDH都因被消化而不能使用。此时可将原始的RNA样品等分为两份,一份经RNase R消化(RNase R+),另一份不处理(RNase R-),以RNase R-组的内参作为计算标准。
3.2 定量计算
将原始RNA样品等分为两份,一份经RNase R消化,另一份不处理,此时认为两组中RNA等量,内参一致,以不处理组的内参(ACTB或GAPDH)作为计算标准。
考虑RNase R消化后如果进行纯化回收,则RNase R+组中RNA浓度/总量可能有变化,不适宜再以RNase R-组的内参(ACTB或GAPDH)作为计算标准。此时可在纯化回收前加入少量其他物种的RNA作为外参,统一以外参标准化样品后进行计算[4]。
Total RNA经RNase R消化后直接进行RT-PCR的,一般可以不做纯化回收,保持70℃ 10 min使酶灭活后直接进行RT即可。
3.3 Total RNA消化后应该是怎样的?
对于真核生物的total RNA,RNase R-组中28S/18S/5S三条带单一明亮(5S较淡),而RNase R+组中28S/18S/5S条带变淡或不可见。但如果RNase R+组中5S条带加亮,且28S/18S条带处有拖尾,则可能是RNA被外源RNA酶降解,而不是RNase R消化产生的。
如图6中使用10 U RNase R消化2.5 μg total RNA,保持37℃ 30 min,之后直接进行电泳检测,结果显示RNase R+组28/18/5S条带变淡(不可见),表明RNase R对total RNA的消化作用。

图6 Total RNA 经RNase R消化后直接进行电泳检测
3.4 实验结果一致性(重复性)差
常见的RNase R消化RNA后进行qPCR检测等实验,容易碰到的问题是多次重复实验的结果差别很大,可能的原因和解决方法如下。
1)加样不准,RNA或RNase R的用量有误差,导致内参不齐或者消化程度不一致。在同一对比实验中,除了是否加RNase R外,应确保其他所有反应条件一致。
2)两次重复实验结果不一致。尽量同时进行RNase R+和RNase R-组的消化实验,使用相同的反应条件,PCR检测使用同样的条件和引物等。
3)检测mRNA和circRNA丰度都没有明显变化。可能由于RNase R消化反应体系、程序不对,或者RNA使用量过多。实验中应使用配套的10X Reaction Buffer配置反应液,使用推荐的反应温度和时间,另外添加MgCl2(Mg2+浓度最高0.5 mM)可以增强RNase R的活性。
4)RNase R+组检测不到circRNA。可能由于circRNA丰度过低未能检测到,或者RNA样品被外源RNA酶降解。实验中应使用RNase-Free(DEPC处理)的枪头、离心管和水等耗材试剂。
5)RNase R+组中circRNA丰度有明显降低。除了考虑RNA样品被外源RNA酶降解外,也有发现少数circRNA耐受RNase R消化力弱的,提示检测的circRNA可能也被消化。
参考文献:
1. Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs. Nat Biotechnol. 2014; 32:453–61.
2. Rybak-Wolf, A. et al.. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed. Mol. Cell. 58, 870–885 (2015).
3. Yibing Yang et al.. Novel Role of FBXW7 Circular RNA in Repressing Glioma Tumorigenesis. J Natl Cancer Inst. 2018 Mar 1;110(3).
4. Pamudurti, Nagarjuna Reddy et al.. Translation of CircRNAs. Molecular Cell , Volume 66 , Issue 1 , 9 – 21.e7.
.png)