9月26日,Theranostics杂志在线发表了加拿大多伦多大学杨柏华教授为通讯作者的综述文章,系统汇总了截至目前circRNA与蛋白相互作用的研究进展[1]。

环形RNA虽然在20多年前就已经被发现了,但真正受到学术界关注还是近几年的事,伴随着二代测序技术的兴起而逐渐受到国内外的热捧。目前circRNA研究更多的是其表达特征与疾病或生理活动的相关性,具体的分子细胞生物学机制报道相对少很多。但随着各种筛选鉴定工作的深入开展,所筛选到的circRNA分子的作用机制必然越来越受到重视。circRNA的功能机制模型,目前报道最多的是作为内源竞争性RNA(ceRNA)竞争性结合miRNA。但也有一些报道发现circRNA分子可直接与蛋白相互作用,在本文中,杨柏华教授专门针对circRNA与蛋白的相互作用进行了系统的汇总和剖析。下面我们就一起拜读一下这篇文章吧:
作者首先大致回顾了circRNA的研究历史和主要研究历程,功能研究的主要进展,包括竞争性结合miRNA等,但总体而言,circRNA功能研究还非常有限,还有很多未解之谜。除了竞争性结合miRNA,circRNA功能模型方面还有一些有趣的报道,包括携带内含子的circRNA(EIciRNAs)促进母基因的转录,circ-RasGEF1B促进ICAM-1基因的mRNA稳定性,以及circRNA直接翻译蛋白或多肽等。
目前已报道的circRNA-蛋白相互作用及其机制
目前已明确的circRNA与蛋白相互作用的报道数目还不是很多,作者在文章中进行了详细的汇总分析,汇总成表格如下:
表1 已报道的circRNA-蛋白相互作用
| 文章题目(杂志,发表时间) | 通讯作者 | 主要机制 |
| Natural RNA circles function as efficient microRNA sponges (Nature, 2013) | 丹麦奥胡思大学Jørgen Kjems教授 | ciR-7/CDR1as通过AGO2蛋白实现竞争性结合miR-7 [2]。后面很多的ceRNA模型中均证明了circRNA可以与AGO2蛋白相互作用,此处不再一一列举。 |
| circRNA Biogenesis Competes with Pre-mRNA Splicing (Molecular Cell, 2014年) | 德国麦克斯·德尔布吕克分子医学研究所 Nikolaus Rajewsky教授和 以色列希伯来大学Sebastian Kadener教授 | MBL蛋白通过结合circMBL促进后者的形成[3]。 |
| Identification of HuR target circular RNAs uncovers suppression of PABPN1 translation by CircPABPN1 (RNA Biology,2017年) | NIH衰老研究所Kotb Abdelmohsen教授 | circ-PABPN1竞争性结合HuR,抑制后者与PABPN1 的mRNA结合,降低PABPN1翻译效率[4]。 |
| Foxo3 circular RNA promotes cardiac senescence by modulating multiple factors associated with stress and senescence responses(European heart journal,2017年) | 加拿大多伦多大学杨柏华教授 | circ-FOXO3与ID-1、E2F1、FAK和HIF1α相互作用,促进这些因子滞留在细胞质中,参与衰老作用过程[5]。 |
| Circular non-coding RNA ANRIL modulates ribosomal RNA maturation and atherosclerosis in humans (Nature Communications, 2016年) | 德国慕尼黑路德维希-马克西米利安大学Lesca M. Holdt和Daniel Teupser | circANRIL与PES1的C端富含赖氨酸的结构域结合,竞争性抑制PES1与核糖体结合,调控核糖体生成过程[6]。 |
| Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2 (Nucleic Acids Research, 2016年) | 加拿大多伦多大学杨柏华教授 | circ-FOXO3与p21和CDK2相互作用,促进p21对CDK2的抑制作用,参与细胞周期的调控[7]。 |
| Induction of tumor apoptosis through a circular RNA enhancing Foxo3 activity (Cell Death & Differentiation, 2017年) | 加拿大多伦多大学杨柏华教授 | circ-FOXO3与MDM2和p53相互作用,促进MDM2介导的p53蛋白的泛素化降解作用。FOXO3蛋白可微弱的结合circ-FOXO3,抑制FOXO3与MDM2的结合,促进FOXO3蛋白的富集[8]。 |
| A circular RNA promotes tumorigenesis by inducing c-myc nuclear translocation(Cell Death & Differentiation, 2017年) | 加拿大多伦多大学杨柏华教授 | circ-Amotl1通过诱导c-Myc入核而促进肿瘤发生。circ-Amotl1还可以与-Myb、NF1、Akt等多种蛋白相互作用[9]。(PS:该文献未在综述文章中列出) |
| The circular RNA circ-Amotl1 interacts with STAT3 increasing its nuclear translocation and wound repair by modulating Dnmt3a and miR-17 function (Molecular Therapy,2017年) | 加拿大多伦多大学杨柏华教授 | circ-Amotl1与Stat3在Dnmt3a启动子区有共定位,存在相互作用,在创口愈合过程中发挥作用。circ-Amotl1还可以与E2F1、E2F4、EGF、ETS-1、AP1、NF1相互作用[10]。(PS:该文献未在综述文章中列出) |
circRNA-蛋白相互作用研究技术
目前最常用的circRNA-蛋白相互作用研究技术主要是基于免疫沉淀分析的方法,包括RNA Pull-down、RIP等。杨教授在文章中也重点介绍了这两个技术的关键注意事项,还介绍了RNase保护分析(RNase protection assay,RPA)和基于杂交的细胞共定位分析技术。
RNA Pull-down
利用生物素标记的DNA片段作为探针,在Co-IP体系中钓取目标circRNA分子,如果有相互作用蛋白,则可以一起被链霉亲和素标记的磁珠一起捕获下来,后续可经过Western Blot或者质谱鉴定分析相互作用蛋白。所用的探针必须是跨过环化位点的,建议长度为30-40nt。为减少线性RNA的干扰,作者建议增加RNase R消化处理。由于circRNA复杂而特殊的结构,实验必须设计严谨的对照,建议同步进行过表达和敲低实验,以有效排除非特异性的信号。
RIP
利用蛋白抗体进行Co-IP实验,分离被捕获的RNA分子进行后续鉴定分析。收取的细胞样品在预冷PBS中洗涤,用Co-IP缓冲液进行裂解,加一抗孵育,用标记有Protein A的琼脂糖凝胶珠进行IP实验。所得到的产物分离RNA后测序或PCR分析。
RNase protection assay,RPA
RNase保护分析(RNase protection assay,RPA)是一种全新的研究RNA-蛋白相互作用的技术。如果一段DNA和RNA能够互补配对,RNase H可以将RNA分子上没有互补配对的部分水解掉,利用该活性,设计多条DNA探针靶向circRNA序列,如果circRNA与蛋白有相互作用,探针则无法准确结合,RNA分子就不会被RNase H降解掉。该方法可获得circRNA与蛋白相互作用的准确序列信息,是非常有效的研究RNA-蛋白相互作用的工具。
荧光原位杂交(FISH)
原位杂交是研究核酸亚细胞定位的重要技术,基于荧光原位杂交(FISH)和蛋白免疫染色(IHC)的共定位分析,可以反映出circRNA与蛋白相互作用的情况。
circRNA-蛋白相互作用的生物学意义
circRNA-蛋白相互作用是揭开circRNA功能神秘面纱的重要技术,从已报道的相关研究可以看出这一相互作用过程也调控了彼此的功能。一方面circRNA与蛋白的相互作用呈现出动态的特征,能够实现对蛋白功能的调控,最典型的如circ-FOXO3,在不同的生理条件下能够动态的与不同的蛋白相互作用,并调控了目标蛋白的功能(如下图所示)。另一方面,蛋白与circRNA的相互作用也动态的调控了circRNA的形成作用等过程,典型的如QKI或MBL与circRNA的相互作用影响了circRNA的形成过程。
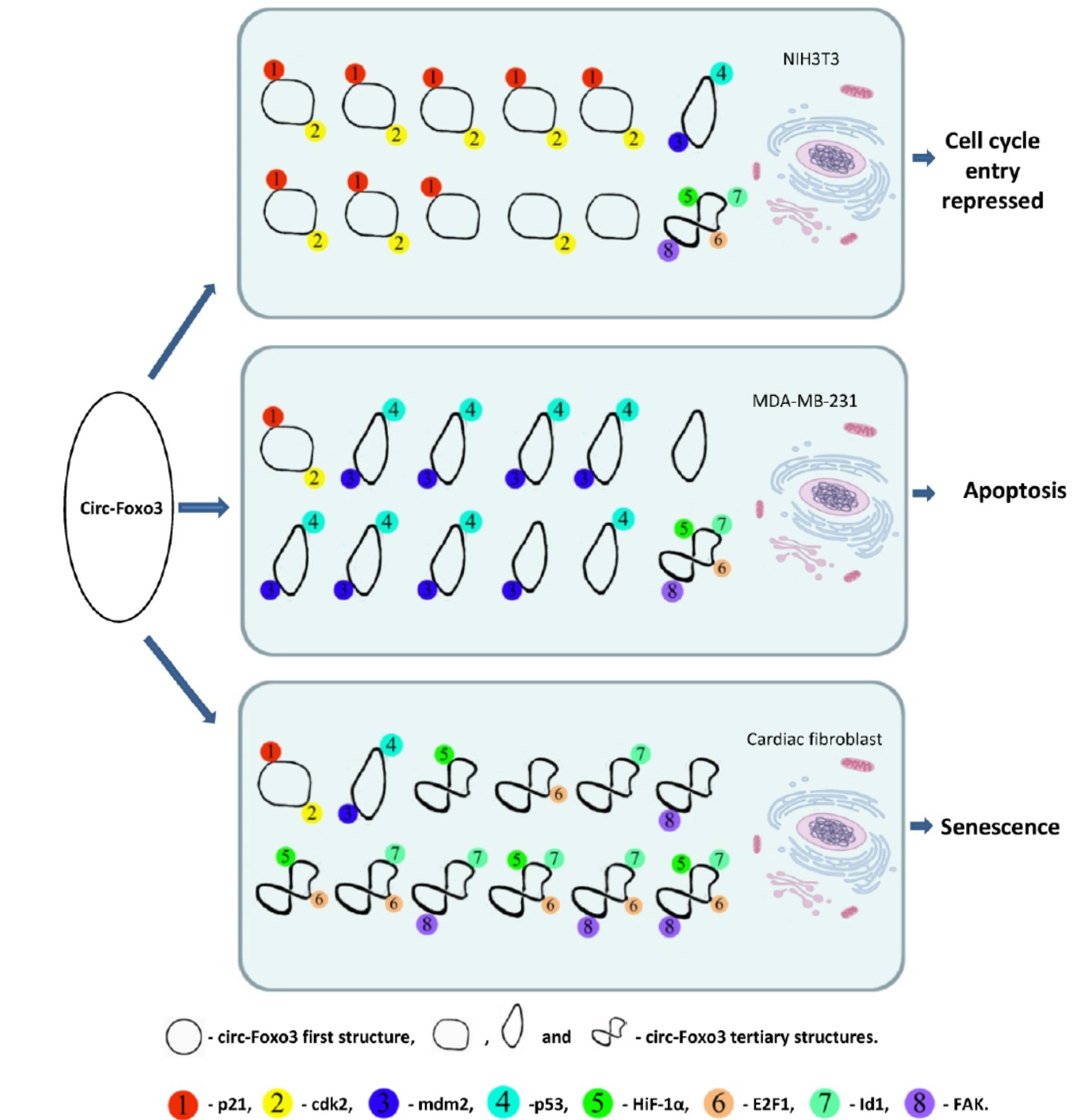
图1 circ-FOXO3在不同生理条件下与不同的蛋白有动态相互作用过程(来自[1])
参考文献:
1. William W Du, C.Z., Weining Yang, Tianqiao Yong, Faryal Mehwish Awan, Burton B Yang, Identifying and Characterizing circRNA-Protein Interaction. Theranostics, 2017.
2. Hansen, T.B., et al., Natural RNA circles function as efficient microRNA sponges. Nature, 2013. 495(7441): p. 384-388.
3. Ashwal-Fluss, R., et al., circRNA biogenesis competes with pre-mRNA splicing. Mol Cell, 2014. 56(1): p. 55-66.
4. Abdelmohsen, K., et al., Identification of HuR target circular RNAs uncovers suppression of PABPN1 translation by CircPABPN1. RNA Biol, 2017. 14(3): p. 361-369.
5. Du, W.W., et al., Foxo3 circular RNA promotes cardiac senescence by modulating multiple factors associated with stress and senescence responses. Eur Heart J, 2016.
6. Holdt, L.M., et al., Circular non-coding RNA ANRIL modulates ribosomal RNA maturation and atherosclerosis in humans. Nat Commun, 2016. 7: p. 12429.
7. Du, W.W., et al., Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2. Nucleic Acids Res, 2016. 44(6): p. 2846-58.
8. Du, W.W., et al., Induction of tumor apoptosis through a circular RNA enhancing Foxo3 activity. Cell Death Differ, 2016.
9. Yang, Q., et al., A circular RNA promotes tumorigenesis by inducing c-myc nuclear translocation. Cell Death Differ, 2017.
10. Zhen-Guo Yang, F.M.A., William W Du, Yan Zeng, Juanjuan Lyu, De Wu, Shaan Gupta, Weining Yang and Burton B Yang, The circular RNA circ-Amotl1 interacts with STAT3 increasing its nuclear translocation and wound repair by modulating Dnmt3a and miR-17 function. Molecular Therapy, 2017.
.png)