4月2日,Molecular Cell杂志在线发表了一篇有关RNA降解的文章,介绍发现一种m6A介导的RNA降解机制。m6A识别蛋白YTHDF2结合靶分子,并募集HRSP12,进而介导RNA内切核酸酶RNase P/MRP复合物切割靶分子。这一过程可作用于mRNA和circRNA。文章的通讯作者是韩国高丽大学的Yoon Ki Kim[1]。
circRNA的降解是认识其功能和表达方式的重要一环,但相关机制的认识还非常有限。本文的模型表明基于m6A识别蛋白YTHDF2的降解作用不仅对线性的mRNA有效,也能对circRNA有效,大大增进了对circRNA降解机制的认知。文章从YTHDF2,HRSP12和RNase P/MRP复合物的相互作用鉴定开始,分别对各个成分进行RNAi干扰,分析它们相互作用及对靶分子降解速率的影响,YTHDF2截短突变表明其N端的100aa是介导其与HRSP12和RNase P/MRP复合物结合的关键区域,最后基于CLIP等技术分析了YTHDF2和HRSP12结合靶分子的特异性,并证明circRNA也是该体系的重要靶分子[1]。下面就让我们一起学习一下本文的内容:
YTHDF2,HRSP12和RNase P/MRP复合物相互作用鉴定
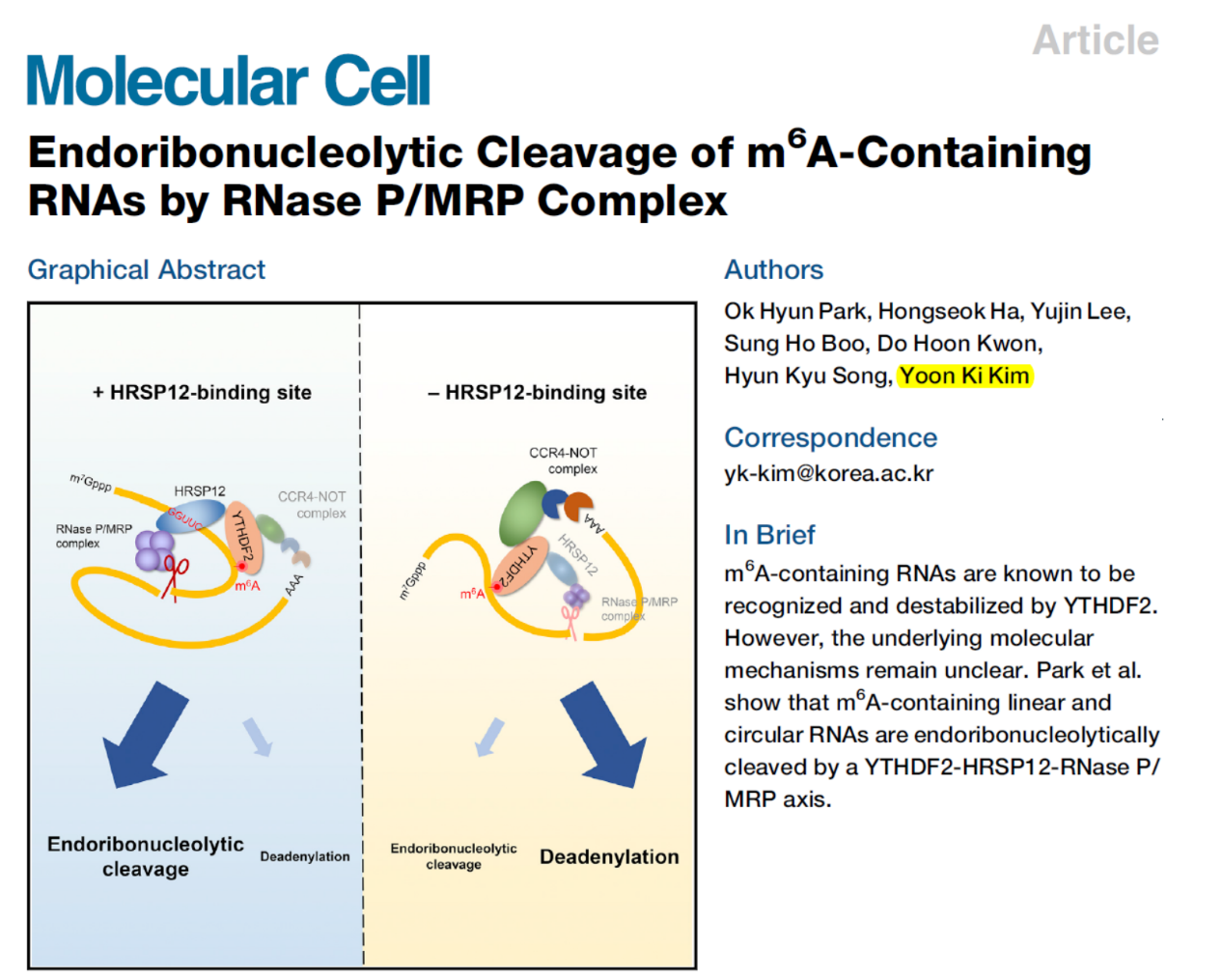
已有文献报道酵母的YTHDF2同源蛋白Pho92与Mmf1(HRSP12的同源蛋白)存在相互作用,在另一项研究中预测到HRSP12可以与RNase P/MRP复合物的亚基POP1存在相互作用的可能。CoIP实验证明了人类细胞293T中YTHDF2,HRSP12和POP1确实存在相互作用,并且RNase A消化并不能抵消它们之间的相互作用,因此三者的相互作用是蛋白之间的作用,并不是由RNA介导的。进一步,体外GST融合标签Pull-down实验,Far-Western实验进一步验证了三者的相互作用。HRSP12的siRNA实验进一步表明,HRSP12是介导三者相互作用的中间桥接分子。此外,作者也在CoIP实验中验证了此前报道的YTHDF2相互作用蛋白CNOT1,且YTHDF2与CNOT1相互作用不依赖于HRSP12。
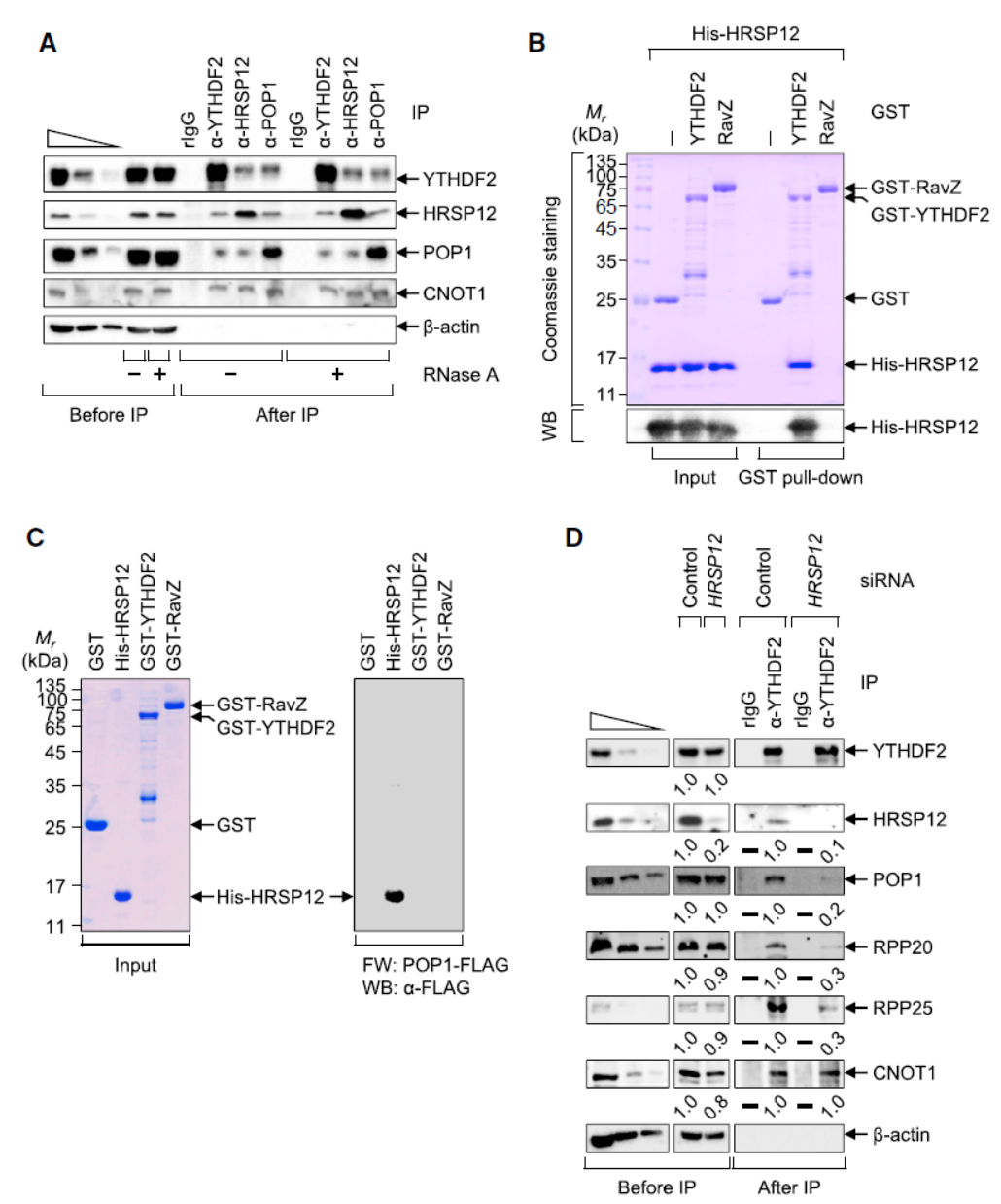
图1 YTHDF2,HRSP12和POP1相互作用([1])
YTHDF2,HRSP12和POP1参与m6A介导的RNA降解
由于上述的相互作用复合体中包含了内切核酸酶组分,这一相互作用是否介导了RNA的降解作用?基于SON和PLAC2基因所构建的mini基因报告系统可用于研究m6A修饰相关的过程,将其中的m6A修饰位点突变后可分析突变对于对应转录产物的影响。利用该模型,作者分析了分别干扰YTHDF2,HRSP12和POP1对报告基因转录产物的表达丰度影响情况,结果表明YTHDF2,HRSP12和POP1分别干扰后均能增加对应转录产物的丰度,延长这些分子的半衰期。有趣的是不论野生型还是突变型的报告基因,干扰均可增加其丰度。糖皮质激素受体通路可介导mRNA的降解(Glucocorticoid Receptor (GR)-Mediated mRNA Decay,GMD),HRSP12也是该RNA降解作用途径中的一个分子,那么本文所发现的这个机制与GMD通路是否一致?还是全新的RNA降解途径?地塞米松是已知的糖皮质激素衍生物,可直接诱导GMD作用,但地塞米松处理并不能增加m6A介导的报告分子降解作用。干扰POP1也不能下调GMD作用。HRSP12的敲除细胞系中回补表达野生型FLAG-HRSP12及P105A/R107E两种突变型基因的结果表明,两种突变型并不能恢复GMD作用,但可以恢复m6A介导的报告分子降解。因此m6A介导的RNA分子降解机制与GMD是不同的通路。
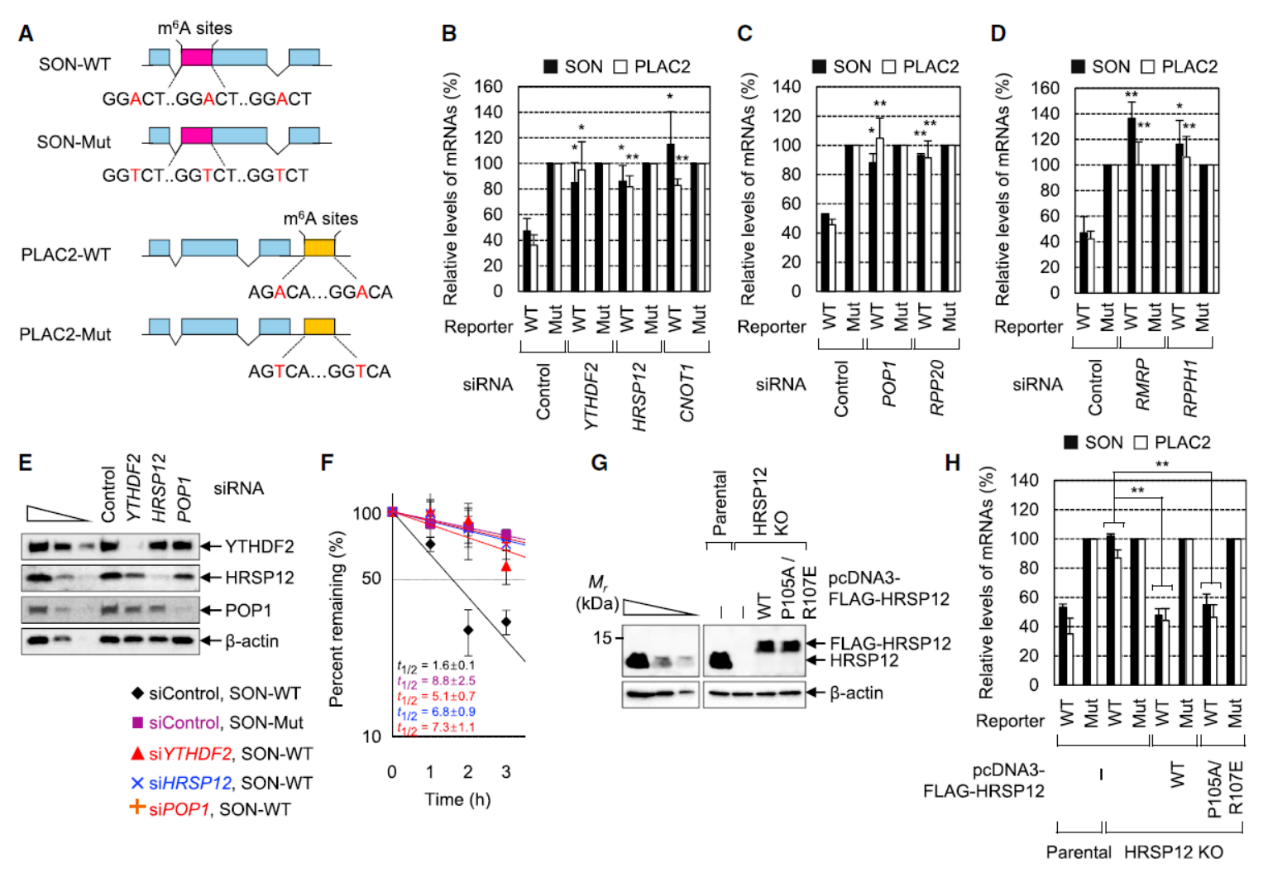
图2 YTHDF2,HRSP12和POP1参与m6A介导的RNA降解作用([1])
YTHDF2的N端100aa介导与HRSP12的相互作用
为弄清楚YTHDF2与HRSP12相互作用的位点,作者构建了截短突变体及基于MS2的β-6bs报告载体,将MS2序列融合于β-6bs报告分子的3’-UTR,MS2识别蛋白分别于不同的YTHDF2截短突变体或SMG5融合,这样可以借助MS2及其识别蛋白强制将靶分子募集于β-6bs报告分子的3’-UTR,通过分析β-6bs报告分子的丰度可监测相关分子是否具有促进靶分子降解的作用。在HAP1细胞中过表达SMG5或YTHDF2均能显著降低报告分子的丰度,但在HRSP12-KO的HAP1细胞中,过表达YTHDF2不能加速报告分子的降解,这表明在该模型中能够有效的分析m6A介导的基于YTHDF2和HRSP12的RNA降解作用。
CoIP和Pull-down实验表明YTHDF2的N端100aa是介导其与HRSP12相互作用的关键区域。在HRSP12-KO的HAP1细胞中分析其功能,结果表明只要YTHDF2的N端100aa缺失的载体均没法恢复RNA降解的作用,因此YTHDF2的N端100aa是介导其与YTHDF2相互作用及m6A介导的RNA降解作用的关键区域。
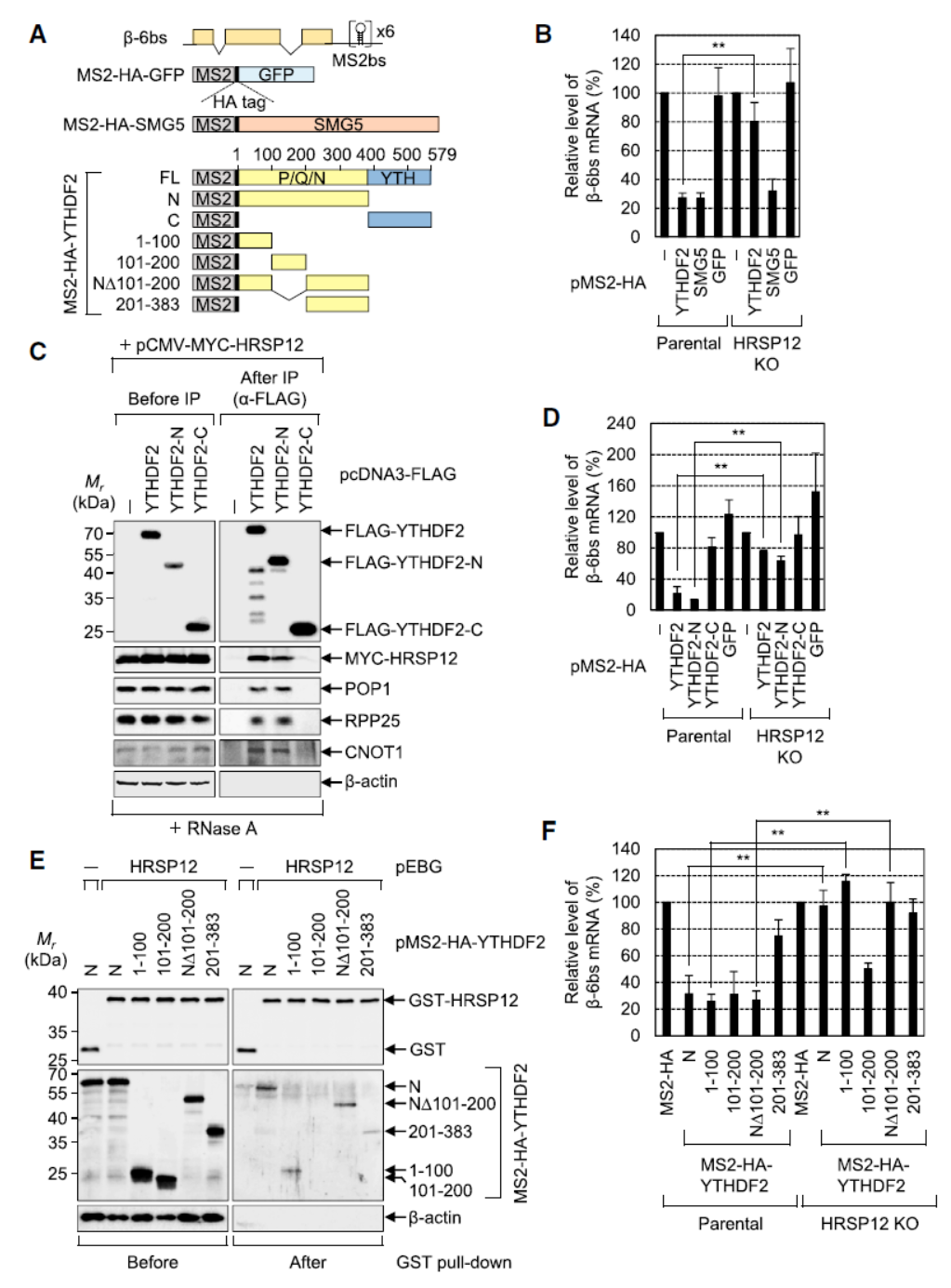
图3 YTHDF2的N端100aa是介导与HRSP12相互作用的关键 ([1])
HRSP12和RNase P/MRP与YTHDF2的结合导致其结合的RNA分子不稳定
之前有研究报道基于RIP-seq和PAR-CLIP Seq分析了Hela细胞中被YTHDF2结合的分子。基于这些研究基础,可将全转录组中的基因分为四大类:没有被YTHDF2结合分子(Non-targets,3905个),仅在CLIP实验中捕获的(CLIP-IP组,1974个),CLIP组富集的分子(CLIP组,3250个)和同时在CLIP和RIP实验中捕获的(CLIP∩IP,1276个)。在Hela细胞中分别在分别干扰YTHDF2,HRSP12或RNase P/MRP组分,基于上述分组绘制各组的累积分布函数曲线(cumulative distribution function)。结果表明,HRSP12和RNase P/MRP与YTHDF2的结合导致其结合的RNA分子不稳定,表现为已知的可与HRSP12结合的RNA分子分布丰度总体增加,半衰期延长。
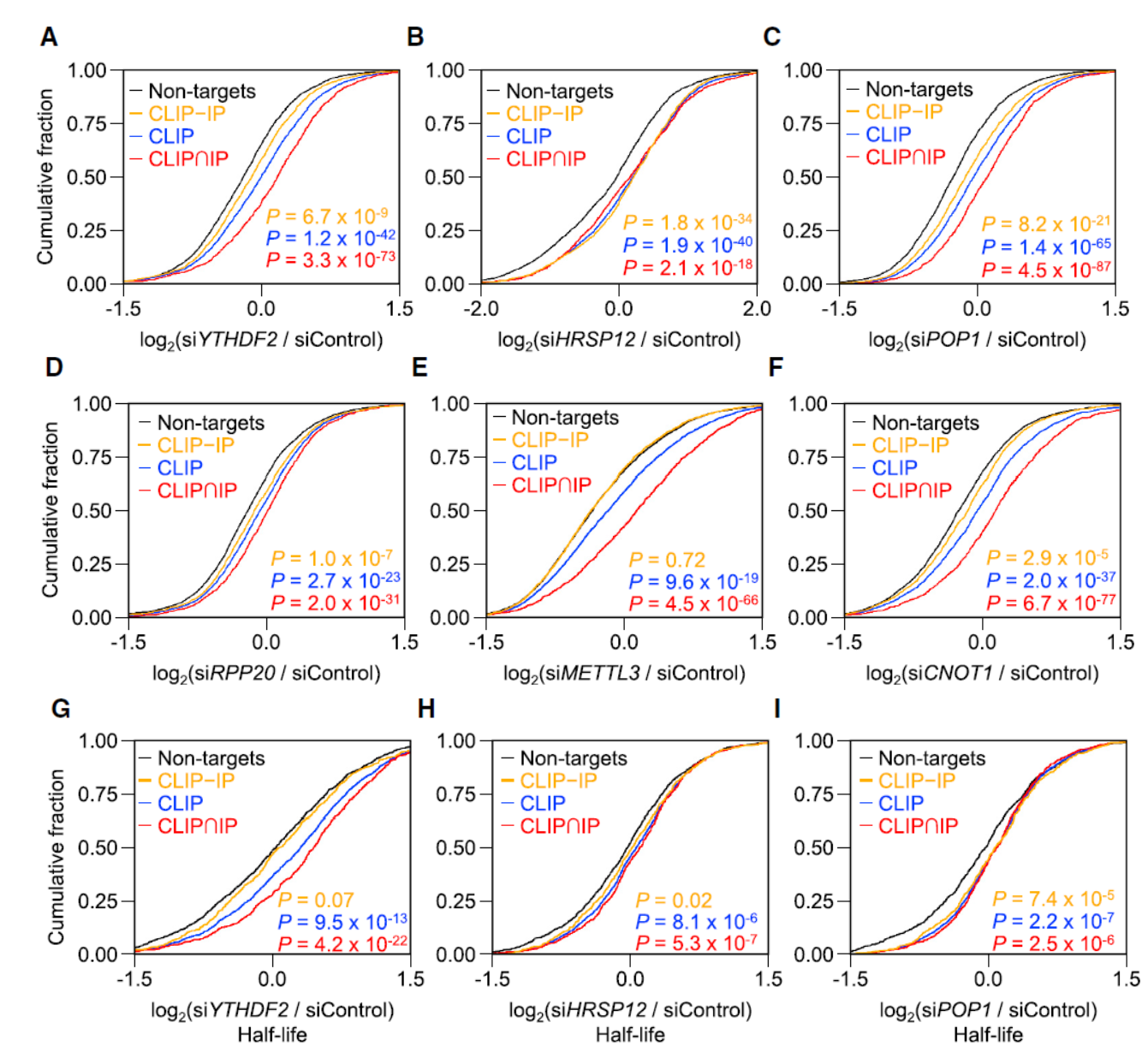
图4 干扰YTHDF2,HRSP12或RNase P/MRP导致YTHDF2结合的RNA分子增多([1])
HRSP12结合位点分析
HRSP12也是一种RNA结合蛋白,该分子结合RNA的motif是怎样的呢?是否与YTHDF2结合的m6A 修饰有重叠?为此,作者利用PAR-CLIP实验分析了HRSP12这些转录组中结合RNA的位点及分布情况。PAR-CLIP测序表明HRSP12结合RNA的区域与YTHDF2分布比较类似。按照HRSP12与YTHDF2分别捕获的分子分类,全转录组中的基因可分为四组:HRSP12与YTHDF2均不能捕获的(Non-targets),HRSP12能捕获但YTHDF2不能捕获的(H-Y),HRSP12不能捕获但YTHDF2能捕获的(Y-H),HRSP12与YTHDF2均能捕获的(H∩Y)。分别干扰YTHDF2,HRSP12或RNase P/MRP组分及CNOT1后分析各组的累积分布函数曲线。结果表明全转录组中,可被HRSP12或YTHDF2结合的RNA分子在分别干扰YTHDF2,HRSP12或RNase P/MRP组分时降解速率减慢。

图5 HRSP12结合分子降解状态分析 ([1])
HRSP12结合位点和RNase P/MRP切割位点分别位于YTHDF2识别位点的上游和下游
PARE-seq(parallel analysis of RNA ends sequencing)可以分析内切核酸酶的切割位点,借助这一技术,作者分析了YTHDF2识别位点,HRSP12结合位点及RNase P/MRP切割位点的分布情况。结果表明HRSP12结合位点和RNase P/MRP切割位点分别位于YTHDF2识别位点的上游和下游。YTHDF2和HRSP12协同促进相互结合RNA分子,并促进基于RNase P/MRP的RNA内切和降解。
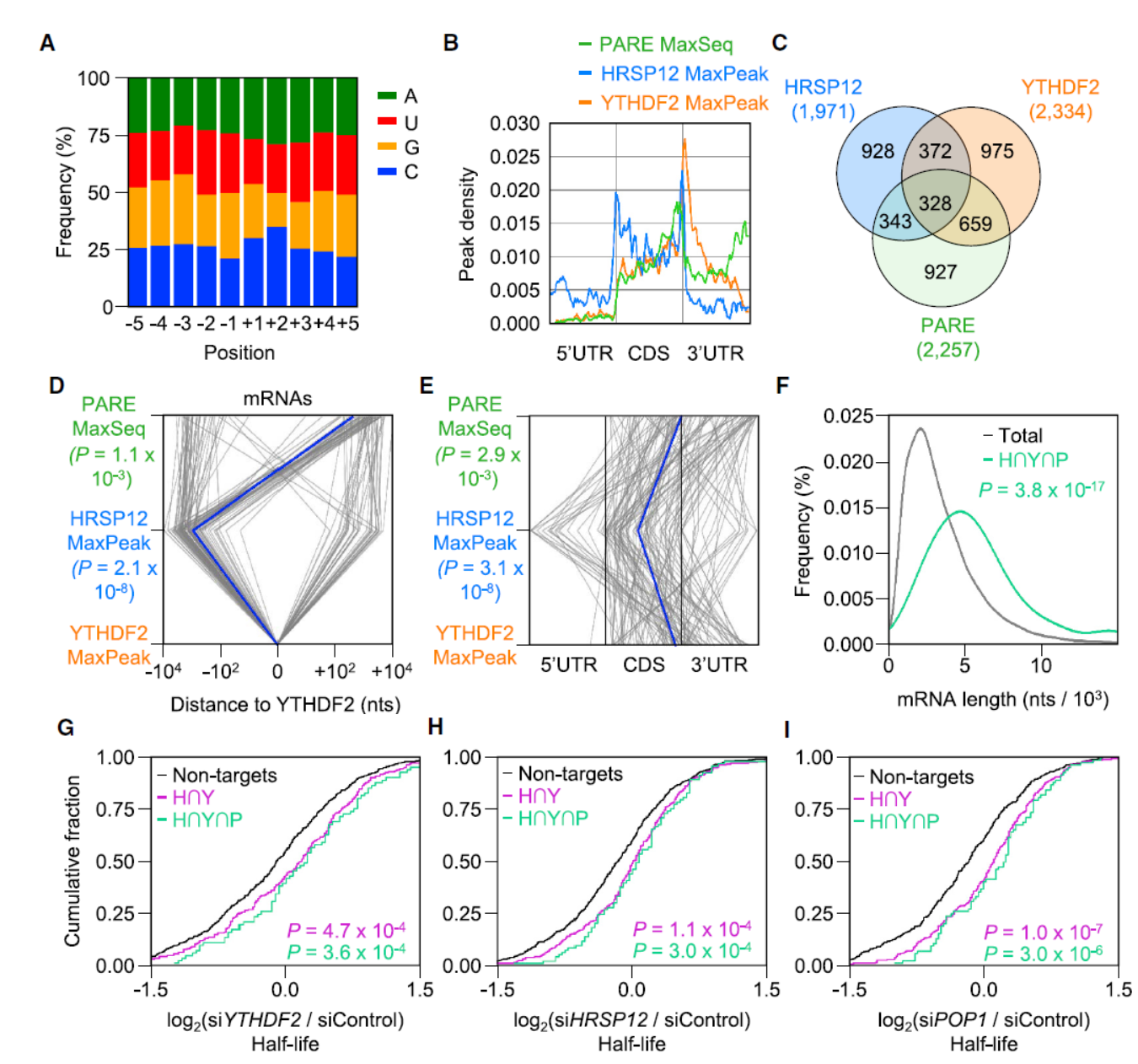
图6 YTHDF2,HRSP12结合位点及RNase P/MRP切割位点分析([1])
circRNA也可以通过YTHDF2- HRSP12介导的RNase P/MRP复合物结合并降解
YTHDF2结合的分子中也包含circRNA分子,作者挑选了其中的三种分子进行了分析:cMATR3,cFAM20B和cSLC45A4。结果表明这些circRNA分子可以通过YTHDF2- HRSP12介导的RNase P/MRP复合物结合并降解。
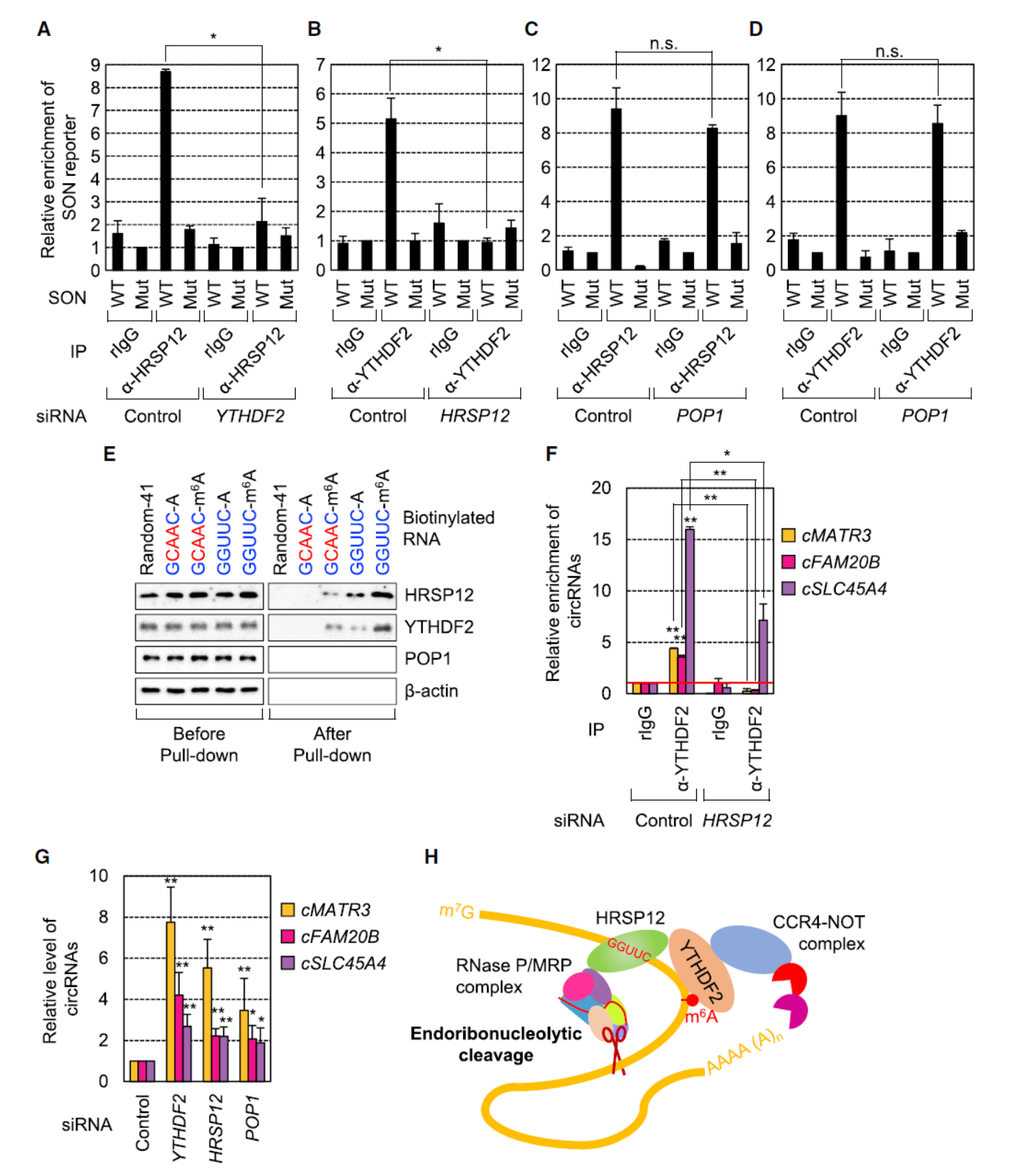
图6 circRNA可通过m6A介导降解作用 ([1])
本文主要探讨YTHDF2- HRSP12介导的RNase P/MRP复合物结合并降解靶分子RNA的机制,是RNA降解作用机制的重要补充。并且验证了该机制在circRNA降解中的作用,对于circRNA表达和降解动态变化相关的机制认识有重要参考意义。
参考文献:
1. Park, O.H., et al., Endoribonucleolytic Cleavage of m(6)A-Containing RNAs by RNase P/MRP Complex. Mol Cell, 2019.
.png)