前不久我们介绍过赵老师团队的circAtlas 3.0整合了纳米孔测序数据,能够提供一些环状RNA的转录本全长信息。
详情见:Nucleic Acids Res | 北京生科院赵方庆团队更新circAtlas 3.0 版本:环状RNA全面信息的综合数据库
很快,在2023年10月12日,Nucleic Acids Research(IF=14.9)再一次刊发了关于纳米孔全长环状RNA的文章“FL-circAS: an integrative resource and analysis for full-length sequences and alternative splicing of circular RNAs with nanopore sequencing”,该研究成果由台湾省中央研究院的庄树谆教授团队分享。
作者认为,短读长测序虽然已经鉴定到了数百万的BSJ(back-spliced junction),然而确定其确切的全长以及可变剪切异构体仍具有很大的挑战性。而纳米孔全长环状RNA测序策略为解决这些问题提供了重要的通道。因此,作者收集了20个细胞系或组织已发表的三代测序数据,从中识别到了人和小鼠大量BSJ以及环状RNA全长异构体(或称为亚型),构建了FL-circAS数据库。
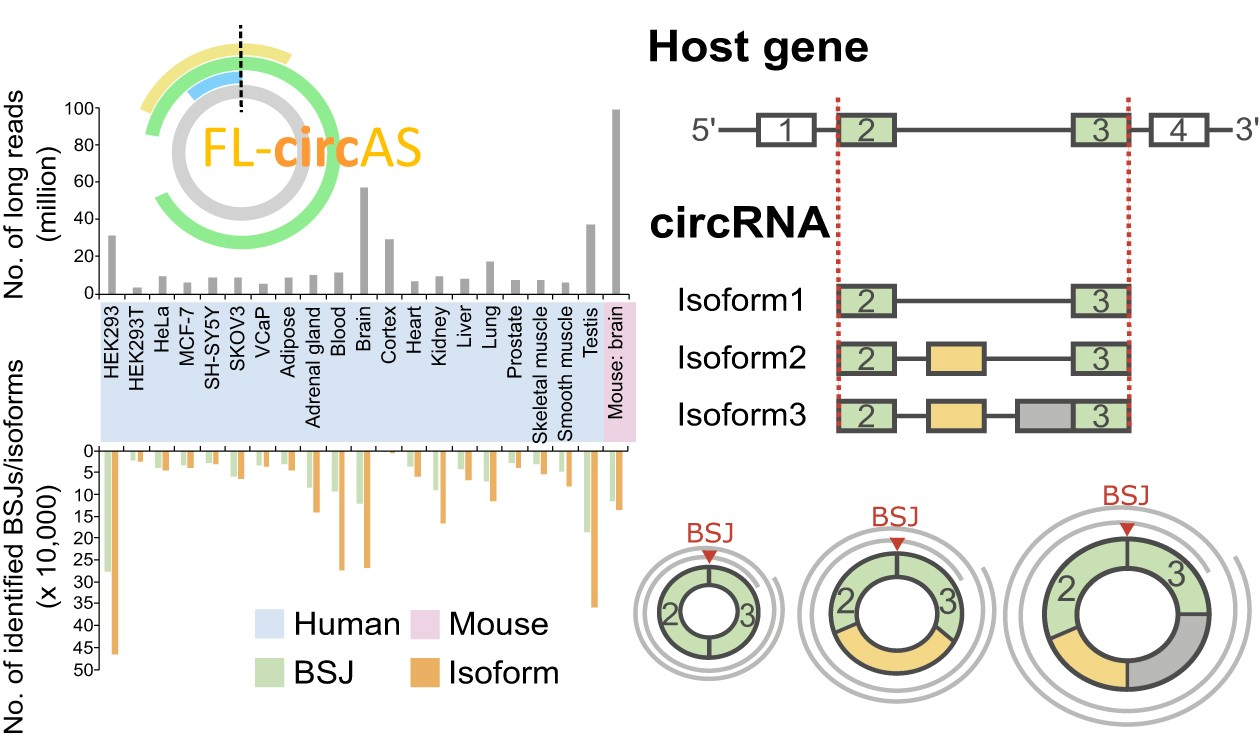 图1. Graphical Abstract
图1. Graphical Abstract
除了提供环状RNA全长和亚型信息,FL-circAS还囊括了环状RNA的多种特征,包括:
环状RNA分子刻画
– 每个BSJ和亚型的表达水平
– 细胞或组织特异性
– 以及每个BSJ发生可变剪切的信息熵
环状RNA的生物学起源
– BSJ侧翼序列的反向互补序列
– BSJ侧翼序列RBP结合位点
环状RNA功能
– miRNA结合位点
– RBP结合位点
– m6A修饰
– 翻译元件与翻译潜能
该数据库可以通过以下网址访问:
https://cosbi.ee.ncku.edu.tw/FL-circAS/
以下为文章的部分结果:
环状RNA全长亚型的识别
FL-circAS从GEO收集了20个细胞系和组织的长读长 RNA-seq(表1),并用isoCirc和CIRI-long来识别环状RNA全长(图 2)
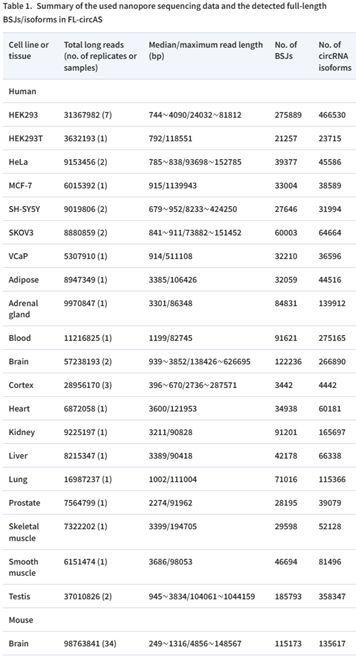 表1. FL-circ中包含的nanopore测序数据
表1. FL-circ中包含的nanopore测序数据
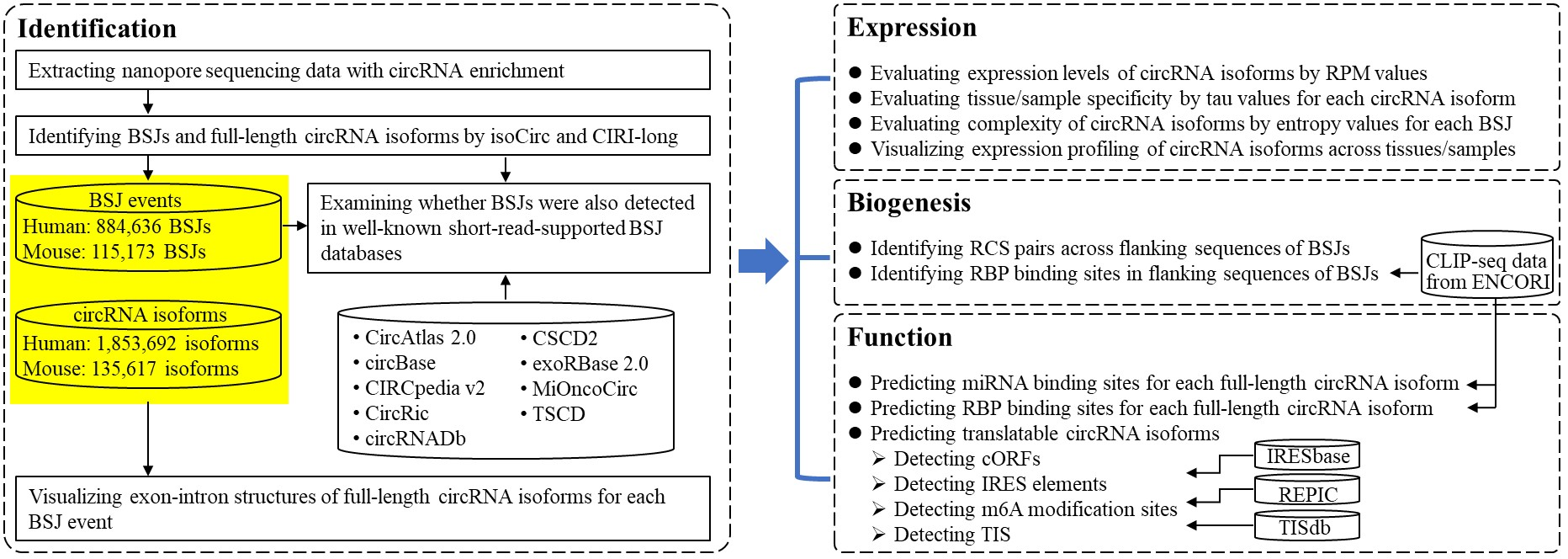 图2. FL-circAS的构建流程图
图2. FL-circAS的构建流程图
案例解析同一BSJ的多个isoforms
第一个案例是TEME138基因来源的环状RNA chr11|61366045|61367998|+,它包含3个具有同样BSJ的isoforms(定义为iso#001、iso#002、iso#003),这些isoforms已在Hela细胞中被RT-PCR和Sanger测序验证。关于该BSJ,FL-circAS总共识别到了214条isoforms,而iso#001、iso#002、iso#003在所有isoforms中具有最多的长读长数据支持。值得注意的是,GENCODE中并没有iso#002、iso#003的外显子注释(图 3A,B);因此,一般短读长测序只能推测出iso#001。从数据库我们可以看到,iso#001主要在HEK293T、brain和testis表达,而iso#002主要在HEK293、adipose、adrenal gland和kidney中表达,另外二者同时在blood中高表达(图 3C)。
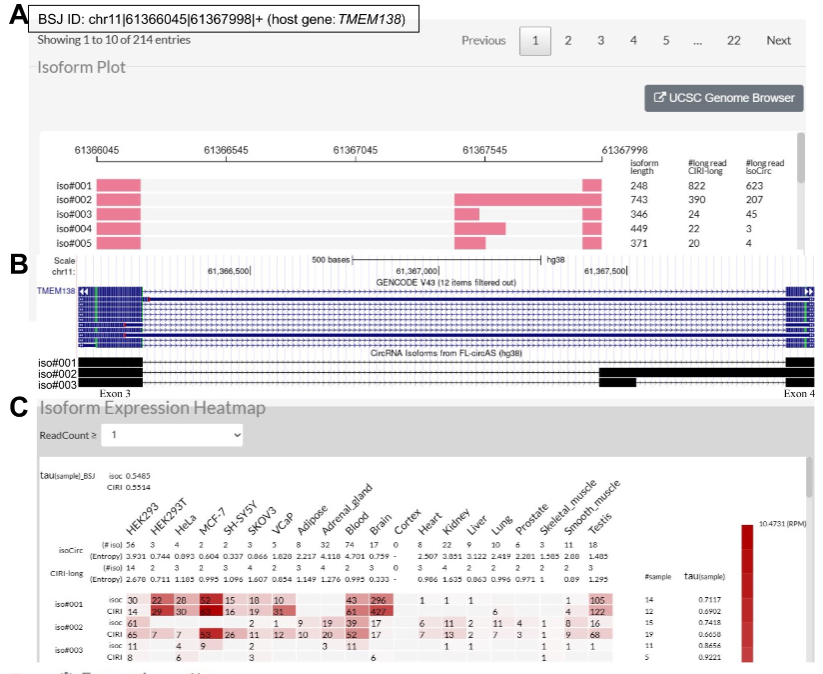 图3. BSJ:chr11|61366045|61367998|+(host gene:TMEM138)案例
图3. BSJ:chr11|61366045|61367998|+(host gene:TMEM138)案例
第二个案例是来源于VAPB基因的chr20|58438945|58441083|+,该BSJ在MCF-7细胞中具有很高的AS信息熵,包含两个高表达的 isoforms(iso#001、iso#002),其中iso#001与 VAPB的外显子一致,而iso#002却存在外显子跳跃(图4E~G)。该BSJ没被验证过,因此,作者对 MCF-7细胞进行RNase R处理,发现该BSJ能够耐受RNase R(图4H);通过PCR-Sanger测序证实了两个isoforms的存在(图4I,J)。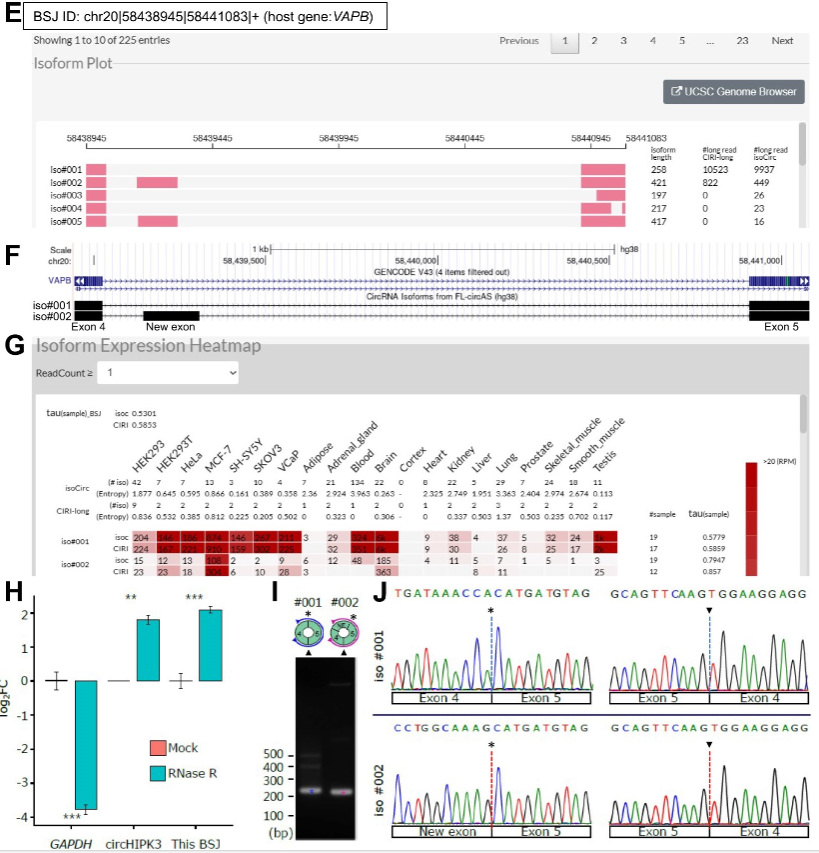
图4. BSJ:chr20|58438945|58441083|+(host gene:VAPB)案例
以上结果再次证明了许多BSJ内部序列是存在AS事件的,而长读长测序是检测环状RNA全长和可变剪切异构体的有效手段。
FL-circAS这个全长环状RNA数据库,不仅多方面刻画了BSJ不同的可变剪切亚型,还提供了circRNA详细的功能和调控信息,这为广大的环状RNA研究者提供了丰富有用的资源。
原文链接:
https://doi.org/10.1093/nar/gkad829
.png)
