2021年Briefings in Bioinformatics也发了类似的总结《The bioinformatics toolbox for circRNA discovery and analysis》,文章中描述了大约100个生物学工具。

<有了这篇文章,我们再也不用秃头整理资料了!\^o^/>
环状RNA起源
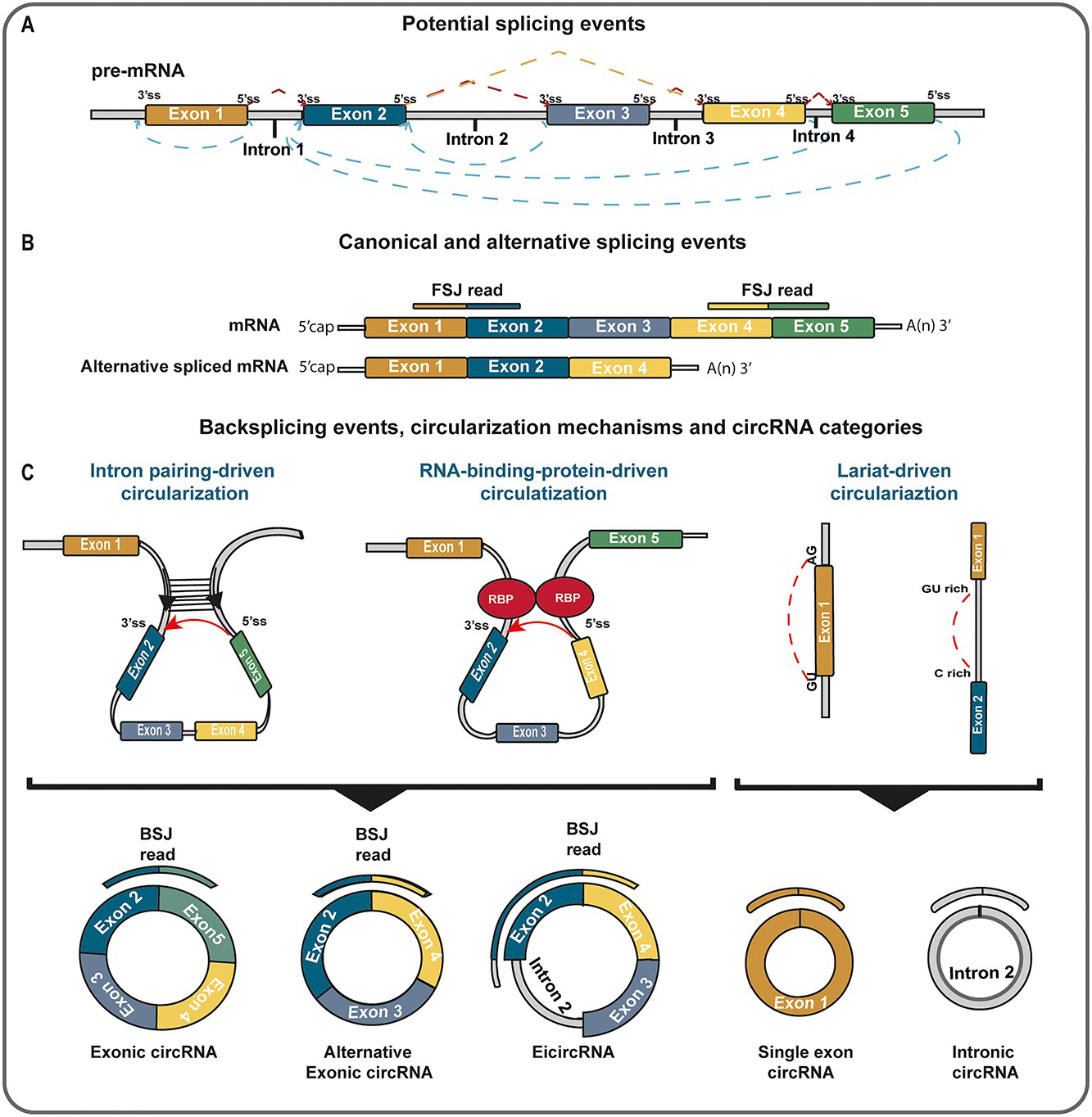
要研究环状RNA,那我们不得不知道环状RNA是如何生成的。目前大部分研究人员已经自动默认环状RNA主要是线性RNA转录后的产物,环状RNA分子主要通过侧翼内含子互补配对、RBP诱导侧翼内含子以及套索驱动3种方式产生(如下图),且根据环状RNA的基因组结构分成了EcircRNA、EIcircRNA、IcircRNA等不同亚型。
环状RNA识别
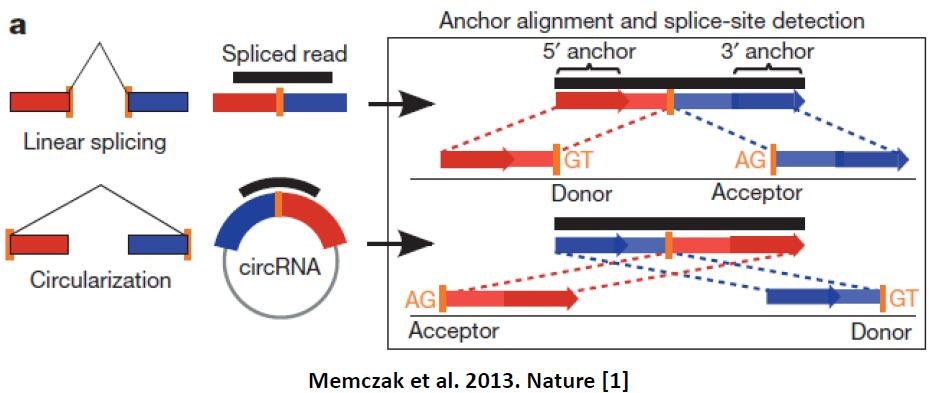
BSJ是识别环状RNA分子的关键,所有预测环状RNA分子的软件——从最初的find_circ到现在常用的CIRI、DCC、CRICexplorer等工具——都基于该理念(请看经典图)。
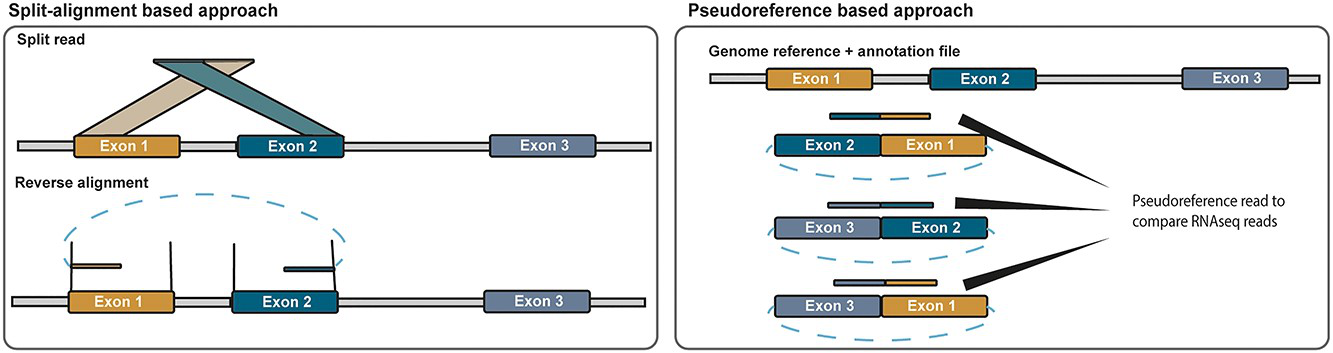
当然,不同的软件除了基本思想一致外,还存在两个门派:一个是Split-alignment based approach,另一个是Pseudoreference based approach。由于BSJ无法比对到正常参考基因组,第一种方法将chimeric reads或unmapped reads分割后再一点点与参考基因组比对;第二种是先推测BSJ的外显子,再将这些外显子拼接成假基因组(pseudo reference),然后再将 chimeric reads或unmapped reads比对到假基因组上(如下图)。

2017年
2018年
环状RNA可变剪切与重构
环状RNA全长测序技术
• circNick-LRS/circPanel-LRS
• circFL-seq
• isoCirc
在环状RNA全长识别、可变剪切分析以及表达定量等方面,三代测序技术有二代测序无法比拟的优势。高通量检测技术Illumina-basedRNA-seq/microarray/NanoString能检测定量BSJ序列,然而它们无法检测序列长度>300nt的环状RNA全长结构;而不同长度的环状RNA,纳米孔测序技术都能胜任。除此之外,三代测序还能够通过RNC建库检测正在翻译的环状RNA分子,并且能通过direct技术在一定程度上捕获到RNA修饰。
技术服务 | circRNA全长纳米孔测序
目前关于三代测序对环状RNA修饰的文章较少,感兴趣的童鞋可以查看文章《Profiling of circular RNA N6-methyladenosine in moso bamboo(Phyllostachys edulis)using nanopore-based direct RNA sequencing》。
环状RNA结构与体外制备
环状RNA全长测序技术解决了得到环状RNA全长序列(即一级结构)的挑战,为后续环状RNA功能研究提供了结构基础,例如预测miRNA的结合位点、RBP的结合位点。然而,对更高级的环状RNA结构的解析,才利于预测环状RNA功能解析,例如实现分子docking。另外,环状RNA的体外制备需要对环状RNA结构进行解构,这已经被应用到反义环状RNA的设计与应用、环状RNA作为药物靶点、可翻译的环状RNA框架等多个领域。
技术服务 | circRNA体外制备
另外,值得一提的是,我国在人工智能应用于生物医学的研究也走在了世界前沿。百度出品的深度学习算法LinearFold能准确预测RNA的二级结构,而5月2日公布的RNA序列设计软件LinearDesign更是登顶Nature。相信这些新的工具将会为环状RNA的研究注入新的活力,同时加速环状RNA在医药方面的开发。
环状RNA表达定量、标准化与差异分析
环状RNA相比线性分子mRNA和lncRNA更难定量。大量bulk RNA-seq数据显示,环状RNA只占整个样品文库的极少部分,其中间序列要受线性分子的影响,定量只能认准BSJ。因此,目前环状RNA定量最佳的建库策略应该是先通过RNase R等策略去除线性,只富集环状RNA分子。
产品推荐 丨RNA制备工具酶系列之RNase R
环状RNA功能预测
环状RNA功能的机制众多,大家最熟悉且研究最多的有三个:
作为miRNA的海绵
与RBP互作
另外,基于已有的研究信息,一些预测工具也被开发出来。catRAPID v2整合了大量环状RNA与RBP信息,是一款非常常用的在线circRNA-RBP预测工具;基于CLIP-seq以及机器学习的工具也频频出现,例如CRIP、CircSLNN、CCSCRSites、Clirc、circ-pSBLA、iCircRBP-DHN以及RBPsuite等,除了个别工具存在在线版本(例如RBPsuite)方便使用,其他工具使用难度都比较大,且RBP分子仅仅局限于37RBP(CRIP收集的CLIP-seq训练集)。
另外,通过circRNA pull-down+质谱的方法可能是一种更有效地方法研究某个环状RNA与哪些RBPs互作。
蛋白质翻译
关于一个circRNA是否能够翻译,我们可以通过CPAT、CPC2等工具先进行翻译潜能预测;另外我们可以用ORFfinder进行ORF的识别。值得注意的是,环状RNA的ORF可能覆盖环化位点BSJ,因此在进行操作时可能需要重复最多4次序列以避免错过有效的ORF或无终止密码子的ORF。
环状RNA的翻译不依赖于帽子结构,被归为“非经典”的形式,目前已发现大约3种翻译机制,包括:
– IRES驱动
– m6A驱动
– 滚环翻译(无终止密码子)
而关于IRES的预测,常用的生信工具有VIPS、IRESfinder、IRESPred、IRESpy、CircPrimer以及DeepCIP;而m6A位点预测常常用SRAMP以及m6Apred;至于滚环翻译的驱动机制目前仍有待进一步研究,并没有相应的工具帮助识别该现象。
另外,Ribo-seq数据也可能存在环状RNA的序列片段,为翻译提供线索,我们可以通过CircPro、CircCode等工具识别潜在翻译的环状RNA;但Ribo-seq文库太短,很难捕获到环状RNA的信息,RNC-seq可能是一种更有效的手段。
目前我们对环状RNA功能认识仍然有限。虽然有 circFunDb等数据库收集环状RNA功能,但由于环状RNA数量太多且一般被作为调控分子,因此,目前仍未形成像Gene Ontology和KEGG那样系统的功能和通路数据库。所以,环状RNA关于功能和通路注释大多通过以下方式开展:
• 对circRNA cis调控基因(许多文献非编码 RNAcis调控基因定义为基因组区域<10kb)进行GO/KEGG/……等注释;
• 对circRNA trans调控基因或表达相关基因进行 GO/KEGG/……等注释;
• ……
关于GO/KEGG/……注释以及富集的工具已经有很多了,但比较常用的可能还是R包 clusterProfiler、在线工具DAVID、在线工具 g:profiler等。
环状RNA实验验证
环状RNA的验证、过表达以及敲低实验关键是设计特异的环状RNA序列,主要包括环状RNA引物设计和siRNA设计。其中CircPrimer是一款 Windows版本的环状RNA引物设计工具,而 circInteractome针对circBase数据库中的环状 RNA进行了引物设计,同时circInteractome还包含了siRNA的信息。
环状RNA数据库
环状RNA研究十年间已经发表的数据库差不多有 60个,之前已经作了比较详细的总结,请查看:
目前各个数据库并没有统一环状RNA的命名,且参考基因组版本也存在差异——大多数使用的是 hg19版本,有一些采用了hg38版本。对此,今年年初,陈玲玲等研究者就该问题给出了环状RNA命名规范的建议,这将有利于环状RNA社区间的交流。
环状RNA可视化工具
• shiny版本的circASViewer,用于查看环状RNA可变剪切事件的小工具;
• shiny版本的circASViewerStatic,该工具与 circASViewer是姊妹工具,能够生成环状RNA可变剪切事件的静态图;
• Rcirc是一个R包,可以像IGV一样可视化环状 RNA的测序数据;
• 在线版本的circVIS主要展示环状RNA与宿主的关系,更像一个数据库(目前无法使用);
• ……
另外,如果要绘制环状RNA与其他分子的互作图,Cytoscape软件是大家的首选。
尾声:一图胜千言
下图对文章原文提到的工具进行了概述,分别从环状RNA研究热度、识别工具、数据库和下游分析工具4个方面以及时间轴等多个维度对环状 RNA生物信息工具进行了总结。
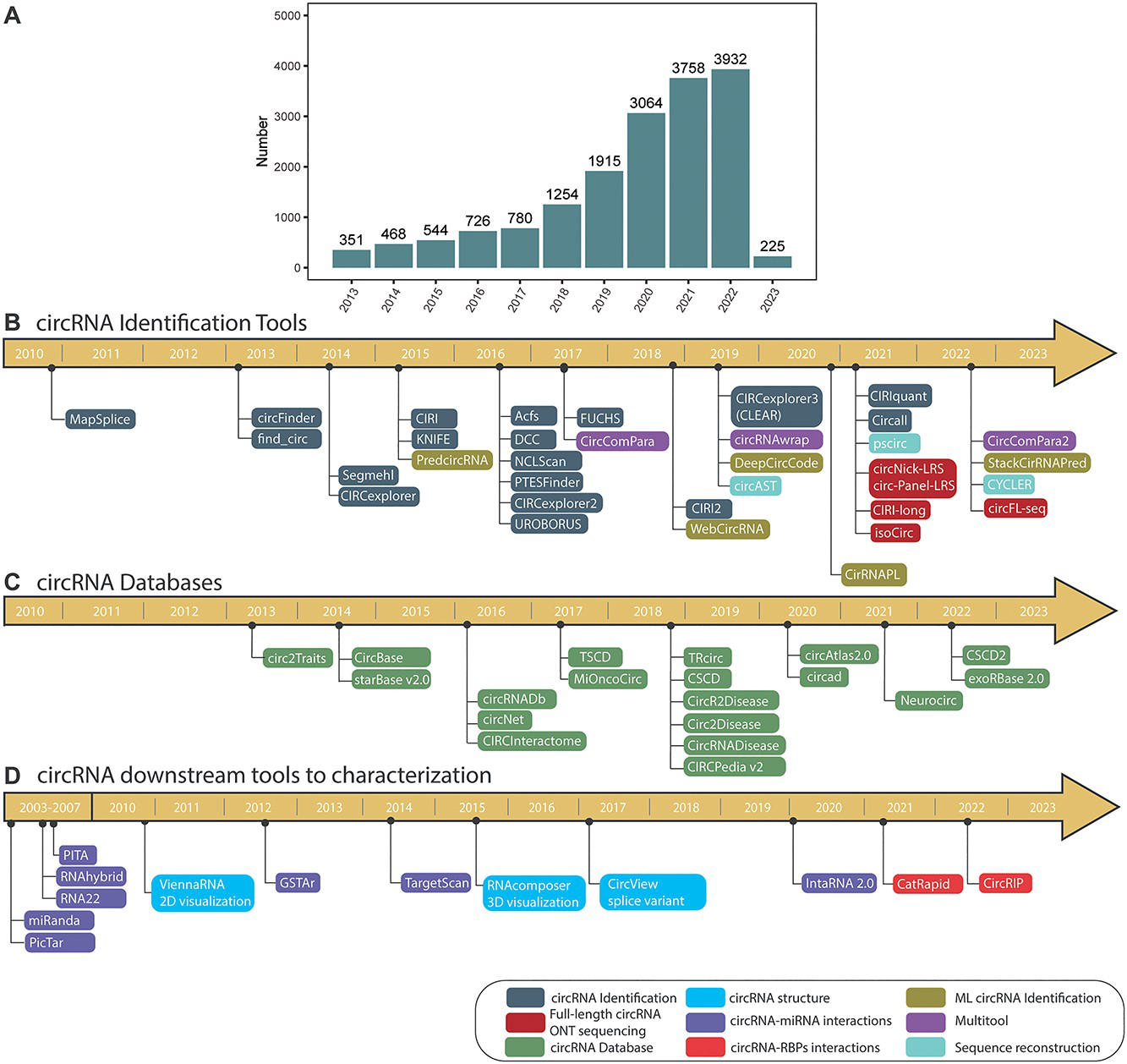
各位看官在环状RNA研究中都用到过哪些工具呢?欢迎留言讨论~

.png)