刚入门circRNA研究的小伙伴们,平时可能会碰到一个很重要的小问题。就是文献中重点研究的circRNA的完整序列到底是什么?CircRNA接头序列怎么找?因为相当一部分文章在正文或补充材料里不会直接给出circRNA的全长序列,或对应的数据库ID。当然随着circRNA研究的推进,这种状况会慢慢改善。但如何准确快速获得感兴趣circRNA全长序列,任然是初学着应该首先掌握的技能。
本文以最近发表的一篇circRNA文献为例,跟大家一起交流如何不通过写代码快速获得circRNA全长序列。

该文研究者发现的一种全新的circRNA,起源于SCD基因的3UTR区,命名为SCD-circRNA2。实验结果表明SCD-circRNA2在肝癌组织中显著上调,是肝癌预后独立预测因子。进一步研究发现RNA结合蛋白RBM3在肝癌中高表达,与肝癌预后不良相关,RBM3高表达促进了SCD-circRNA2的表达,是SCD-circRNA2的上游调控因子。
注:
硬脂酰辅酶A 去饱和酶(stearoyl-CoA desaturase, SCD) 是合成单不饱和脂肪酸的限速酶,是调节肝脏脂肪生成和脂类氧化的关键控制点。该酶活性的改变会导致脂质代谢异常,从而引起各类脂肪代谢相关疾病的产生。
下面我们一起根据文献给出的数据通过3步来寻找SCD-circRNA2的全长序列。
收集文献中circRNA序列相关信息
首先我们罗列出文章中SCD-circRNA2相关信息。
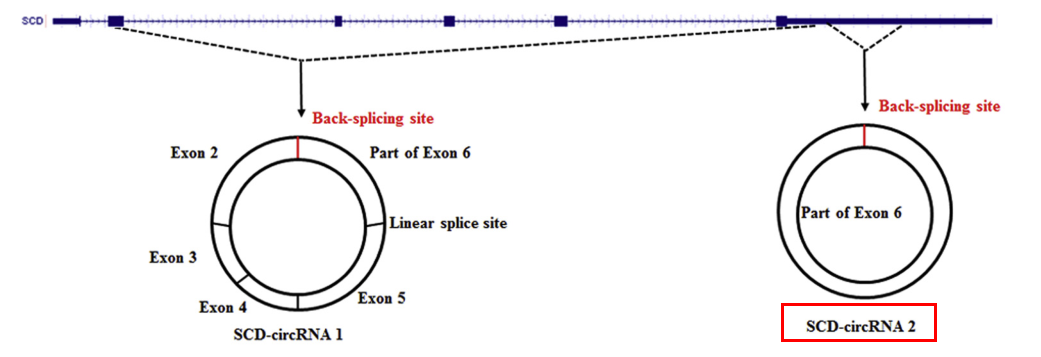
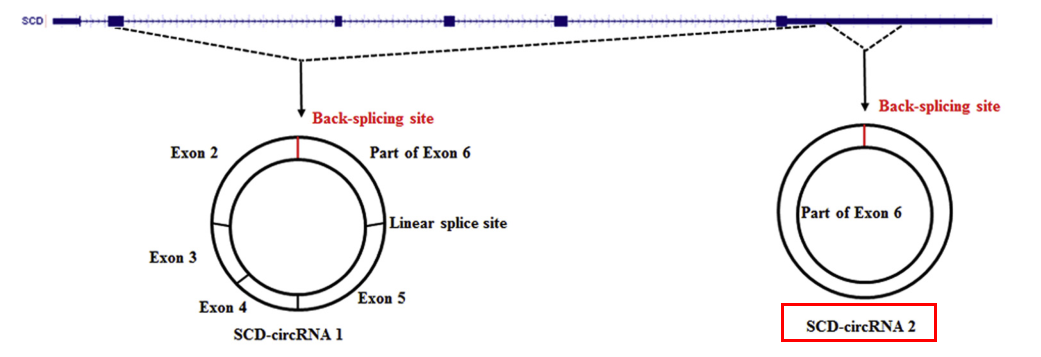
- Fig1c给出了SCD-circRNA1和SCD-circRNA2在SCD基因上起源定位示意图。Part of Exon6说明SCD-circRNA2来源6号外显子部分序列。
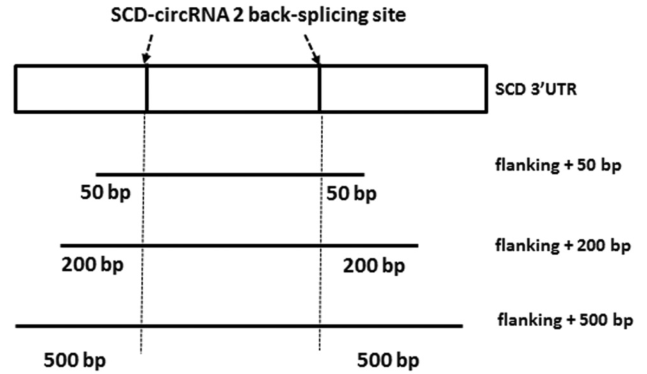
- Fig6a给出SCD-circRNA2序列侧翼序列相对位置,
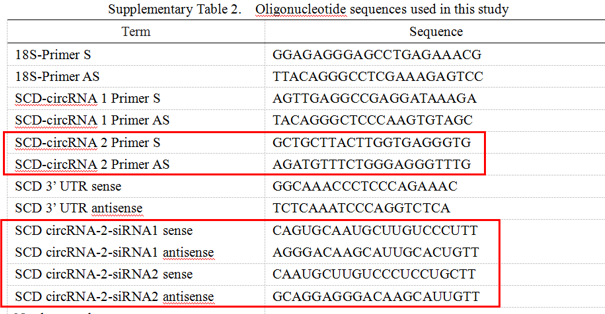
- 补充材料Supplementary Table 2材给出了SCD-circRNA2的PCR,siRNA和northblot探针序列。
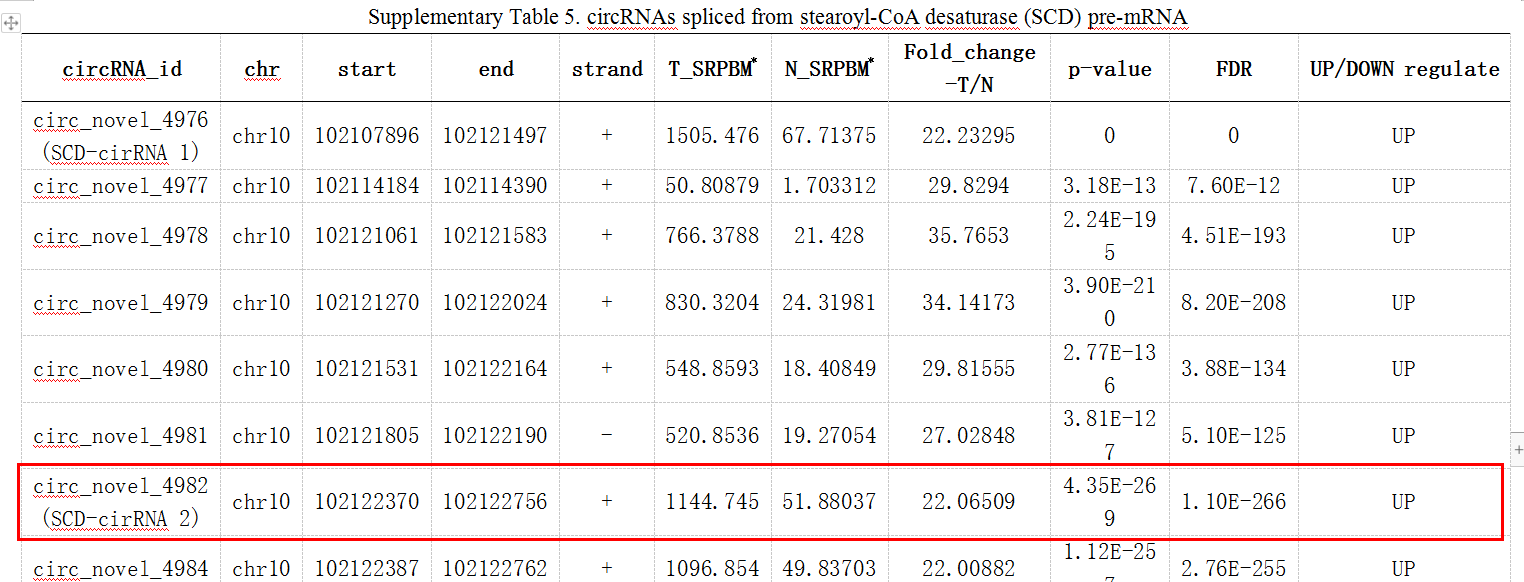
- 补充材料table5给出circRNA高通量测序SCD-circRNA2的转录起始终止染色体坐标
chr10:102122370-102122756
2.通过在线数据库UCSC genome browser检索circRNA序列
对于全新circRNA,一般类似circbase等数据库不会收录,所以检索不到。这里我们可以根据上面circRNA测序提供的SCD-circRNA2染色体坐标在UCSC genome browser(http://genome.ucsc.edu/)网站上进行分析。
- 进入UCSC基因组浏览器后,鼠标悬停Genomes处,点击进入Human GRCh37/hg19界面(此处选的是hg19基因组版本,不知道基因组版本的情况下,可能还要试试hg38版本)。
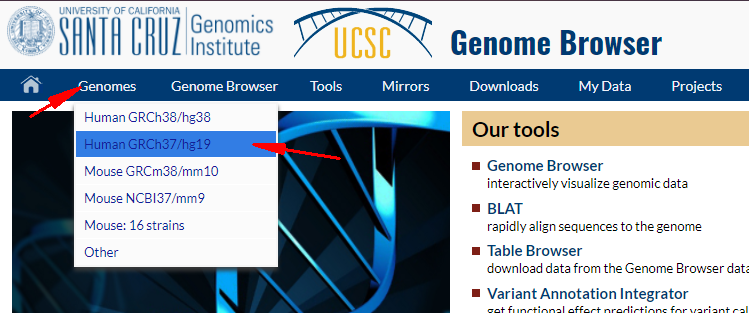
- 将circRNA染色体坐标chr10:102122370-102122756粘贴进搜索框,鼠标点击go按钮。页面会刷新进入如下界面,可以看到浏览器已经跳转到SCD基因区。

- 查看SCD基因的详细信息并确认。
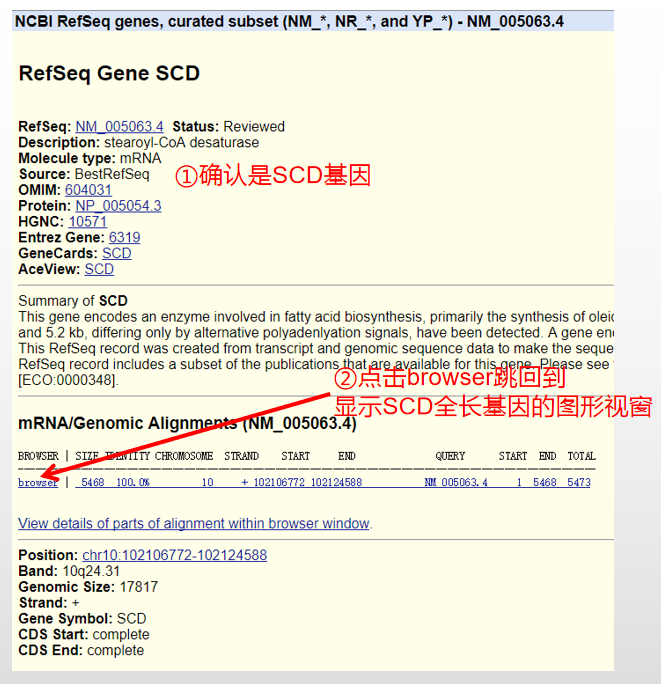
- 查看SCD基因全长结构,下图可知道SCD基因由6个外显子组成。
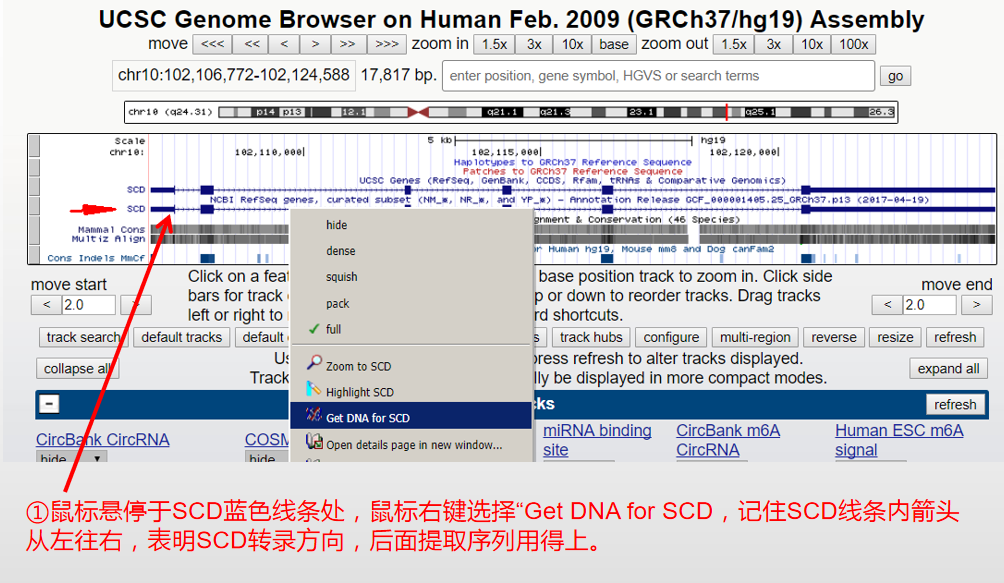
- 根据上图点击“Get DNA for SCD”
注:UCSC坐标系统以0开始的
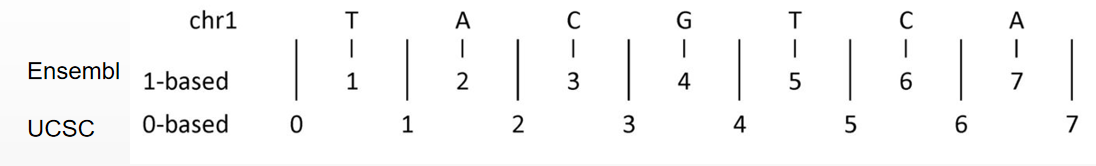
由于Ensembl 数据库用 1起始的坐标系统,而UCSC 用0起始的坐标系统,因此这里需要将SCD-circRNA2的起始坐标加上1,即SCD-circRNA2坐标为chr10:102122371-102122756
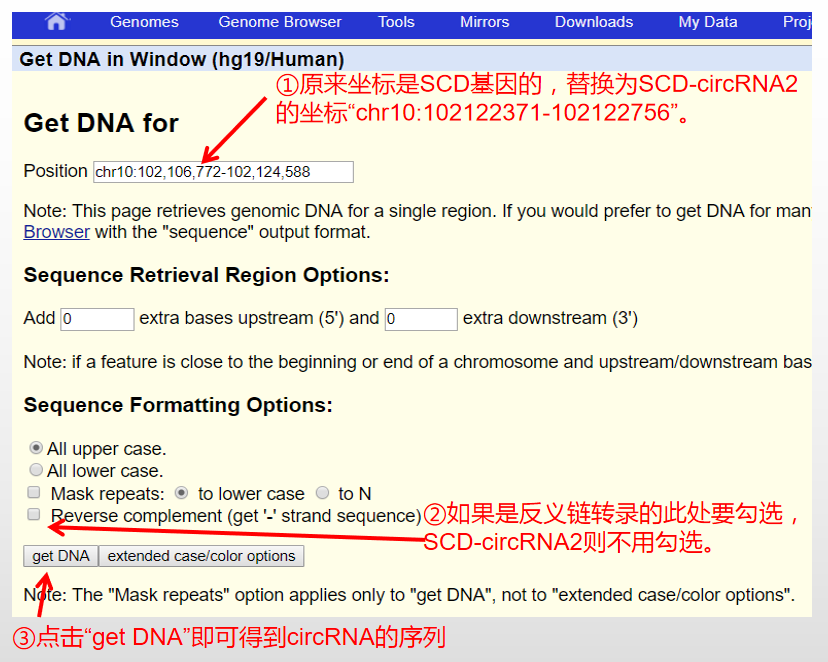
- 获得SCD-circRNA2的全长序列,并转换得到接头序列,转换方式见下图。
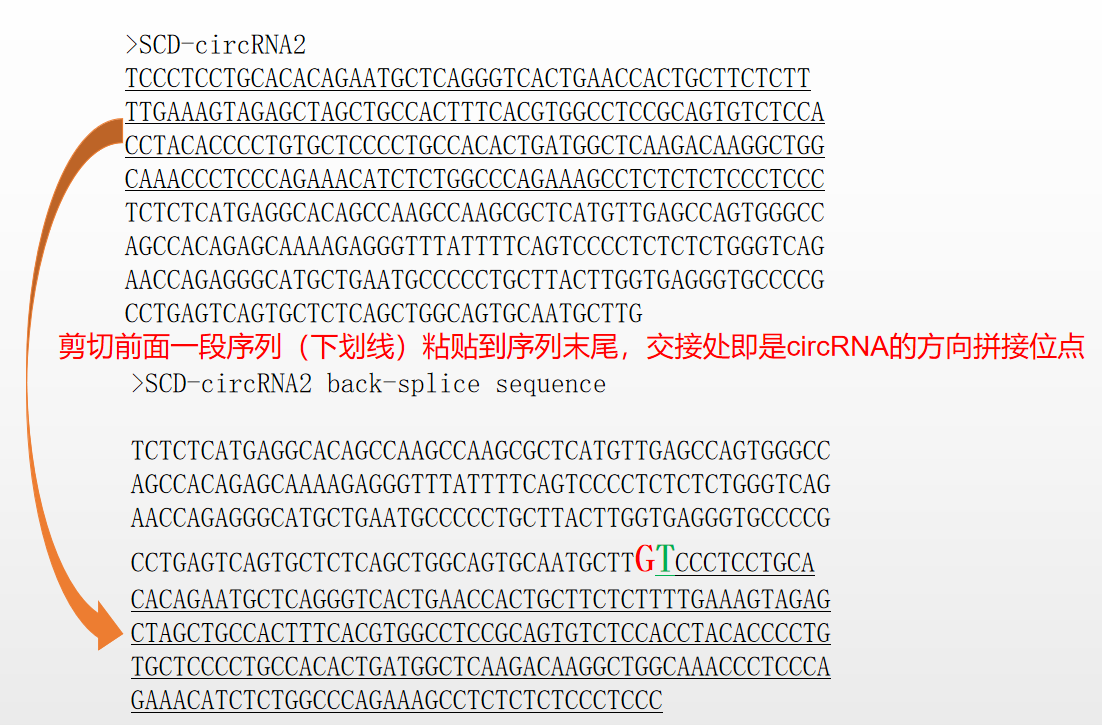
3.验证获得序列的正确性
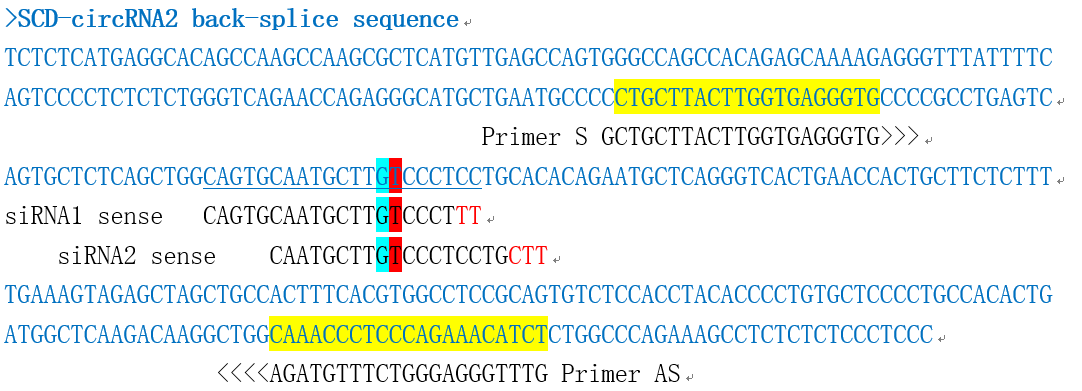
- 利用文献中给出的PCR引物和siRNA序列,考察是否可以对应到已获取序列中。
- 利用UCSC genome browser的blat工具验证获取SCD-circRNA2序列准确性。
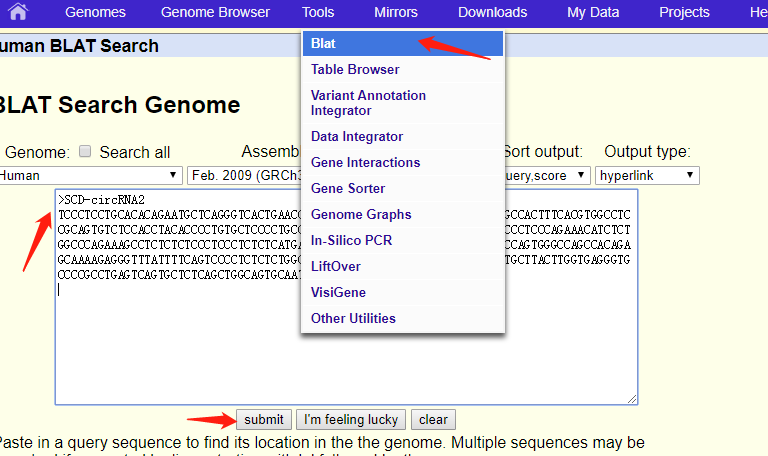
点击submit提交后,跳转到下面界面。
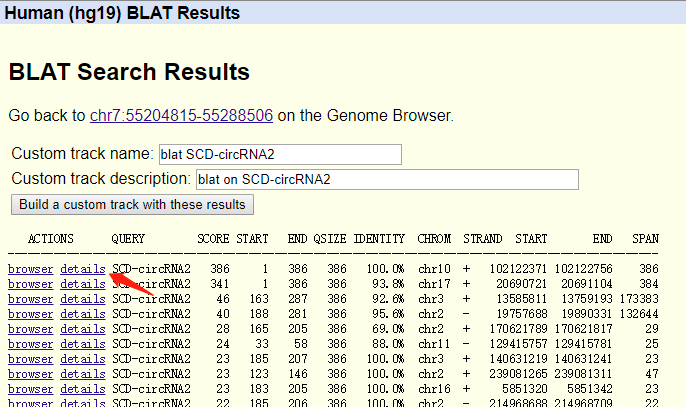
点击第一条记录的browser,跳转到下面图形界面。
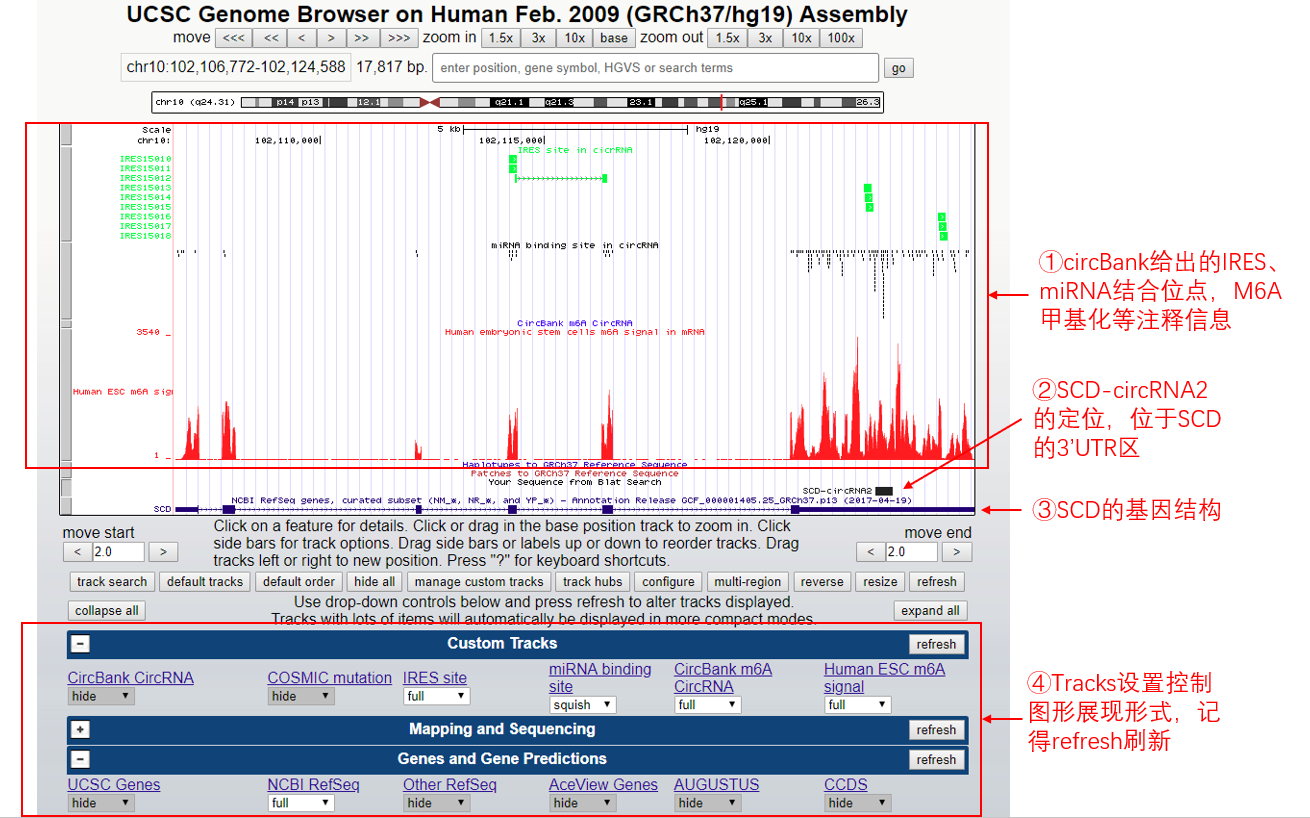
上图SCD-circRNA2和SCD的定位图是不是跟文献的Fig1c一致了。
参考文献:
- Dong W, Dai Z, Liu F, et al. The RNA-Binding Protein RBM3 Promotes Cell Proliferation in Hepatocellular Carcinoma by Regulating Circular RNA SCD-circRNA 2 Production[J]. Available at SSRN 3346501, 2019.
- Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, Haussler D. The human genome browser at UCSC. Genome Res. 2002 Jun;12(6):996-1006.
- Kent WJ. BLAT-the BLAST-like alignment tool. Genome Res. 2002 Apr;12(4):656-64.
—————————————————END————————————————————
报名参加“circRNA测序及数据挖掘入门培训班”,解锁更多新技能。

本期培训班由具有多年circRNA-seq高通量测序及数据分析经验,以丁向明博士领衔的专业讲师团队授课。讲师成员在circRNA测序及数据挖掘方面具有深厚研究功底,团队打造的人类circRNA综合数据库——circBank,贴近circRNA实际科研需求,上线短短两个月即被多篇Molcular Cancer、Aging-US等国际知名期刊文章引用。
授课讲师结合团队多年研究经验精心准备,从circRNA-seq实际案例出发,手把手教您通过R语言和生信工具实现circRNA差异统计分析、基因富集分析和热图、火山图、RCircos图、分子调控网络图绘制等。培训内容以基础教学为主,为零经验、想入门生信分析的您提供最佳学习路径。
培训时间:2019年8月1-2日(周四、五)
地点:广州歌尔爵斯酒店(禺东西路40-2号,广州火车东站附近)
名额有限,限报30人,报完即止;
培训费用:3000元(交通费与食宿费自理,仅提供茶歇和午餐)
优惠政策
1. 2019年7月15日前报名,优惠500元;(以汇款到账日期为准)
2. 报名前10名,可免费参加“第五届circRNA研究论坛”
3. 团购优惠:2人组团报名,每人可优惠100元、3人组团报名,每人可优惠200元、4人及以上组团报名,每人可优惠300元;
4. 发票内容为:培训费,缴费发票均在现场领取。(仅开增值税普通发票)
报名方式
1. 长按识别下方二维码,填写信息报名;
2. 邮件发送姓名、单位、电话到联系人邮箱,主题注明报名“circRNA测序分析培训班”;
3. 汇款时请务必注明学员姓名、单位,汇款后将汇款凭据电子版发送至邮箱cq@geneseed.com.cn,以确保汇款安全到账。
联系人:陈老师
联系电话:13302232899
联系邮箱:cq@geneseed.com.cn

长按二维码报名
.png)