大家在研读RNA与蛋白相互作用技术的文献中肯定看到过CLIP实验,究竟这个技术是什么?能用来解决哪些问题?今天就让我们通过一篇综述文章来一起学习一下CLIP技术:
这篇综述是Molecular Cell杂志于2018年2月1日发表的,文章的作者是伦敦大学学院(UCL)女王广场神经生物学研究所的Flora C.Y. Lee和Jernej Ule [1]。

什么是CLIP技术?
在研究RNA与蛋白相互作用的实验技术中经常能看到CLIP这个技术的影子,那么什么是CLIP技术呢?CLIP的英文全称是:Crosslinking and Immunoprecipitation。不难理解,CLIP的意思就是基于交联后的免疫沉淀分析。交联的预处理能够锁定RNA结合蛋白(RBP)与靶分子的准确结合位置,因此CLIP实验能准确获得相互作用的分子位点,是高精度分析RBP与RNA相互作用的有力工具。
根据交联预处理条件,沉淀方法及实验目的等体系的不同,CLIP实验可划分为很多种技术,如下表所示:
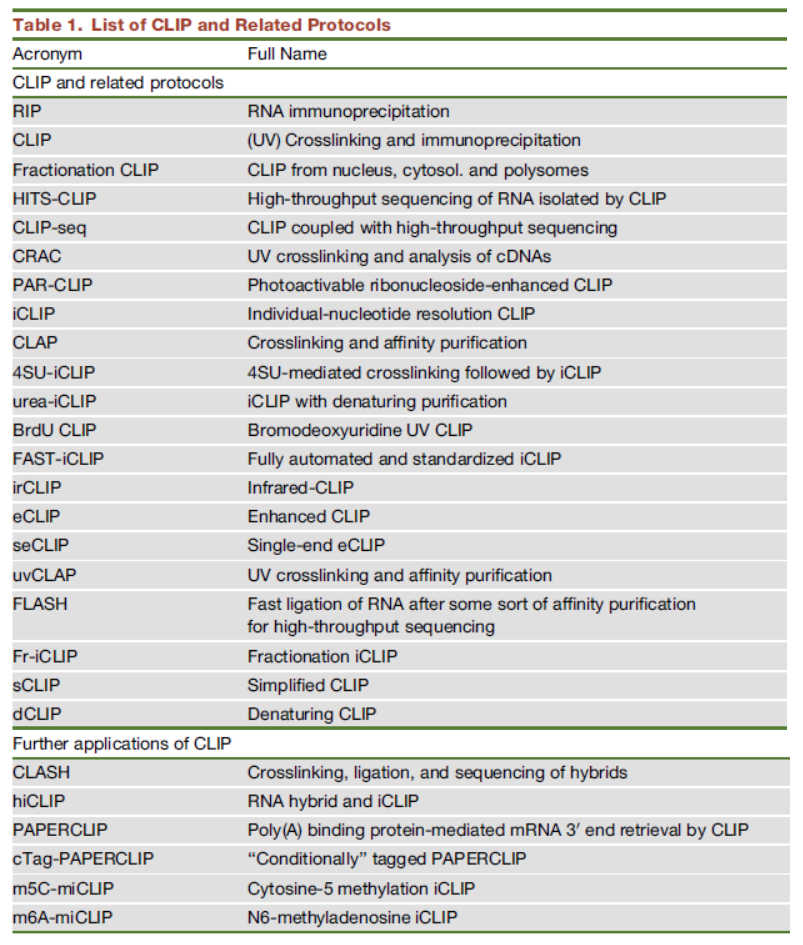
表1 CLIP技术的分类 ([1])
CLIP实验的核心步骤
这么多分类的CLIP方法一定让人眼花缭乱了,但仔细分析就会发现,这些分类只是因为它们的实验目的和所使用的分离方法等有所差别而导致的,核心的CLIP实验主要包含了以下几个方面:待检测样品的交联预处理和细胞裂解;RNA片段你的纯化回收;cDNA文库构建与测序分析。具体的每一部分详细的内容如下图所示:
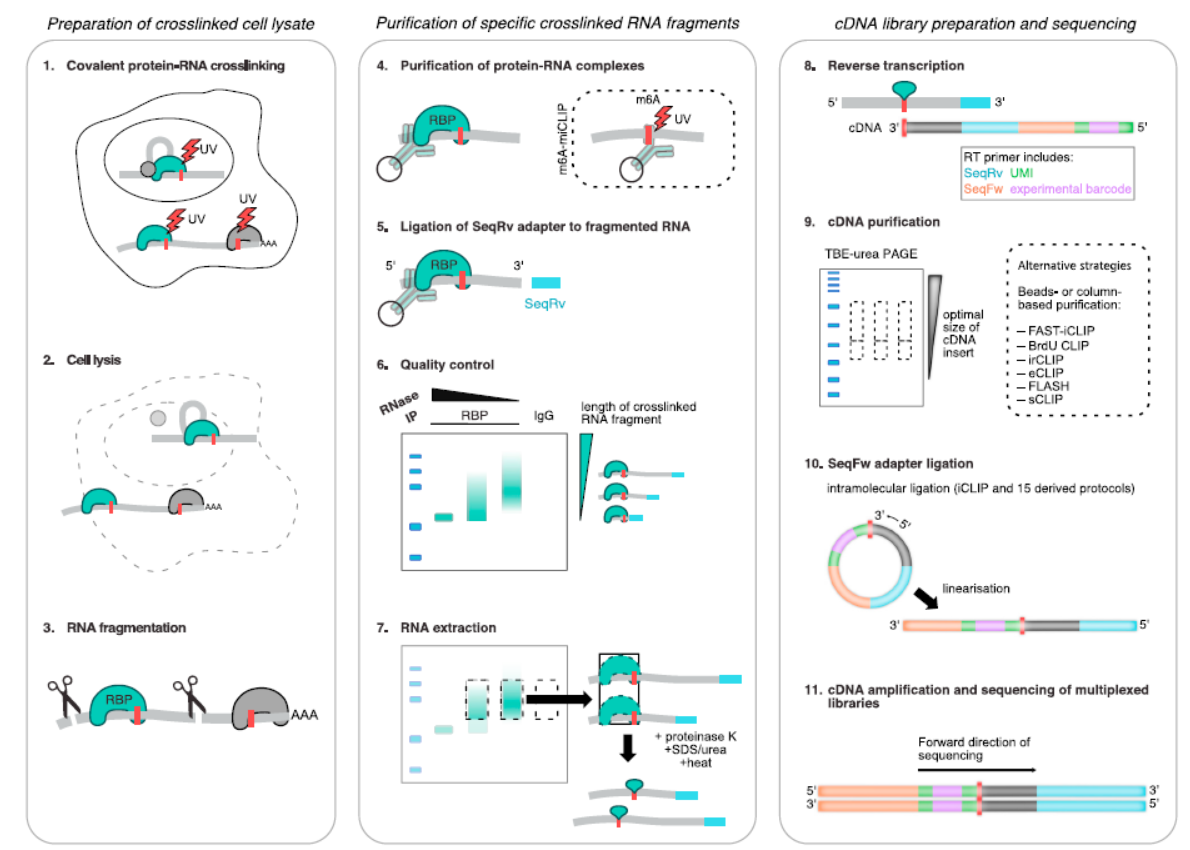
图1 CLIP实验的主要核心步骤 ([1])
CLIP中常用共价交联条件
共价交联是CLIP实验的关键步骤,有利于锁定RBP与RNA瞬间结合的位点。紫外交联是最通用的较量方法,紫外线不能诱发蛋白分子之间的交联,只有当一些氨基酸侧链与RNA碱基充分接近的时候才能被紫外线诱导共价交联。常用UV-C(254nm)进行,单层细胞用150 mJ/cm2 的能量下照射40S。PAR-CLIP中用到光诱导活化的特殊碱基4SU或6SG,它们先通过培养孵育掺入到RNA链中,然后可通过UV-A(365nm)进行交联,但这种交联方法存对一些RBP与蛋白的相互作用存在一定的影响,这两种试剂也有一定的细胞毒性作用。在m5C-miCLIP中,用到一种RNA甲基化酶的突变体(NSun2),这种突变体能够直接与m5C碱基共价交联,可用于靶分子的分离。m6A-miCLIP中用到的是抗m6A抗体,通过UV-C实现m6A碱基与抗体的共价交联。
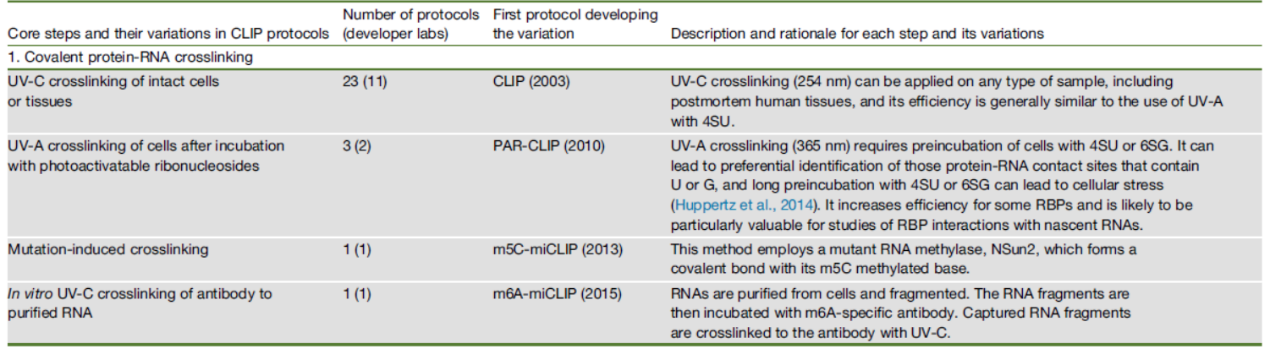
表2 CLIP中常用的共价交联方法 ([1])
CLIP中常用细胞裂解条件
CLIP实验中裂解细胞的Buffer常用离子型的去污剂,以充分解离没有共价交联的蛋白复合体和游离的RNA序列,通常是进行全细胞的裂解。但在一些特殊需求的CLIP体系中,需要首先分离亚细胞组分,然后分析其中的RBP-RNA相互作用情况。
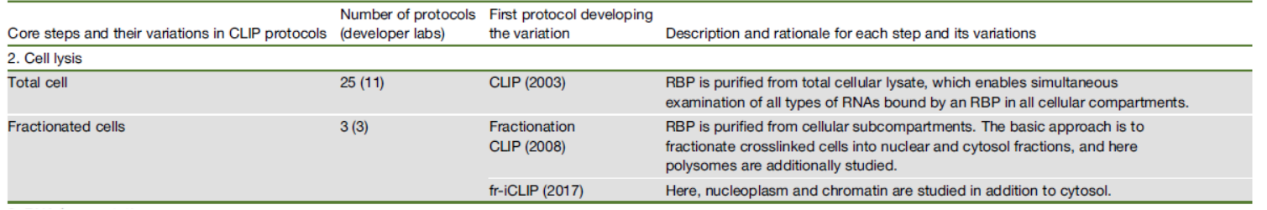
表3 CLIP中常用的细胞裂解方法 ([1])
CLIP中常用RNA片段化方法
在分离蛋白-RNA复合物之前,需要先通过酶解等方法将没有被蛋白结合的RNA区域消化掉,以充分保留和富集RBP结合RNA的片段。早期的CLIP体系中用RNAse T1或RNase A,这两种酶有一定的偏好性,不能非常准确去除RBP结合位点之外的linker序列。在iCLIP中引入了RNase I,这种酶目前没有发现有序列偏好性,因此能更好的在CLIP体系中反映RBP的结合位点。irCLIP体系中引入了S1核酸酶,这种酶酶切产物是3’ -OH而非3’ -磷酸基,因此可以省去后续去3’ -磷酸基的反应步骤,对后面的加3’ -接头的步骤有帮助。
在RNA片段化的过程中需要注意酶切过度和酶切不足的问题,因此需要针对反应的体系进行条件优化,常用SDS-PAGE的方法分析酶切条件是否合适。在一些CLIP体系中为更精确的获得相互作用位点,还在裂解液中片段化处理的基础上增加on-Bead的RNase消化步骤。
除了酶切进行RNA的片段化,在一些特殊的CLIP体系中还用到盐离子介导的RNA片段化条件,例如在m6A-miCLIP中用到锌离子进行RNA片段化。
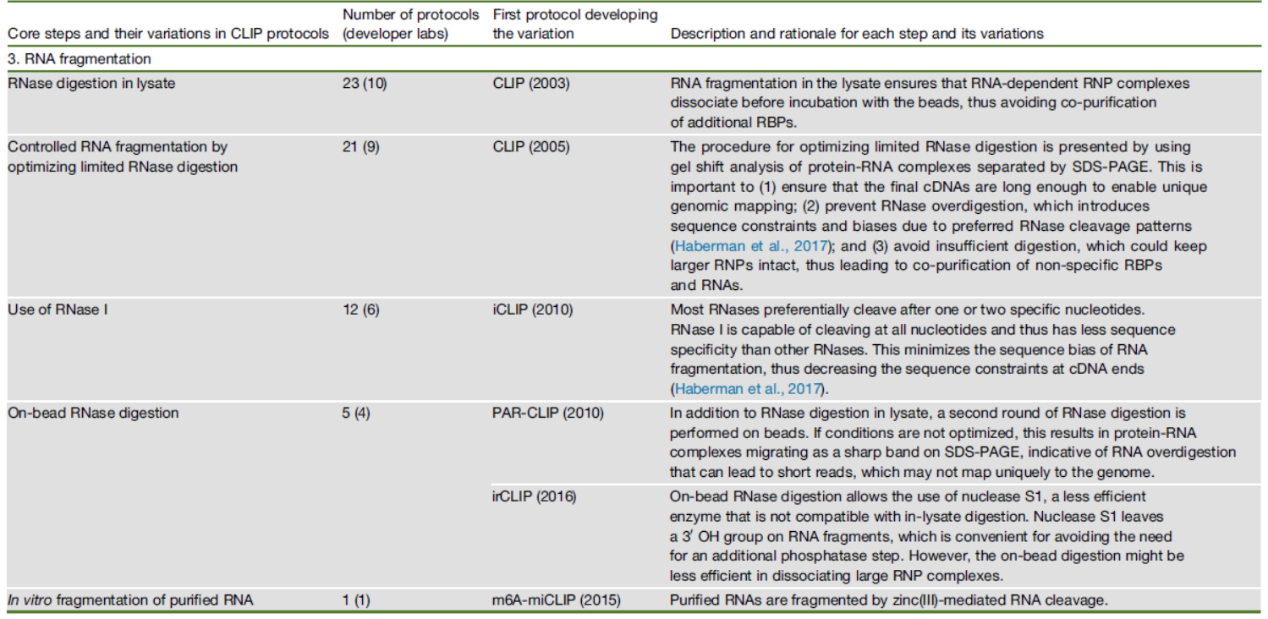
表4 CLIP中常用的RNA片段化方法 ([1])
CLIP中常用蛋白-RNA复合物纯化方法
基于免疫沉淀分离RBP-RNA复合物是比较高效快速的分离方法,IP的条件可以是在温和的Buffer 体系,也可以在比较强的去污剂条件下进行。如果将待分离的蛋白融合表达标签,则更有利于使用特殊的洗脱条件。
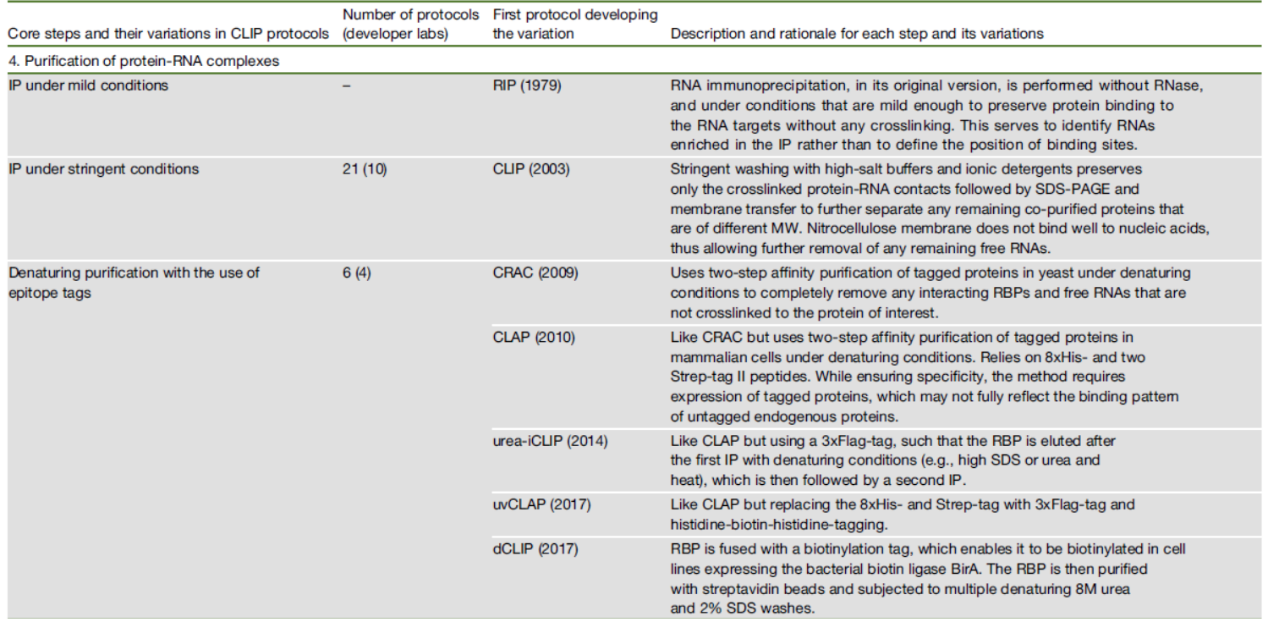
表5 CLIP中常用的蛋白-RNA复合物纯化方法 ([1])
CLIP中SeqRv接头连接方法
在分离得到RBP-RNA片段复合物后,需要将RNA片段的3’端加上接头,以方便进行下一步的反转录操作。大部分CLIP体系都采用标准的3’端接头,SeqRv。但在eCLIP和sCLIP中采用特殊的接头,eCLIP中采用带Barcord的模板转换接头,sCLIP利用增加多聚A尾实现3’端加反转录引物互补序列。CLIP实验中接头的添加可以在分离得到RNA片段之后,也可在分离RBP-RNA复合物的珠子上进行接头的连接。
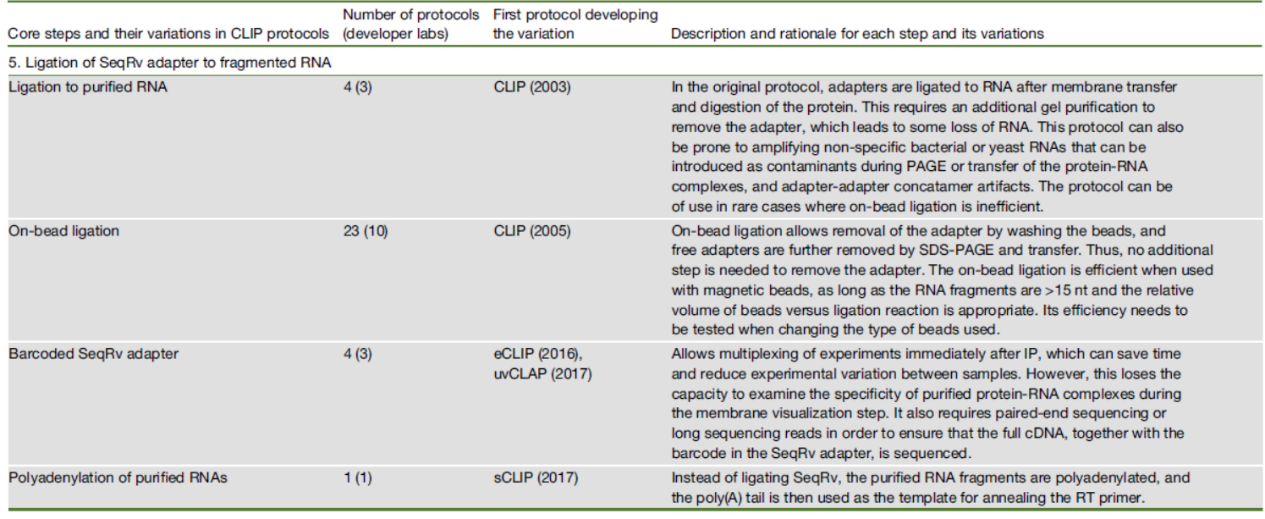
表6 CLIP中常用的SeqRv接头连接方法 ([1])
CLIP中常用回收产物质量控制方法
为监测所获得的的RNA产物是否合格,需要在连接接头之后进行质量控制,最常用的方法是用PAGE电泳的方法看片段大小的分布情况。如果条件允许,利用安捷伦2100等设备分析所获得的RNA片段质量也是可以的。
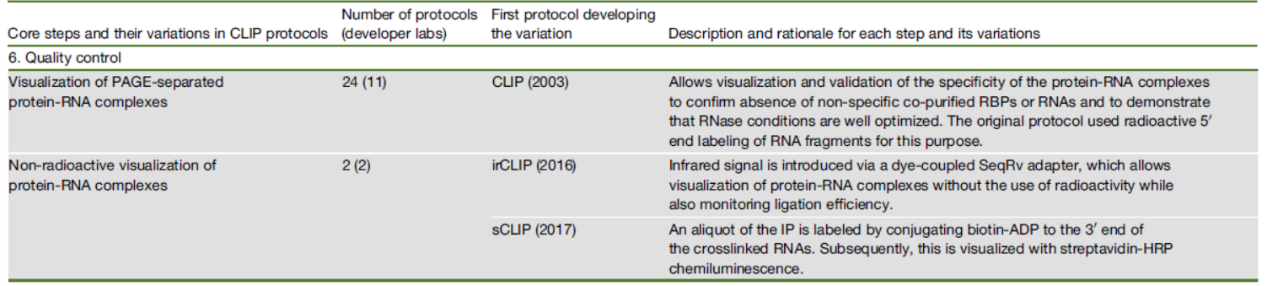
表7 CLIP中常用RNA片段质量控制方法 ([1])
CLIP中常用RNA片段分离方法
在获得了RBP-RNA复合物之后,需要将所结合的蛋白去除,常用地是用蛋白酶K消化去除,添加尿素或SDS有助于提高蛋白酶K消化的效率。最终得到的RNA片段可能会带有一小段多肽序列。
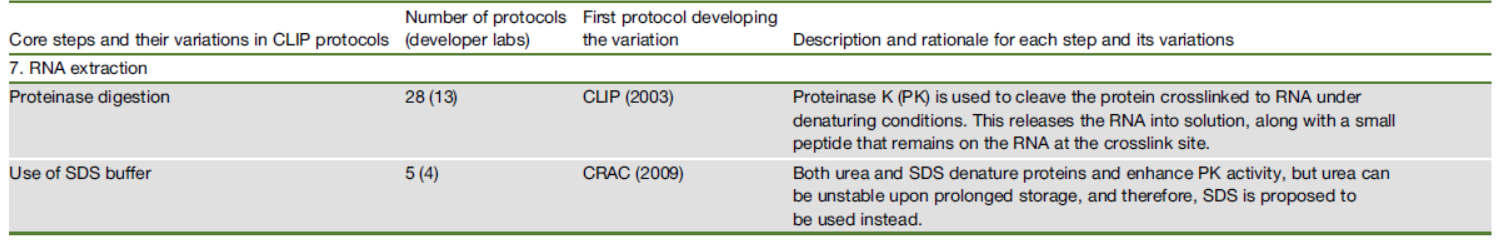
表8 CLIP中常用的去除蛋白的方法 ([1])
CLIP中常用反转录反应
SeqRv接头带有反转录的通用引物,可以用改引物对获得的RNA片段进行反转录反应,iCLIP中还用到UMI标签进行单分子RNA来源的标记示踪。
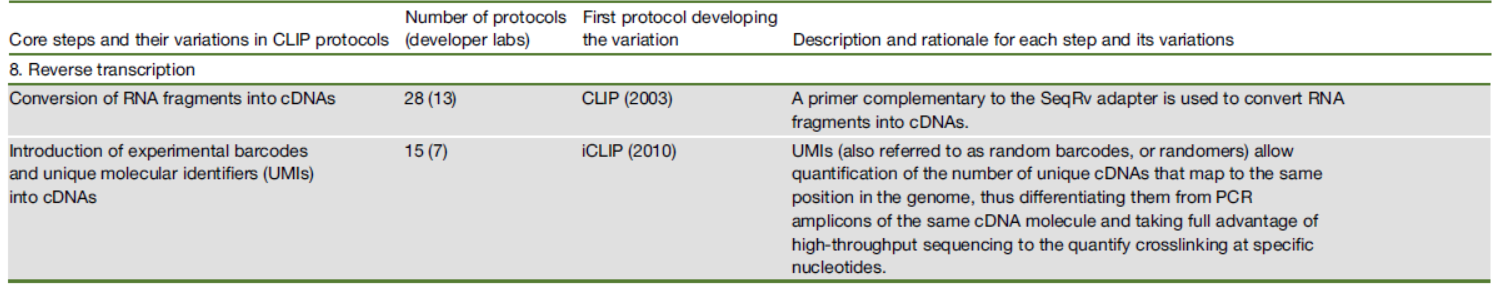
图9 CLIP中常用的反转录方法 ([1])
CLIP中常用cDNA纯化方法
传统的CLIP实验中可通过丙烯酰胺变性胶或TBE-尿素变性胶进行cDNA的回收。在BrdU CLIP中,反转录反应中掺入了BrdU碱基,因此可以通过抗体富集cDNA。Fast-iCLIP中将反转录的引物带5’-生物素标记,通过链霉亲和素珠子实现cDNA的快速富集纯化。还有其它基于柱子纯化的方法能够实现cDNA的快速纯化。
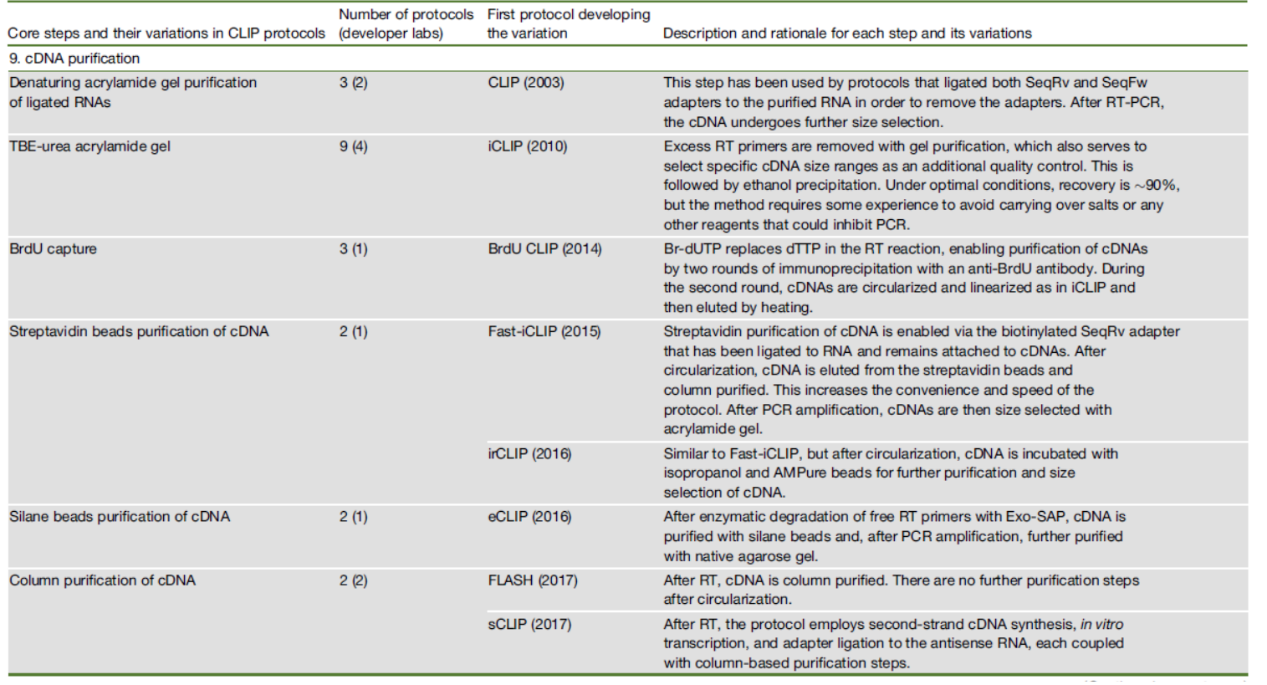
图10 CLIP中常用的cDNA纯化方法 ([1])
CLIP中常SeqFw接头连接方法
在早期的CLIP体系中通过在RNA片段的5’端连接SeqFw接头的方法实现这一侧接头的连接(同时将SeqFw和SeqRv接头接到RNA片段的两侧),iCLIP等体系中采用了另一种策略,只在RNA片段中加SeqRv接头,在反转录过程中引物的上游带上SeqFw的序列,然后在第一链cDNA反转录的产物利用circligase酶实现cDNA的成环,这样就能在这个环状单链cDNA分子中实现后续的建库扩增。
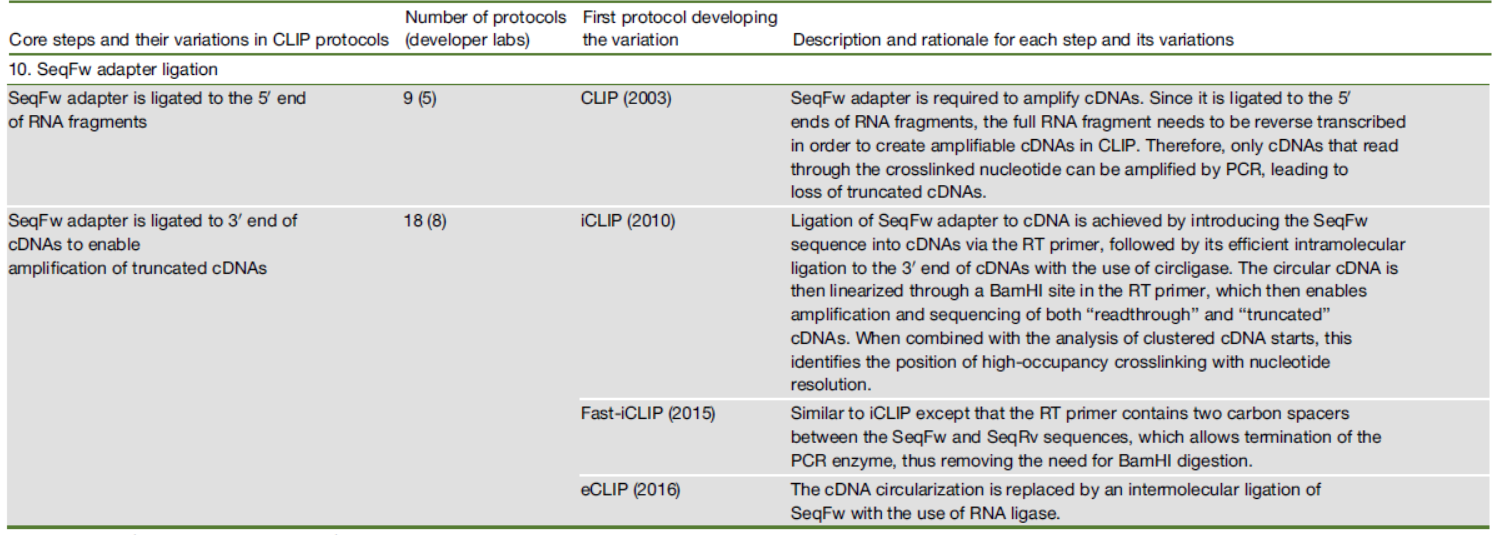
图11 CLIP中常用SeqFw接头连接方法 ([1])
CLIP中常用文库构建与测序分析方法
在早期的CLIP方法中,采用普通的一代测序方法,大规模的进行Sanger测序实现对结合位点的分析,但随着二代测序的不断成熟,后续的CLIP体系普遍采用高通量测序的方法进行序列测序和分析。
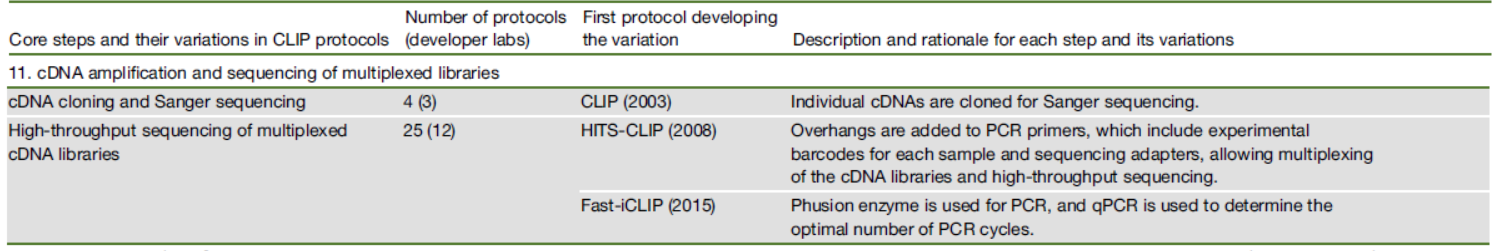
图12 CLIP中常用文库构建与测序方法 ([1])
本文系统回顾总结了CLIP实验的主要分类,实验条件和应用范围,通过本文的学习能较全面的了解CLIP实验基本原理,主要用途和实验细节注意事项。
circRNA与蛋白的相互作用同样适合通过CLIP体系进行分析,因此本文的相关内容值得各位同仁学习借鉴。
参考文献:
1. Lee, F.C.Y. and J. Ule, Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Mol Cell, 2018. 69(3): p. 354-369.
.png)