金秋十月已进入尾声,在十月份的下半月里,circRNA的研究进展保持平稳,也有一些亮点报道,如Nature Communications报道的人干细胞多能性有关的circRNA及机制研究等等。下面汇总如下:
1. 人干细胞多能性相关circRNA研究
10月27日,Nature Communications杂志(IF2016=12.124)在线发表了台湾中央研究院Kuo Hung-Chih为通讯作者的文章,报道发现circBIRC6参与干细胞多能性调控[1]。

文章报道发现干细胞中特异的RNA剪切因子ESRP1调控了circBIRC6的形成过程,circBIRC6通过竞争性结合miR-34a和miR-145参与调控干细胞多能性。ESRP1受到Oct4,Nanog等转录因子的调控。本文首次报道干细胞多能性相关的circRNA分子,是circRNA功能研究的重要扩展。
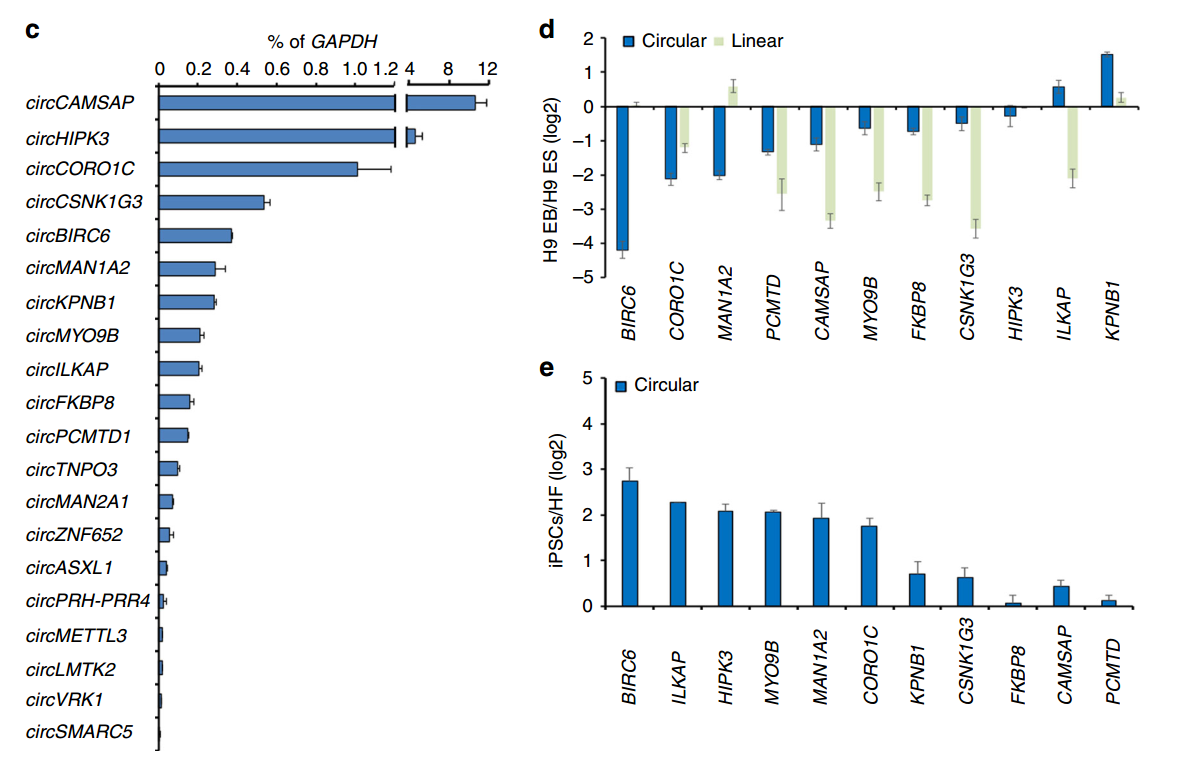
图1体外分化实验分析多能性相关circRNA分子 (来自[1])
2. 三阴乳腺癌中circGFRA1竞争性结合miR-34a
10月16日,Journal of Experimental & Clinical Cancer Research杂志(IF2016=5.189)在线发表了中南大学湘雅医院曾朝阳和中山大学附属肿瘤医院唐海林为共同通讯作者的文章,介绍在三阴乳腺癌中发现circGFRA1和GFRA1通过竞争性结合miR-34a在肿瘤发生发展过程中起作用[2]。

作者通过芯片法筛选了三阴乳腺癌细胞系与人乳腺上皮细胞中差异表达的circRNA,在高表达的circRNA中找到了circGFRA1。经过QPCR验证了circGFRA1在三阴乳腺癌细胞系和临床标本中的表达情况。与临床预后状况进行Kaplan-Meier生存分析后,结果表明,高表达circGFRA1的预后更差。干扰circGFRA1后抑制细胞增殖并促进细胞凋亡。预测分析结合荧光素酶分析后表明circGFRA1与GFRA1均可结合miR-34a,且circGFRA1与GFRA1的表达存在相互调控的机制。
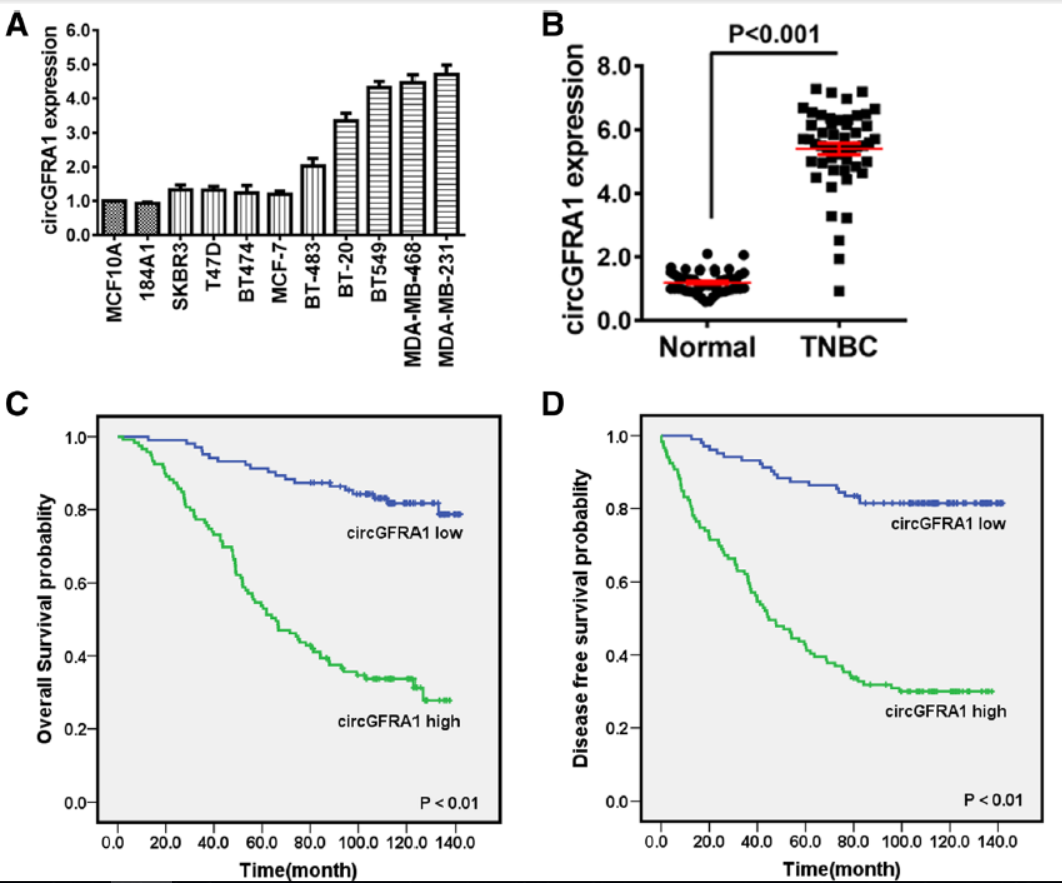
图2 三阴乳腺癌中高表达circGFRA1预后较差 (来自[2])
3. RAISE:一种新的circRNA测序分析流程方案
10月16日,International Journal of Oncology杂志(IF2016=3.0789)在线发表了北京协和医院肝脏外科郑永昌为通讯作者的文章,介绍他们开发的分析circRNA表达状况的流程方案:RAISE [3]。

概括而言RAISE流程的主要是通过Hisat2,STAR及circRNA_finder进行mapping和过滤比对验证,然后利用mapsplice,acfs和find_circ等工具筛选circRNA分子。作者利用该工具分析了来自于肝脏和脑组织的circRNA测序数据,发现在不同个体间存在较大差异,建议相关研究要充分考虑到个体间差异的影响。
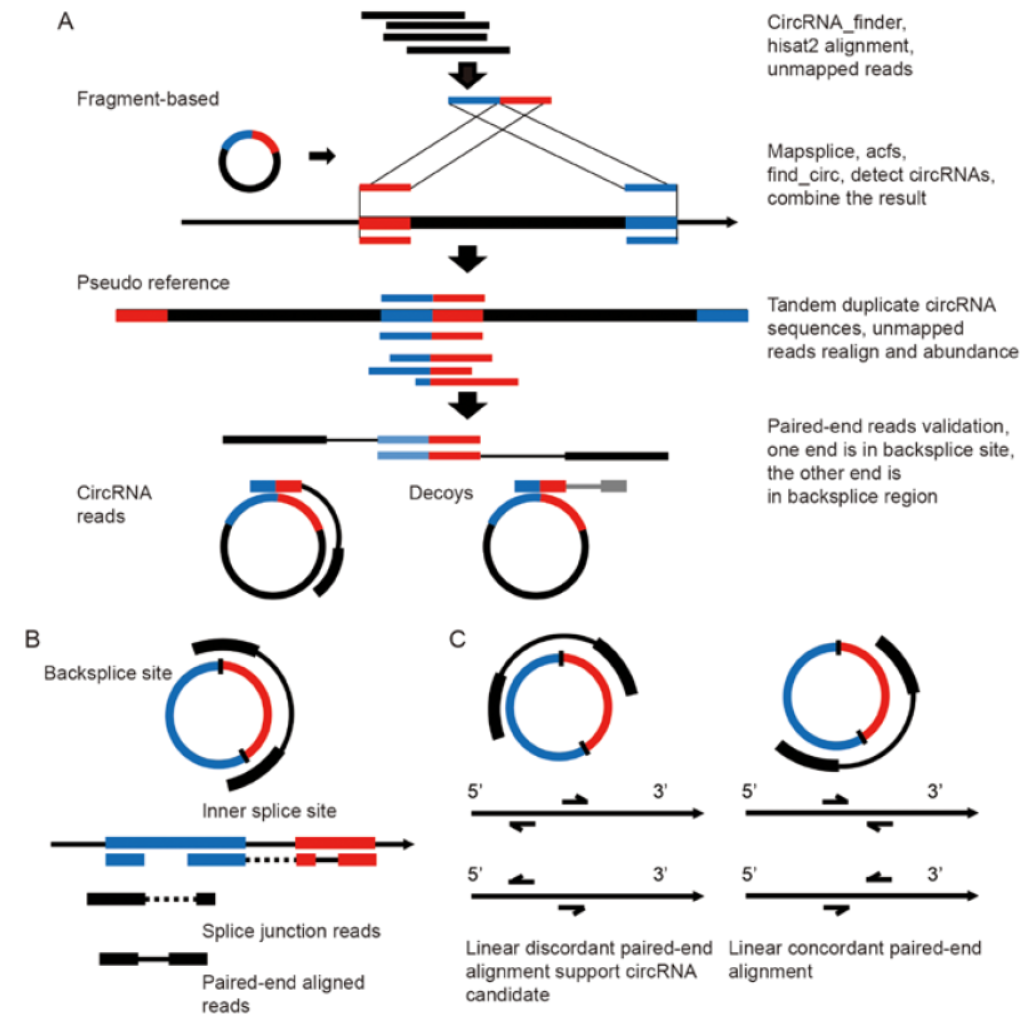
图3 RAISE技术流程 (来自[3])
4. 外周血circRNA可作为乳腺癌诊断标志物
10月13日,Clinica Chimica Acta杂志(IF2016=2.873)在线发表了大理大学附属第一医院Zhang Ruo-Peng和大理大学基础医学院Xiong Wei 为共同通讯作者的文章,介绍分析了乳腺癌外周血中疾病特异性的circRNA分子筛选与临床相关性的情况[4]。

作者基于芯片法分析了五例乳腺癌和健康志愿者对照组的外周血总circRNA变化情况,共找到41种变化超过2倍的circRNA分子,其中19种上调。后续又收取了20例标本进行了QPCR验证和ROC曲线分析,最终表明hsa_circ_0001785可能在乳腺癌的诊断中有一定价值。
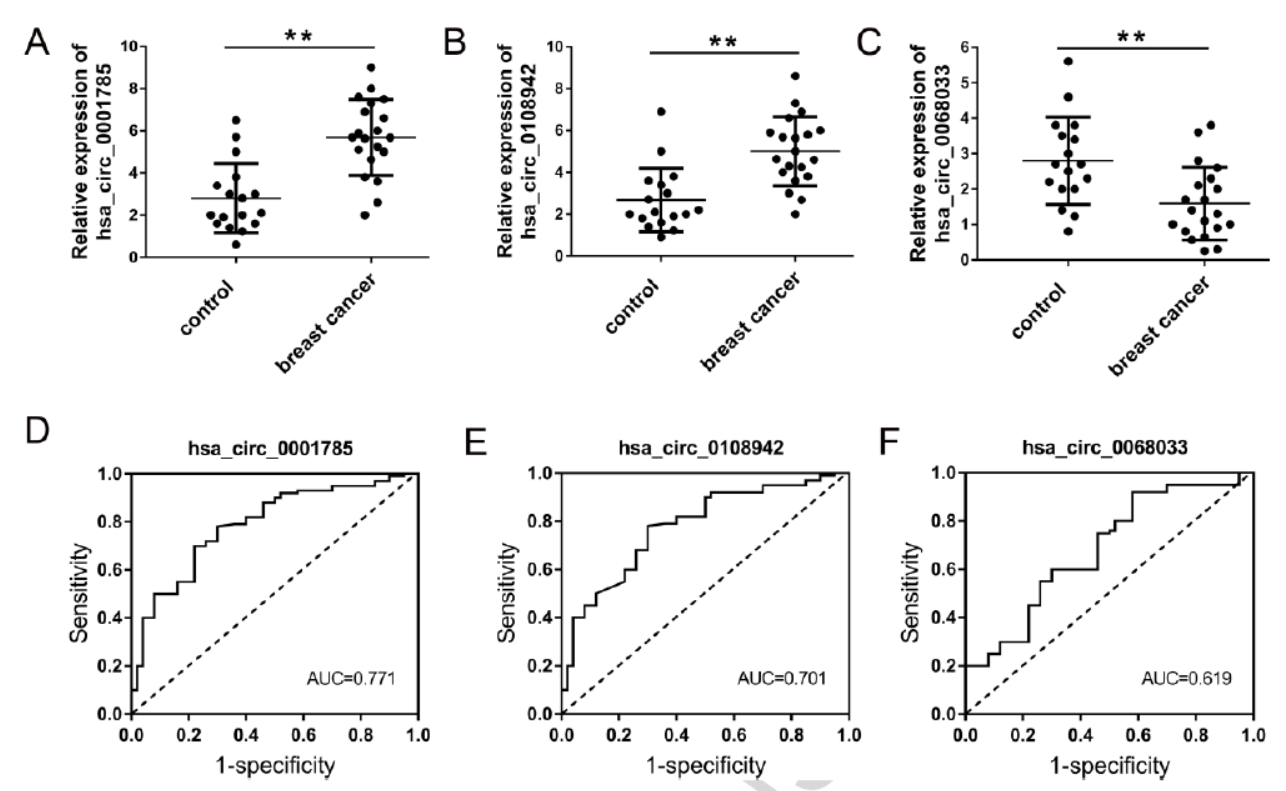
图4 乳腺癌特异性外周血circRNA (来自[4])
5. circWDR77调控FGF-2影响血管平滑肌细胞增殖
10月14日,BBRC杂志(IF2016=2.466)在线发表了山东大学附属省立医院崔连群为通讯作者的文章,介绍发现circWDR77通过竞争性结合miR-124影响FGF-2的功能,调控血管平滑肌细胞增殖和迁移[5]。

作者通过芯片法分析了高糖体外诱导血管平滑肌细胞中差异表达circRNA分子,共得到983种,其中458种为上调,其中31种circRNA和22种分别上调和下调超过了2倍。circWDR77便是在上调最明显的circRNA分子之列。信息学分析发现circWDR77可竞争性结合miR-124,影响FGF-2的功能。干扰circWDR77可明显抑制细胞增殖。RIP实验和荧光素酶实验证明circWDR77与miR-124相互作用。
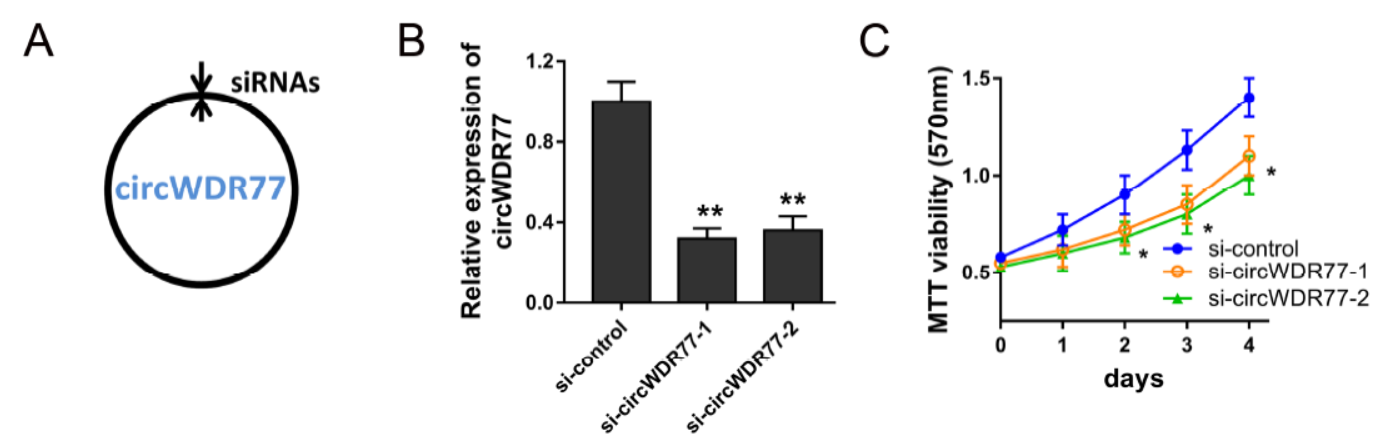
图5 干扰circWDR77可抑制血管平滑肌细胞增殖 (来自[5])
6. 肺转移的结肠癌患者circRNA的表达发生变化
10月16日,International Journal of Molecular Medicine杂志(IF2016=2.341)在线发表了昆明医科大学附属第一医院王昆华院长和罗华友为共同通讯作者的文章,分析了有肺转移的结肠癌患者circRNA的表达变化情况[6]。

作者通过芯片法分析了有无肺转移的结肠癌临床标本,比较了差异表达的circRNA,然后基于信息学技术预测分析了可能相互作用的miRNA分子和通路。共找到了431种差异超过2倍的circRNA分子,其中192种上调。
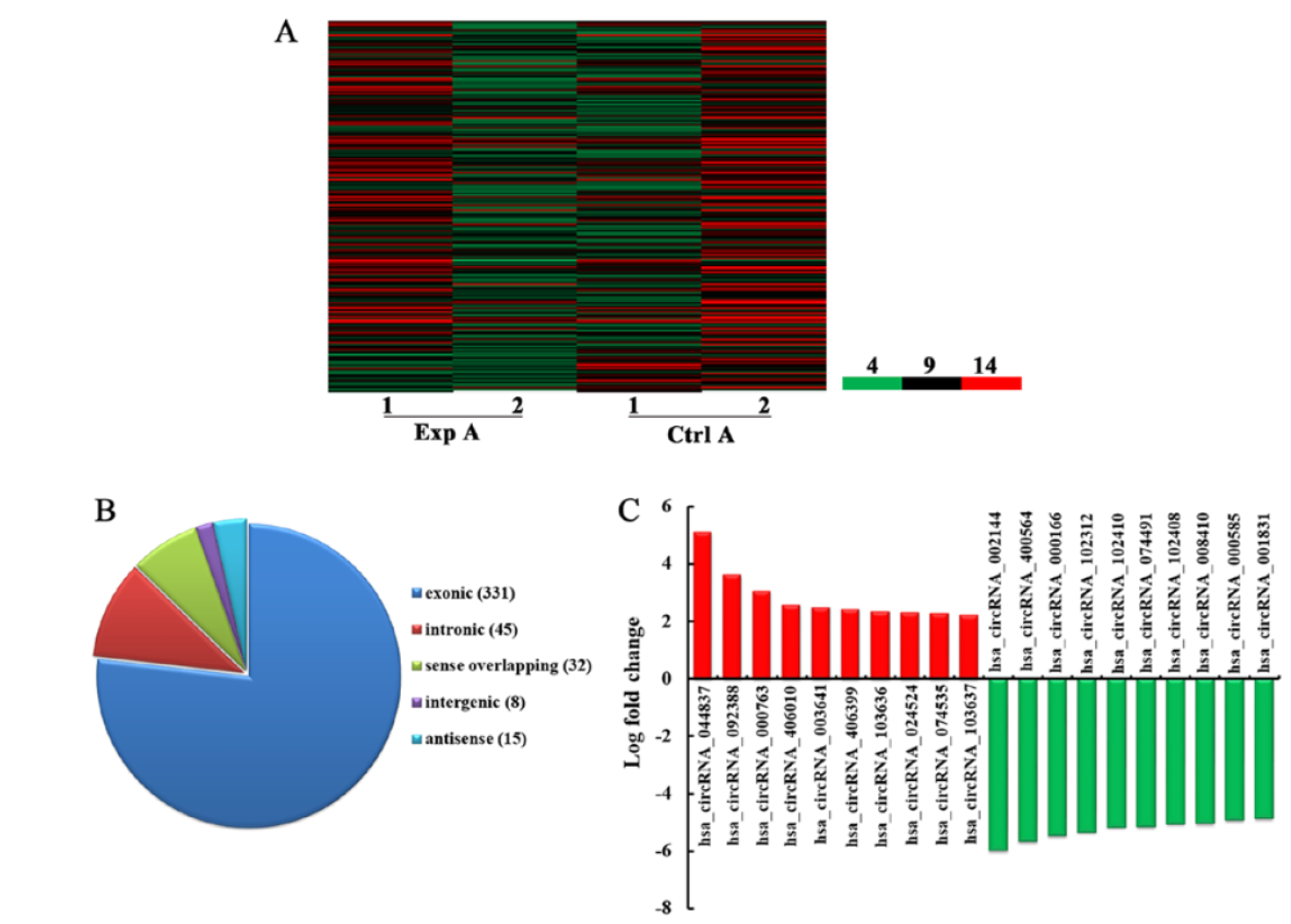
图6 肺转移结肠癌中circRNA表达差异 (来自[6])
7. circRNA参与小鼠IL-6通路调控
10月12日,Molecular Medicine Reports杂志(IF2016=1.692)在线发表了广州医科大学附属口腔医院郭吕华和杨岚为共同通讯作者的文章,介绍在小鼠巨噬细胞RAW264.7中发现Calcitonin相关多肽(CGRP)通过circRNA mmu_circRNA_007893影响IL-6通路[7]。

本文作者希望找出CGRP诱导IL-6表达的通路相关的circRNA分子,通过芯片法筛选,找到了该过程中差异表达的circRNA分子,其中表达增高的circRNA分子中就有mmu_circRNA_007893。后续经过实验验证和相互作用miRNA分子预测和验证,最终表明mmu_circRNA_007893在CGRP诱导IL-6表达的作用过程中发挥重要的调控作用。
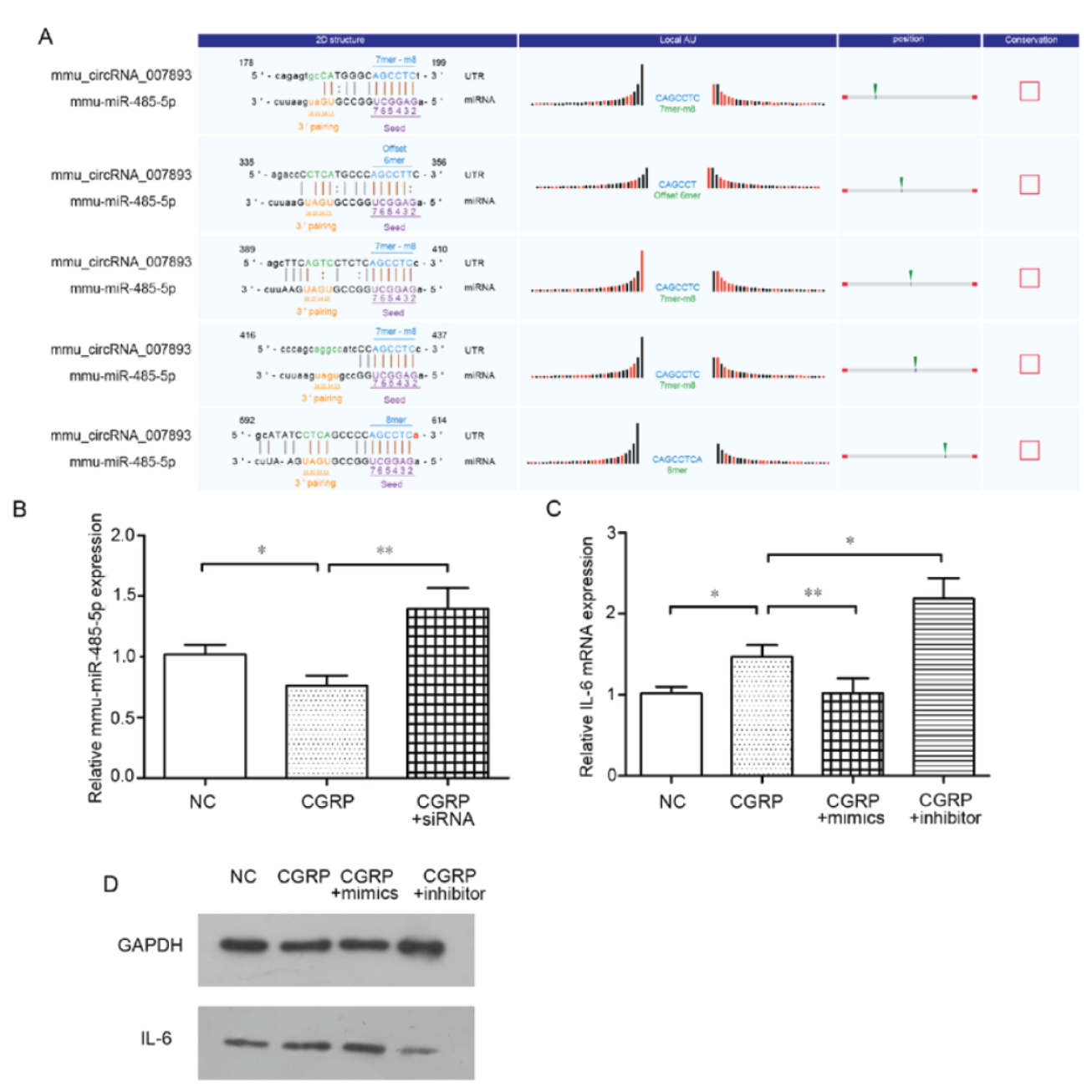
图7 mmu_circRNA_007893影响IL-6通路 (来自[7])
8. 畜牧学相关circRNA研究进展:
牛成肌细胞分化相关circRNA研究
10月26日,Cell Death & Disease杂志(IF2016=5.965)在线发表了西北农林科技大学陈宏教授为通讯作者的文章,介绍发现circLMO7参与调控牛的成肌细胞分化作用[8]。

作者挑选了胎牛和成年牛的肌肉组织进行RNA-Seq分析,共得到了12981种circRNA分子,其中还有530种为携带内含子的circRNA(ciRNA)。作者也针对所得到的circRNA分子进行了基因组定位,分子大小,包含的外显子情况等参数进行了汇总比较。相对于胎牛肌肉组织,circLMO7是下调最明显的circRNA分子之一。作者针对该分子做了一系列的生信分析和相关实验分析。
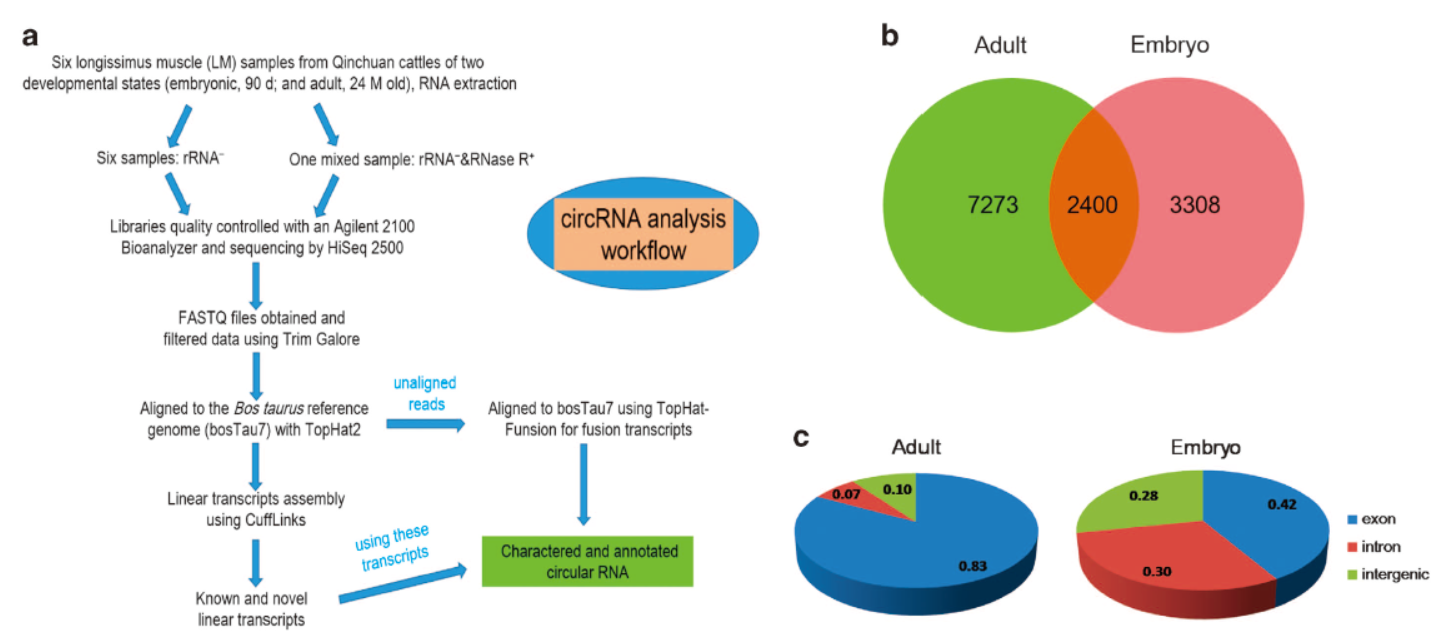
图8 牛成肌分化中circRNA变化情况 (来自[8])
circFUT10调控心肌细胞增殖
10月18日,Journal of Cellular Physiology杂志(IF2016=4.08)在线发表了西北农林科技大学陈宏教授为通讯作者的文章,介绍发现 CircFUT10通过竞争性结合miR-133a调控心肌细胞增殖和分化[9]。

本文的工作也是基于上述文章中的测序结果,分析了CircFUT10在调控心肌细胞增殖和分化过程中的作用,结果表明CircFUT10通过竞争性结合miR-133a发挥作用。
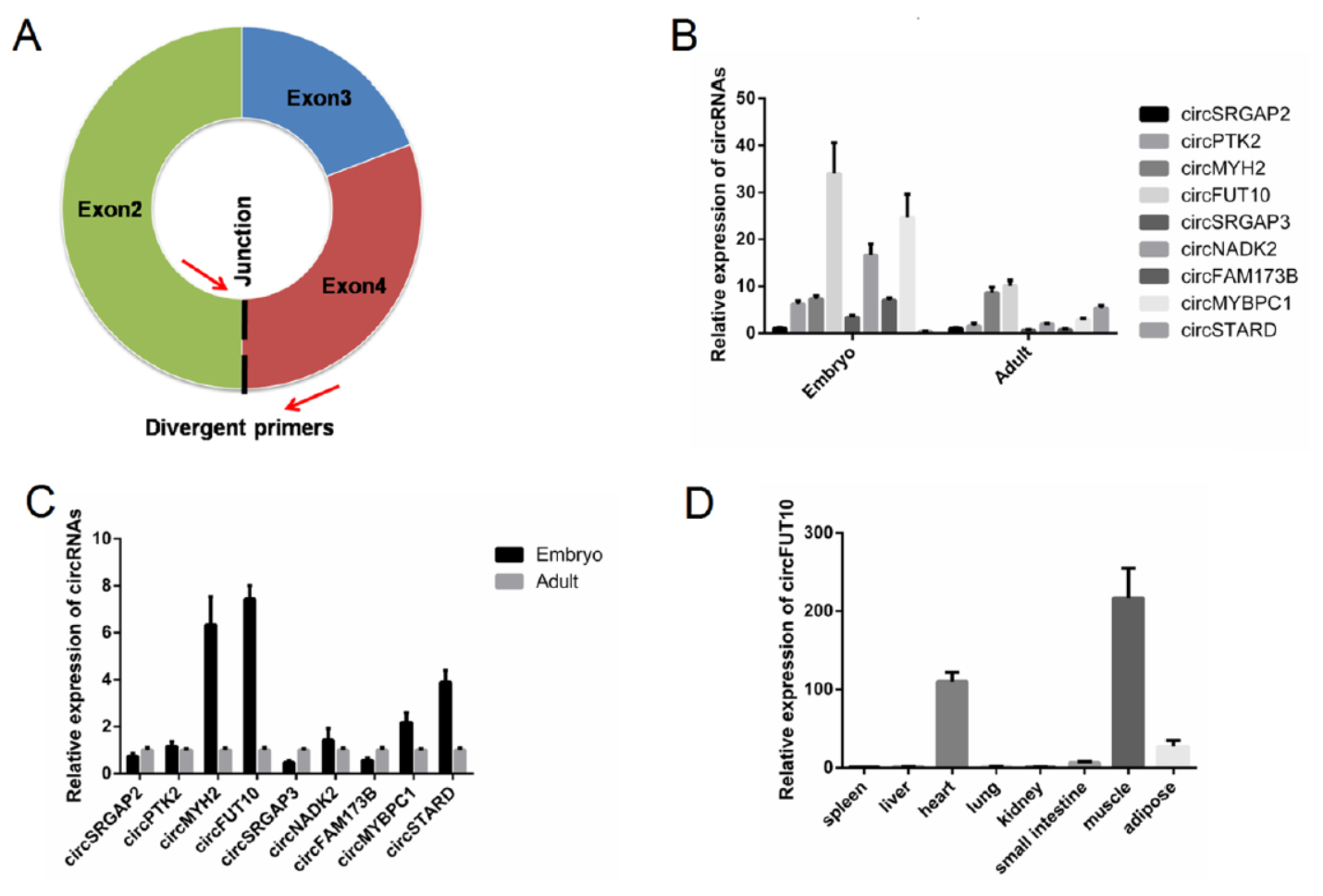
图9 CircFUT10调控心肌细胞增殖和分化 (来自[9])
circRNA在鸡肌肉发育中的作用
10月3日,DNA Research杂志(IF2016=5.404)在线发表了华南农业大学动物科学学院聂庆华为通讯作者的文章,介绍发现在鸡的肌肉发育过程中circRNA的动态表达变化[10]。

作者通过RNA-seq分析了不同发育阶段鸡胚的肌肉组织中circRNA表达变化情况,发现随着发育进程,circRNA的表达呈现一定的动态变化规律。
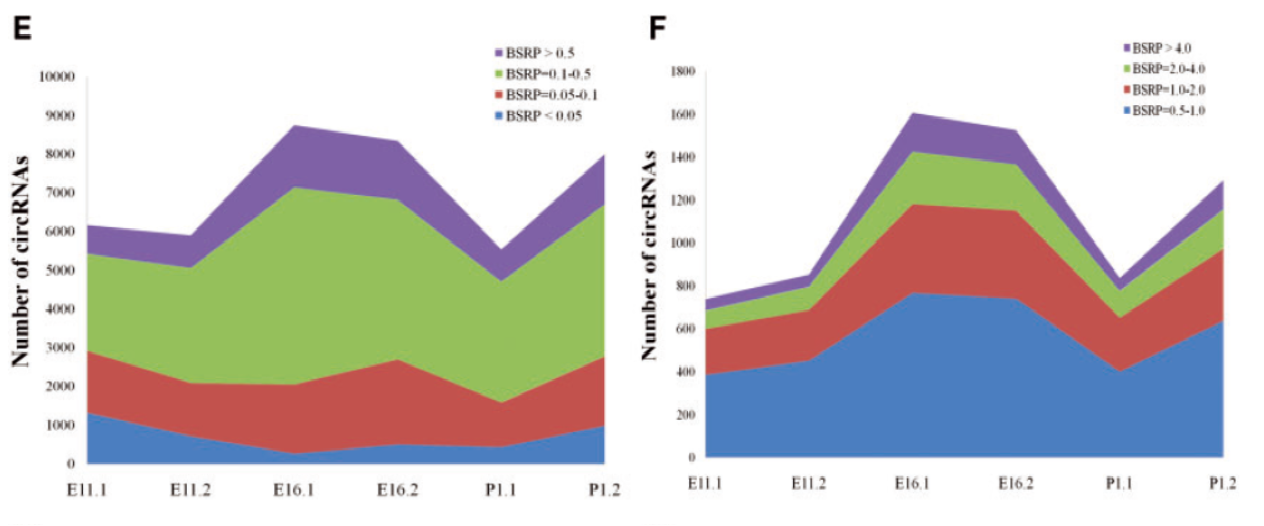
图10 鸡胚肌肉组织中circRNA随着发育状态呈现动态变化 (来自[10])
10. circRNA相关综述
circRNA-蛋白相互作用研究进展
9月26日,Theranostics杂志(IF2016=8.712)在线发表了加拿大多伦多大学杨柏华教授为通讯作者的综述文章,系统汇总了截至目前circRNA与蛋白相互作用的研究进展[11]。文中作者系统分析回顾了circRNA-蛋白相互作用的研究进展,主要研究技术及这种相互作用的生理意义。circRNA-蛋白相互作用会对彼此都产生一定的影响,circRNA的存在可以调控蛋白的定位等状态,蛋白与circRNA的结合也会影响circRNA的生成等过程。这初步说明circRNA-蛋白相互作用是非常重要的细胞调控机制,目前的研究还处于早期,非常值得深入探索。

circRNA作为临床诊断标志物的进展情况回顾
10月24日,Journal of Molecular Medicine(IF2016=4.686)杂志在线发表了慕尼黑技术大学Agnes Görlach1 和 Stefan Holdenrieder 共同撰写的综述文章,回顾总结了circRNA作为临床诊断标志物的进展情况[12]。

非编码RNA翻译的研究进展
10月12日,Experimental Cell Research杂志(IF2016=3.546)在线发表了安徽医科大学公共卫生学院叶冬青教授为通讯作者的综述文章,介绍目前关于非编码RNA翻译多肽的研究进展[13]。非编码RNA翻译多肽已经是众所周知的了,目前已报道的包括lncRNA,pri-miRNA和circRNA都可以翻译多肽。

circRNA研究依然在持续中,总体文章报道的数量也呈现稳步增长的趋势。相信随着研究的不断深入,会有更多有趣的新发现报道,让我们拭目以待吧。
参考文献:
1. Chun-Ying Yu, T.-C.L., Yi-Ying Wu, Chan-Hsien Yeh, Wei Chiang, Ching-Yu Chuang & Hung-Chih Kuo, The circular RNA circBIRC6 participates in the molecular circuitry controlling human pluripotency. Nature Communications, 2017.
2. He, R., et al., circGFRA1 and GFRA1 act as ceRNAs in triple negative breast cancer by regulating miR-34a. J Exp Clin Cancer Res, 2017. 36(1): p. 145.
3. Li, L., et al., Comprehensive analysis of circRNA expression profiles in humans by RAISE. Int J Oncol, 2017.
4. Yin, W.B., et al., Circulating circular RNA hsa_circ_0001785 acts as a diagnostic biomarker for breast cancer detection. Clin Chim Acta, 2017.
5. Chen, J., et al., Circular RNA WDR77 target FGF-2 to regulate vascular smooth muscle cells proliferation and migration by sponging miR-124. Biochem Biophys Res Commun, 2017.
6. Zeng, Y., et al., Altered expression profiles of circular RNA in colorectal cancer tissues from patients with lung metastasis. Int J Mol Med, 2017.
7. Deng, T., et al., Calcitonin generelated peptide induces IL6 expression in RAW264.7 macrophages mediated by mmu_circRNA_007893. Mol Med Rep, 2017.
8. Wei, X., et al., Circular RNA profiling reveals an abundant circLMO7 that regulates myoblasts differentiation and survival by sponging miR-378a-3p. Cell Death Dis, 2017. 8(10): p. e3153.
9. Li, H., et al., CircFUT10 reduces proliferation and facilitates differentiation of myoblasts by sponging miR-133a. J Cell Physiol, 2017.
10. Ouyang, H., et al., Circular RNAs are abundant and dynamically expressed during embryonic muscle development in chickens. DNA Res, 2017.
11. William W Du, C.Z., Weining Yang, Tianqiao Yong, Faryal Mehwish Awan, Burton B Yang, Identifying and Characterizing circRNA-Protein Interaction. Theranostics, 2017.
12. Gorlach, A. and S. Holdenrieder, Circular RNA maps paving the road to biomarker development? J Mol Med (Berl), 2017. 95(11): p. 1137-1141.
13. Li, L.J., et al., Translation of noncoding RNAs: Focus on lncRNAs, pri-miRNAs, and circRNAs. Exp Cell Res, 2017.
.png)