- 怎么提取和逆转录circRNA ?
常用细胞组织等样品提取total RNA,其中包含mRNA、lncRNA、miRNA和circRNA等所有的RNA种类,暂没有方法直接提取circRNA。但可以对total RNA进行RNase R消化处理,以使circRNA得到富集。
对total RNA或RNase R处理的RNA进行逆转录,应使用随机引物,不能使用oligo(dT),因为circRNA没有polyA尾。逆转录过程中circRNA模板是呈环形的,逆转录的cDNA第一链是线性的单链DNA,后续的PCR产物是线性的双链DNA。
- 高通量测序得到的circRNA怎么验证其真实存在?
高通量测序等方法筛选得到的circRNA一般要做进一步的鉴定和验证,涉及到的实验主要有PCR检测、circRNA全长鉴定和RNase R消化实验等。
2.1 PCR检测
对于circRNA的PCR检测,常分别使用Divergent Primer和Convergent Primer检测cDNA和gDNA模板,根据相应条带有无即可判断检测的序列是否为circRNA。另外Sanger测序确认Splice Junction序列仍是目前鉴定circRNA的“金标准”,因此PCR产物是必须要做Sanger测序的。
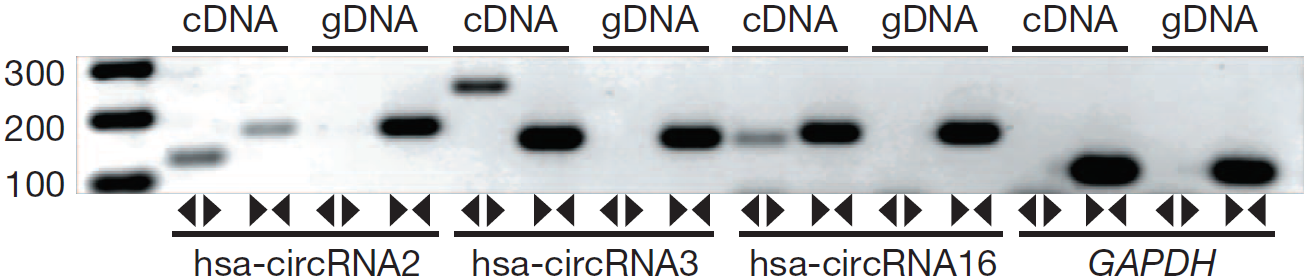
图1 分别使用Divergent Primer和Convergent Primer在cDNA和gDNA中检测hsa-circRNA2、hsa-circRNA2、hsa-circRNA2和GAPDH。在cDNA中以Divergent Primer能检测到对应大小的条带,以Convergent Primer检测到的条带大小不同;在gDNA中以Divergent Primer检测不到对应大小的条带,表明circRNA是反向成环的(Memczak S et al., 2013)。
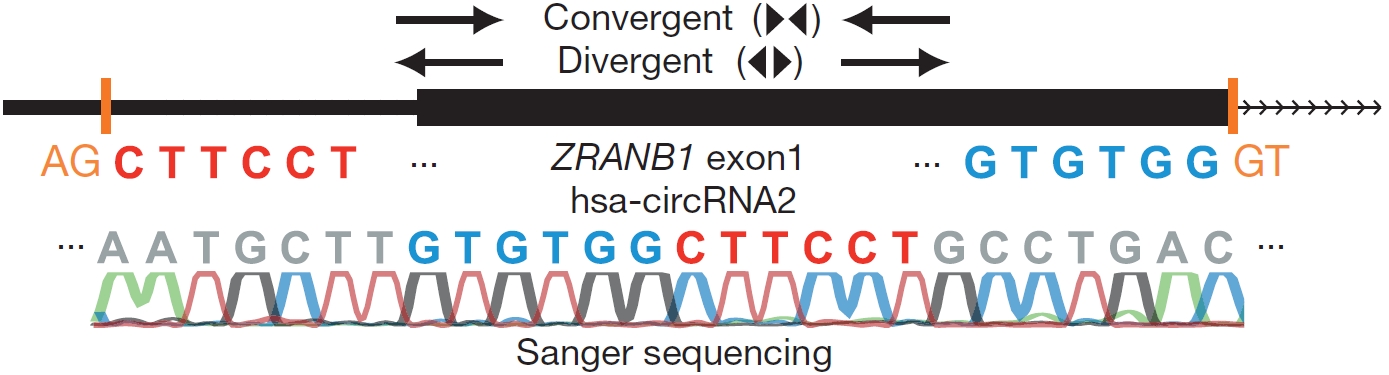
图2 对hsa-circRNA2的Divergent Primer扩增产物进行Sanger测序,鉴定验证的Splice Junction序列为反向成环(Memczak S et al., 2013)。
2.2 circRNA全长鉴定
目前circRNA参考序列信息多来自circBase中收集的高通量测序数据,高通量测序的局限在于:1)reads长度在150nt左右,不能测到circRNA的全长序列。2)后续mapping和Junction reads的筛选鉴定可能因为可变剪切而导致预测的circRNA spliced序列不准确。另外来自两条或更多pre-mRNA的剪切会因反式剪切(trans-splicing)形成tsRNA (trans-spliced RNA),这类tsRNA可能和circRNA有相同的Splice Junction序列,导致circRNA的筛选识别出现假阳性(Yu,C et al., 2014; Chuang TJ et al., 2018)。
因此对于circBase已收录或新发现的circRNA,都应进行全长鉴定以确定其真实的成环spliced序列。
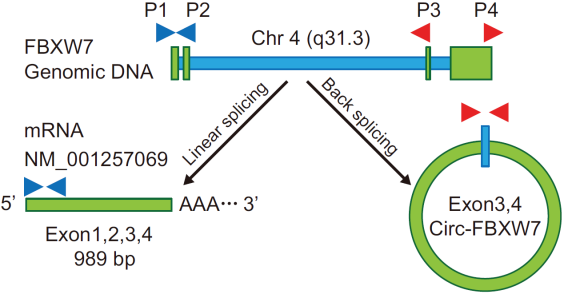
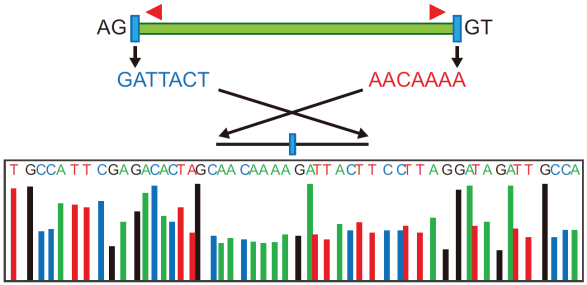
图3 通过PCR扩增和Sanger测序确定FBXW7的exon3和exon4经反向剪切形成620nt的circRNA,而不是circBase里收录hsa_circ_0001451的1227nt(Yibing Y et al., 2018)。
CircRNA全长鉴定的实验原理主要基于两点考虑:1)circRNA是反向剪切成环的,而线性RNA不成环,因此使用Divergent Primer进行反向PCR扩增,若为circRNA则有条带,若为线性RNA则无条带。2)逆转录过程中因模板置换在cDNA第一链中生成滚环产物,即在一条cDNA链中有多个Splice Junction相连接,此时两个Splice Junction中间即为circRNA spliced序列全长。
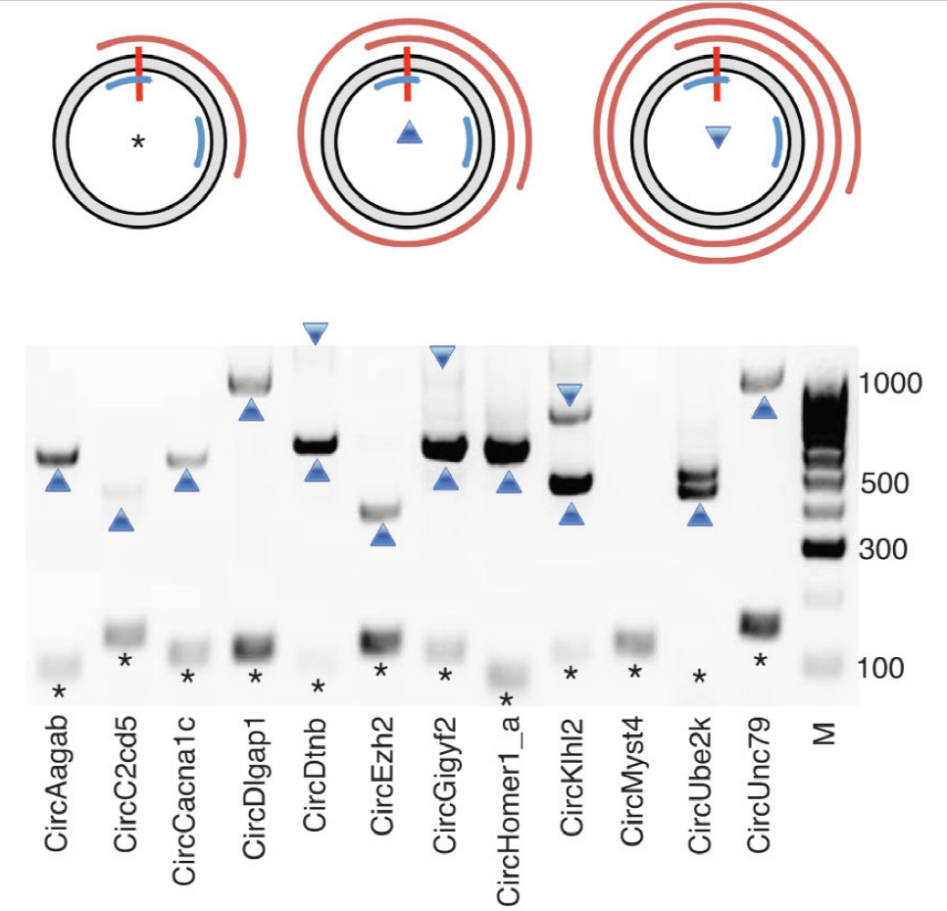
图4 上半部分星号代表未滚环的转录,蓝色向上箭头代表滚环+1圈的转录,蓝色向下箭头代表滚环+2圈的转录,红色曲线代表转录产生的cDNA。下半部分为电泳检测PCR产物条带,除circMyst4外都有滚环产物的条带(You X et al., 2015)。
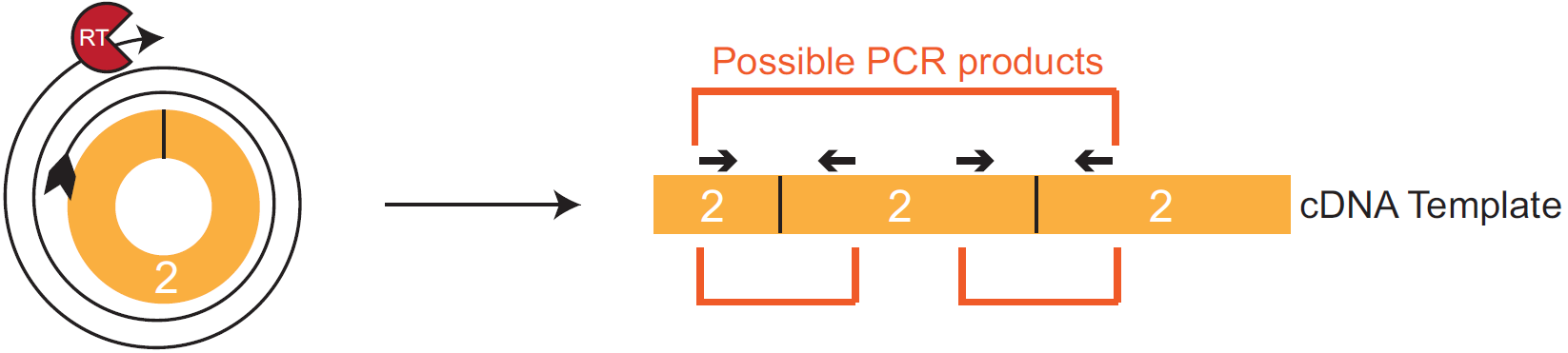
图5 以circRNA exon2示例滚环转录和cDNA模板,可能的PCR产物会跨越3个exon2的序列,序列内两个Splice Junction中间即为 circRNA exon2的全长(Barrett SP et al., 2016)。
CircRNA全长鉴定应使用Divergent Primer进行反向PCR扩增,PCR产物克隆至T载体后经Sanger测序以确定全长序列。需注意滚环转录产物在circRNA较短的时候更容易产生,若PCR检测无滚环转录的条带,可使用至少两对Divergent Primer分别进行反向PCR扩增,使扩增序列相互交叉,对比后即可拼接出circRNA的全长序列。
2.3 RNase R消化
和线性RNA不同,circRNA具有闭合环状结构,因此不易被核酸外切酶降解。现常用RNase R进行RNA的消化实验,以证明circRNA耐受消化。
RNase R消化实验一般为消化total RNA后分别检测线性RNA(mRNA)和circRNA,线性RNA丰度有明显降低而circRNA丰度基本不变,证明线性RNA被消化而circRNA耐受消化。依据实验目的不同,主要有PCR检测和Northern blot检测。
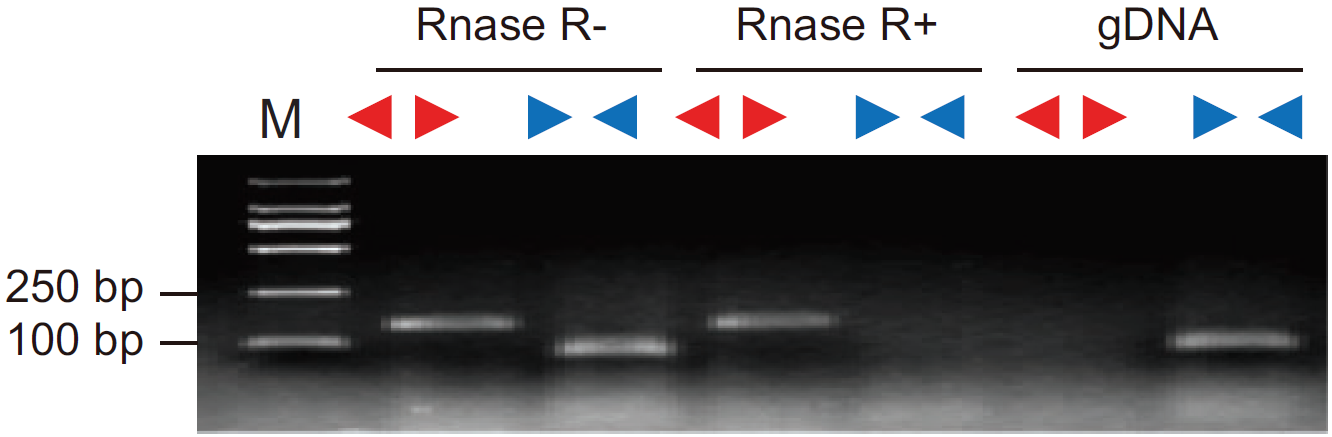
图6 RNase R消化后分别以Divergent Primer和Convergent Primer检测cDNA样品。RNase R+组中Divergent Primer有条带,Convergent Primer无条带;RNase R-组中Divergent Primer和Convergent Primer都有条带,表明检测的基因呈环形,且耐受RNase R消化(Yibing Y et al., 2018)。
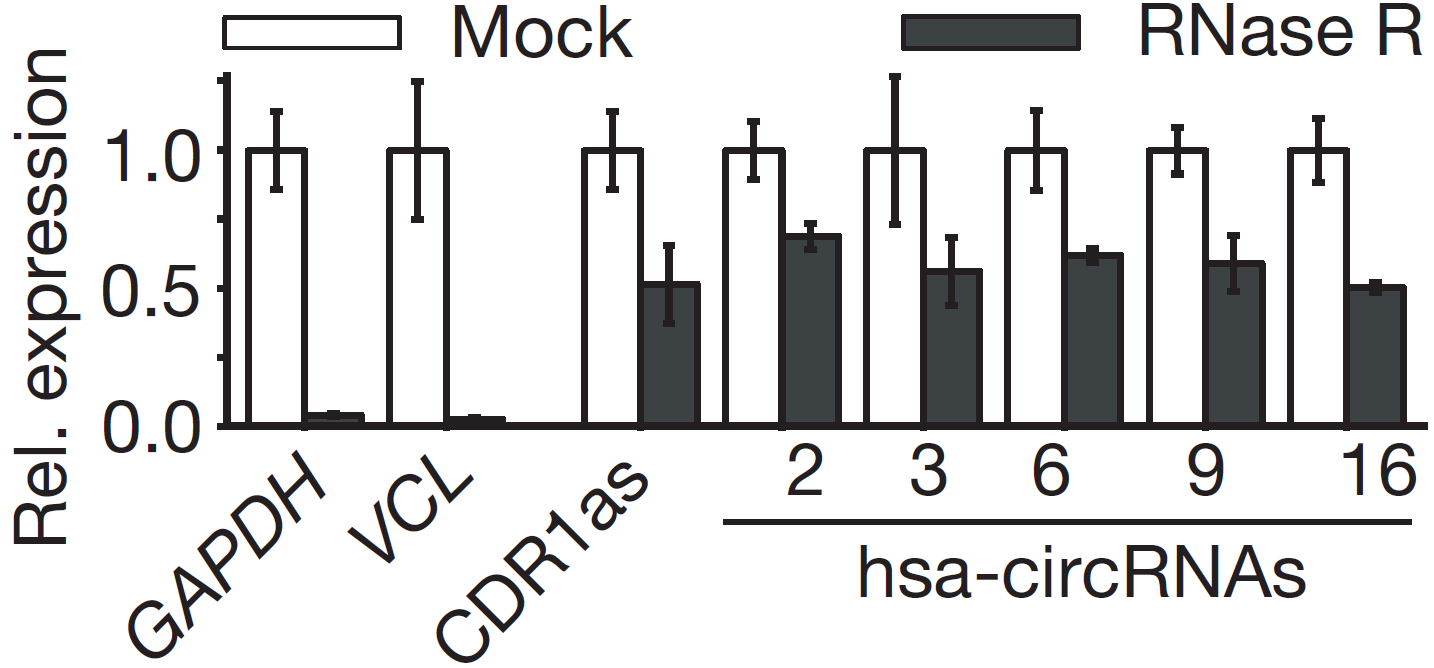
图7 RNase R消化后进行qPCR检测。RNase R组中线性的GAPDH和VCL相对表达量大幅降低,而hsa-circRNA 2/3/6/9/16和CDR1as则只有轻微降低,和GAPDH相比,circRNA显示有最少10倍的耐受力,表明circRNA比线性RNA耐受消化(Memczak S et al., 2013)。
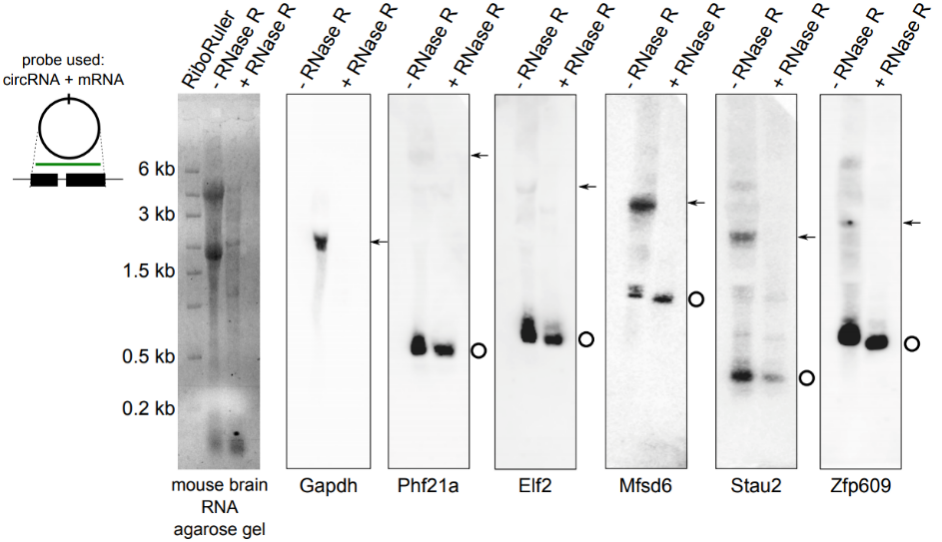
图8 将2 µg mouse total RNA进行RNase R消化,以3U/µg 于37℃孵育15分钟,加入10%线虫RNA作为外参后纯化回收,消化产物直接电泳显示RNase R+组中28S/18S条带变淡。使用同时检测circRNA和mRNA的探针进行Northern blot实验,结果显示RNase R+组检测不到线性的Gapdh、Phf21a、Elf2、Mfsd6、Stau2或Zfp609,可以检测到对应的circRNA,表明mRNA被消化,而circRNA耐受消化(Rybak-Wolf, A. et al., 2015)。
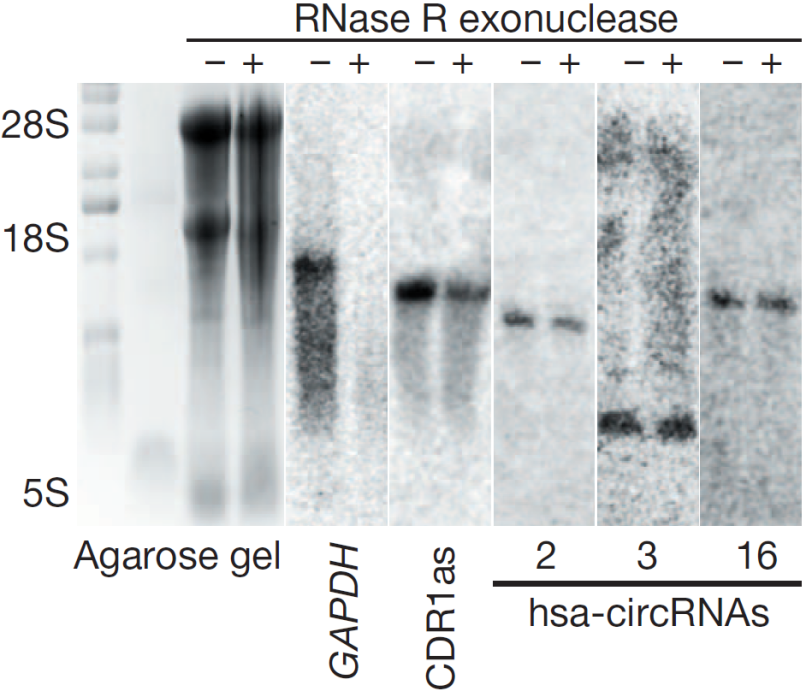
图9 RNase R消化产物直接电泳显示RNase R+组中28S/18S/5S条带变淡。Northern blot检测显示RNase R+组中无线性GAPDH的条带,而CDR1as和hsa-circRNA 2/3/16能检测到条带。表明线性RNA被消化,而circRNA耐受消化(Memczak S et al., 2013)。
另外,Northern blot可用探针特异性检测内源的circRNA和线性RNA,过程中无PCR扩增,能够真实地反映circRNA和线性RNA的定性定量分析结果,是circRNA鉴定中必不可少的一个实验。但因难度或实验条件限制,不少文章里都省略了Northern blot检测。
- circRNA过表达怎么做?
CircRNA的形成和线性RNA不同,因此对circRNA的过表达不能直接将序列连接到常规的真核表达载体(如pcDNA3.1)来进行。
已明确的circRNA形成依赖于侧翼序列中的反向互补序列(RCMs,如Alu元件)或与能调控circRNA生成的蛋白(如QKI)的结合位点,因此构建载体时可以在circRNA序列上下游分别增加一段反向互补的序列,质粒转染细胞后会先转录出上游+circRNA+下游的线性RNA链,再依靠长下游的反向互补配对促使成环。
具体实验中构建载体时可以在circRNA的侧翼各延伸1kb(一般会包含Alu或重复序列),使用真核表达载体构建一个上游侧翼1kb+circRNA+下游侧翼1kb的载体,一般都可以成功地过表达构建的circRNA。另外也可以使用商业化有通用成环框架的circRNA表达载体(如pLCDH-ciR)来构建,只需要连接circRNA序列进入载体即可。
- circRNA过表达成功标准?
载体构建好后瞬时转染细胞进行RT-PCR检测以验证过表达效率。1)qPCR检测有过表达倍数,质粒瞬转效率高,基因本底丰度不太高的话一般都可以过表达50倍以上;2)使用Divergent引物检测过表达的PCR产物是大小正确的单一条带,PCR产物进行sanger测序确定成环序列准确。满足这两个条件才可以认为载体实现了对circRNA的准确高效率过表达。
- 怎么查找circRNA侧翼的Alu元件?
Alu元件在RepeatMasker中有注释,在UCSC Genome Browser可以根据基因组位置查找circRNA侧翼的Alu元件。
图为hsa_circ_0006404在基因组上的位置,红色箭头处可以手动输入以调整上下游延伸的长度,绿色箭头指示SINE行中即为可能存在的Alu元件(或其他重复元件),点击灰框进去可以查看详细的注释信息和序列。
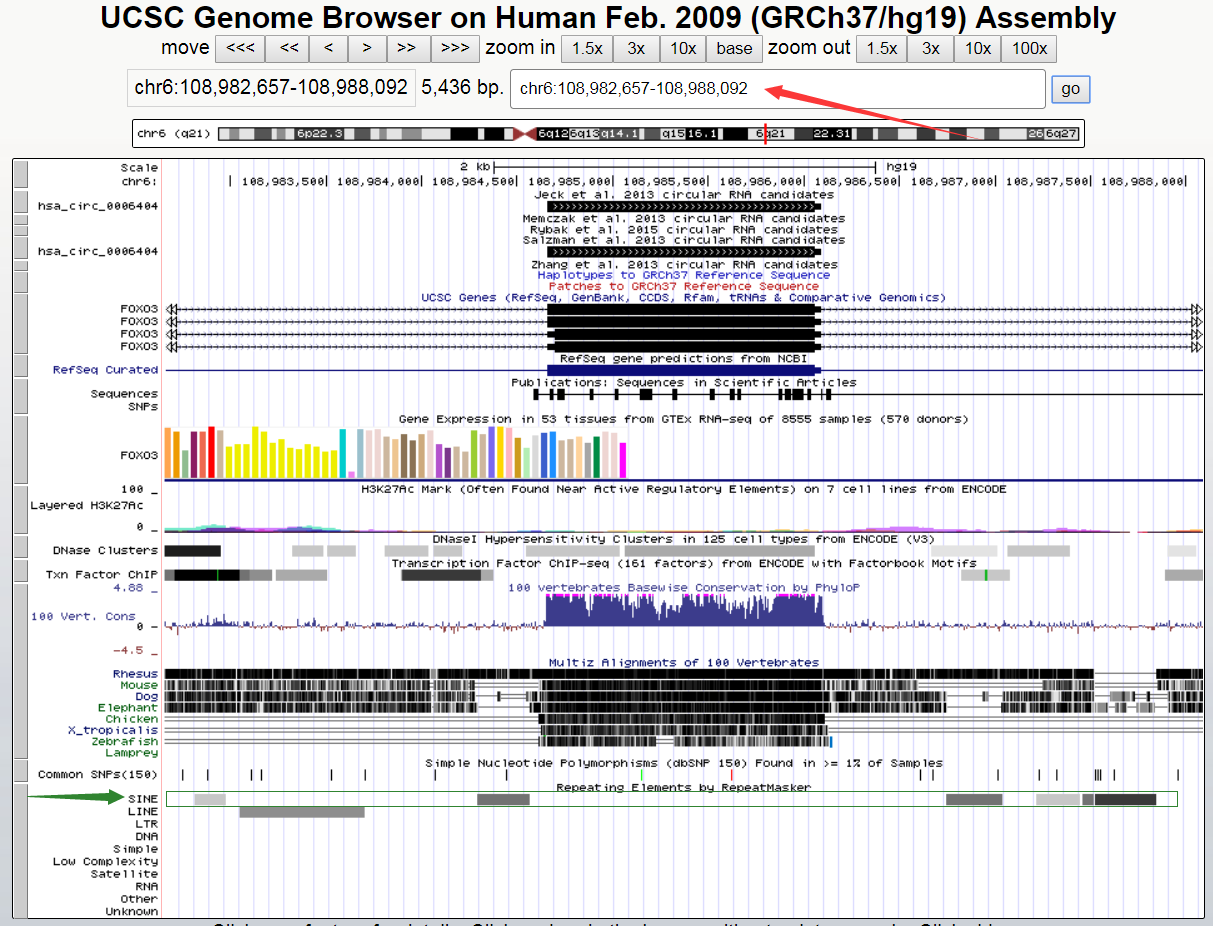
图10 UCSC Genome Browser
- circRNA 的沉默(干扰)怎么做?
基于RNAi技术对基因进行沉默,实验中一般设计合成靶标基因的siRNA,瞬转细胞后进行RT-PCR检测。
外显子来源的circRNA因为和mRNA有同样的外显子序列,在序列内部设计siRNA靶标无法保证特异性,因此只能针对junction位点处设计靶标序列,前后移动范围有限,siRNA通常很难设计。
EiciRNA或者内含子来源的环形RNA,除了针对junction位点处设计靶标外,也可以尝试在内含子序列上设计。
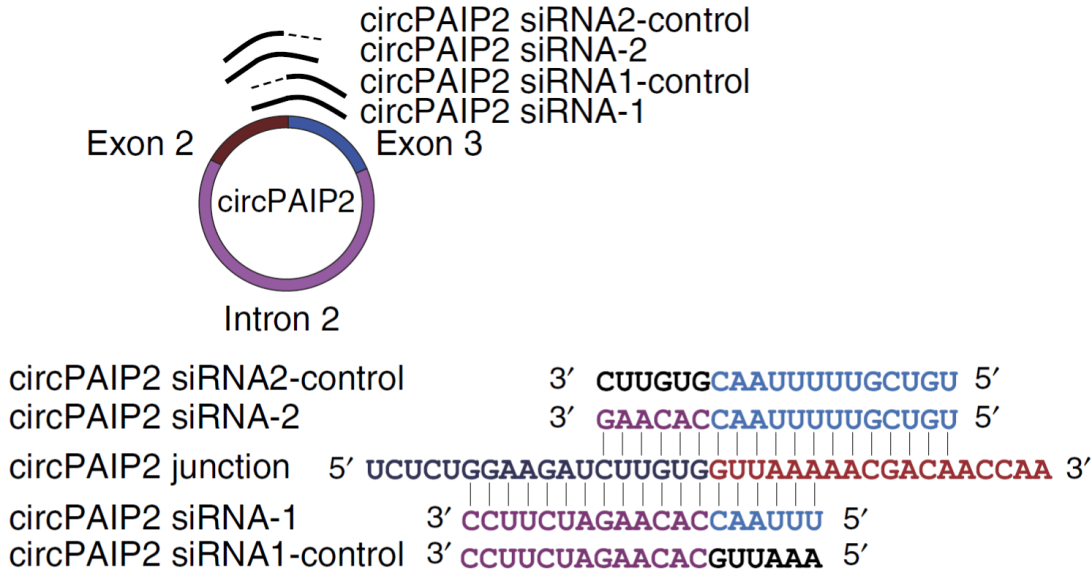
图11 circRNA的siRNA设计示意图(Li Z et al., 2015)
- CRISPR/Cas9可以用来做circRNA敲除吗?
CRISPR/Cas9利用gRNA引导靶标DNA来实现对特定位置的切割,能快速高效地对基因进行敲除或编辑。
以基因组DNA上特定位置为模板转录可能同时形成mRNA或circRNA等,若以CRISPR/Cas9对基因组DNA特定位置进行敲除,除了敲除circRNA外,也可能造成mRNA被敲除,因此直接用CRISPR/Cas9敲除circRNA而不影响对应的mRNA几乎无法实现。另外考虑circRNA侧翼的Alu元件,敲除位置(gRNA靶标位置)的设计选择也有很大局限性。只有几种特殊的情况可以用CRISPR/Cas9来敲除circRNA。
一种情况是直接敲除circRNA的成熟序列来源。例如来源于X染色体的Cdr1as表达不是性别特异的,也检测不到同一位置来源的其他RNA表达,因此可以直接敲除Cdr1as的成熟序列对应的gDNA区域。
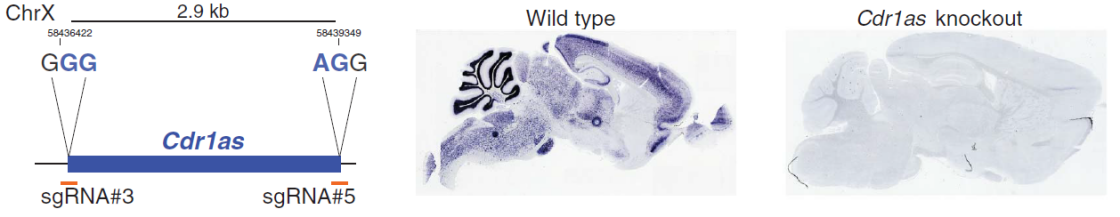
图12 小鼠Cdr1as敲除(M Piwecka et al., 2017)
一种情况是敲除circRNA侧翼的Alu元件。例如circHIPK3有特别长的flanking introns,敲除侧翼的Alu元件后不影响对应的mRNA表达,这种敲除也是成功的。但大多数circRNA侧翼的Alu元件都不止一个,靶标敲除哪一个Alu,会不会影响mRNA或其他线性RNA的表达,在实验设计时都很难确定。
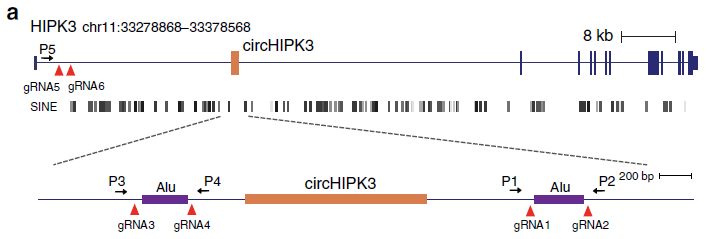
图13 敲除circHIPK3侧翼的Alu元件(Zheng Q et al., 2016)
另外一种策略,靶标同一段DNA,使circRNA和对应的mRNA都被敲除,然后再恢复mRNA,间接实现敲除circRNA而不影响对应的mRNA。
- 怎么验证circRNA的miRNA sponge功能?
CircRNA作为miRNA的sponge,其核心依据是circRNA的序列可以和miRNA结合,可以利用体外的双荧光素酶实验来验证。一般将circRNA的序列连接到psiCHECK2载体上,将载体和miRNA mimics同时转染细胞,之后检测荧光素酶活性可以判断circRNA序列和miRNA的结合情况。
另外也可以进行FISH实验,分别以荧光探针标记circRNA和miRNA,若circRNA和miRNA有共定位的情况,推测circRNA和miRNA可能有结合。
- circRNA为什么要做RNase R消化?
RNase R是一种核糖酸外切酶,可以消化几乎所有的线性RNA分子,但不易消化环形RNA、套索结构或3’突出末端少于7个核苷酸的双链RNA分子。
CircRNA具有闭合环状结构,因此不易被核酸外切酶降解,使用RNase R消化RNA,可以用来证明circRNA耐受消化,也可以对circRNA进行富集。RNase R消化是鉴定和验证circRNA必不可少的一个实验。
- 怎么知道一个circRNA是否可以翻译蛋白?
CircRNA翻译蛋白是一个新的热门研究方向,但受限于分析和验证方法及其难度,目前仅有不到10篇文章报道。
考虑感兴趣的circRNA是否能翻译蛋白,可先以ORF Finder分析序列基础,预测是否有合适的ORF。有一个跨越junction的ORF是最理想的,序列内部不跨越junction的ORF可能和mRNA一样,没办法特异研究circRNA。也可以参考circRNADb数据库,已收录的circRNA都有相应的ORF注释信息。
有了序列基础后也要考虑circRNA翻译的机制,如考虑IRES介导可以在circRNADb参考相应的预测信息,或者使用其他工具预测IRES元件,也可以考虑m6A介导的翻译。
预测ORF有了序列基础,再分析IRES和m6A或其他机制介导翻译,下一步就可以进行实验验证了。实验可以分为两部分,一是用特异性的抗体或引入标签抗体进行检测,验证是否能检测到预测的蛋白(多肽),二是验证IRES或m6A介导的翻译机制。
参考文献:
- Memczak S, Jens M, Elefsinioti A, et al.. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature. 2013;495(7441):333–338.
- Yu,C.Y., Liu,H.J., Hung,L.Y., Kuo,H.C. and Chuang,T.J.. (2014) Is an observed non-co-linear RNA product spliced in trans, in cis or just in vitro? Nucleic Acids Res., 42, 9410–9423.
- Chuang TJ, Chen YJ, Chen CY, Mai TL, Wang YD, Yeh CS et al.. Integrative transcriptome sequencing reveals extensive alternative trans-splicing and cis-backsplicing in human cells. Nucleic Acids Res. 2018 Apr 20;46(7):3671-3691.
- You X, Vlatkovic I, Babic A, Will T, Epstein I, Tushev G, et al.. Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity. Nat Neurosci (2015) 18(4):603–10.10.1038/nn.3975.
- Barrett SP, Salzman J. Circular RNAs: analysis, expression and potential functions. Development (Cambridge, England) 2016;143:1838–47.
- Yibing Yang et al.. Novel Role of FBXW7 Circular RNA in Repressing Glioma Tumorigenesis. J Natl Cancer Inst. 2018 Mar 1;110(3).
- Rybak-Wolf, A. et al.. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed. Mol. Cell. 58, 870–885 (2015).
- Li Z, Huang C, Bao C et al.. Exon-intron circular RNAs regulate transcription in the nucleus. Nat Struct Mol Biol. 2015 Mar;22(3):256-64.
- M Piwecka et al.. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function. Science. 2017 Sep 22;357(6357).
- Zheng Q, Bao C, Guo W et al.. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nat Commun. 2016 Apr 6;7:11215.
.png)