刚刚结束的三月是一个不平凡的月份,集中出现了多篇重磅级的环状RNA研究论文,包括Cell Research和Molecular Cell杂志接连发表了三篇高水平的研究论文介绍证明环状RNA可翻译蛋白;一篇Nature文章介绍Alu元件在基因表达调控方面的机制,涉及到了环状RNA;一篇Current Biology文章介绍果蝇母系遗传的内含子来源环状RNA参与调控发育活动;一篇Nature Communication文章介绍RNA结合蛋白FUS与神经元环状RNA的形成有关;一篇Cancer Research文章介绍环状RNA与肿瘤的增殖和转移。下面山人就和大家一起回顾一下三月份的这些重要研究进展吧:
1. 环状RNA存在m6A修饰,可翻译蛋白
环状RNA可能翻译蛋白不是最新才提出的,之前已有体外实验和零星的报道,但在高等模式生物中一直还没有非常有说服力的论文发表。就在3月份里,连续出现了三篇文章介绍发现了环状RNA可翻译蛋白的现象。先是3月10日,Cell Research杂志在线发表了中科院上海计算生物学研究所王泽峰教授为通讯作者的研究论文,介绍发现了环状RNA中m6A修饰,并且该修饰能促进环状RNA翻译[1]。接着是3月15日和16日,Molecular Cell杂志连续发表了两篇文章,介绍发现环状RNA可翻译蛋白。
环状RNA存在m6A修饰,并且该修饰能促进环状RNA翻译:

在本中王教授给出的m6A修饰调控环状RNA翻译的机制是:由METTL3/METTL14-WTAP丹巴复合物进行m6A修饰,FTO进行m6A去修饰调控。含有m6A修饰的位点通过募集YTHDF3进而募集eIF4G2,进而启动蛋白翻译过程[1]。该论文具有里程碑式的重要意义,首先证明了环状RNA可进行翻译活动,其次,还是首次报道环状RNA存在m6A修饰,填补了环状RNA化学修饰方式研究的空白。
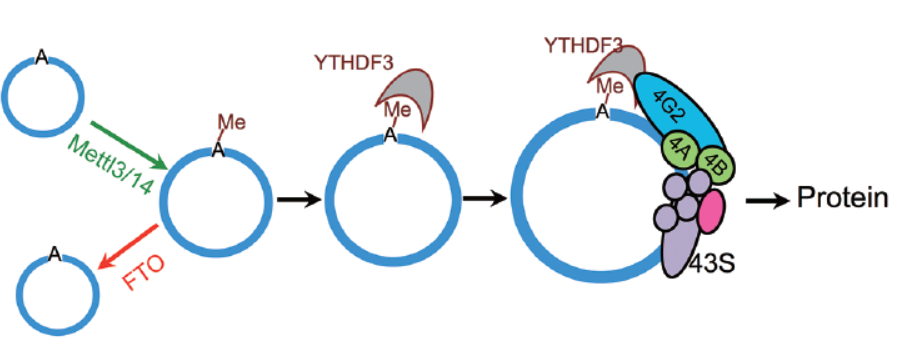
图1 m6A修饰调控环状RNA翻译(来自[1])
环状RNA存在m6A修饰:
也是在3月10日,国际著名预印本杂志BioRxiv在线发表了哈佛大学医学院Cosmas C. Giallourakis和Alan C. Mullen为共同通讯作者的论文,介绍发现了环状RNA也存在m6A修饰[2]。

文中作者介绍发现了RNase R消化后的样品中依然存在m6A修饰的RNA,因此怀疑环状RNA存在该修饰方式。在人的胚胎干细胞和Hela细胞中分别分析带m6A修饰的环状RNA表明环状RNA的m6A修饰具有细胞类型特异性的特征。METTL3和METTL14是环状RNA的m6A修饰酶,YTHDF1 和YTHDF2是环状RNA的m6A修饰的读取酶。有趣的是,作者还发现环状RNA的m6A修饰位点在线性的mRNA中往往不被修饰,并且与环状RNA在同一位点进行m6A修饰的线性mRNA往往不稳定。这种环状RNA-mRNA互作方式是由YTHDF2介导的[2]。由此,作者提出了环状RNA的表观组学概念。这篇文章虽然还不是正式经过同行评议后正式发表的论文(预印本就是专门提供快速发表方式的,不经过同行评议),但本文的故事对于研究环状RNA的m6A修饰以及环状RNA-mRNA互作问题都具有非常重要的借鉴意义。
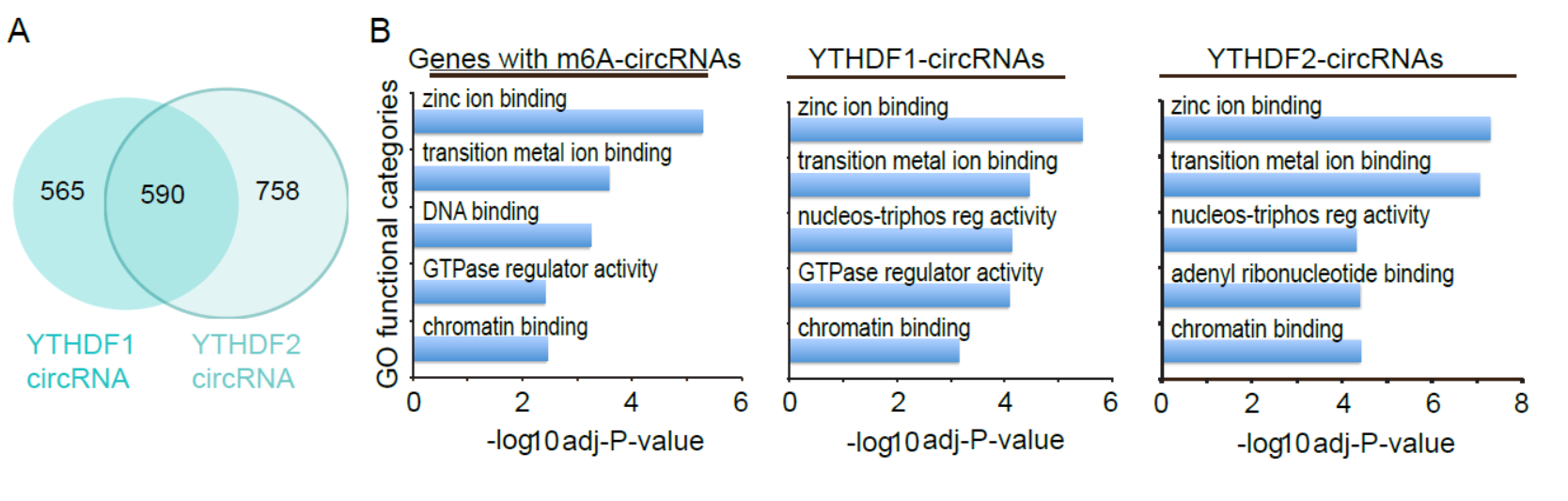
图2 具有m6A修饰的环状RNA(来自[2])
环状RNA翻译蛋白:
3月15日,Molecular Cell在线发表了以色列希伯来大学的Sebastian Kadener教授,介绍基于果蝇大脑中核糖体印迹分析(ribosome footprinting)发现大量的环状RNA翻译蛋白或多肽的情况[3]。

作者在果蝇的大脑组织中进行核糖体印迹分析,发现了有些环状RNA可以结合到核糖体上,于是作者认为环状RNA有可能具有翻译蛋白的功能。经过一系列的严谨实验论证,作者得出结论:部分环状RNA可翻译蛋白。核糖体印迹分析中证明circMbl中的终止密码子处有核糖体结合。circMbl所编码的蛋白在蛋白质谱中得到证据[3]。
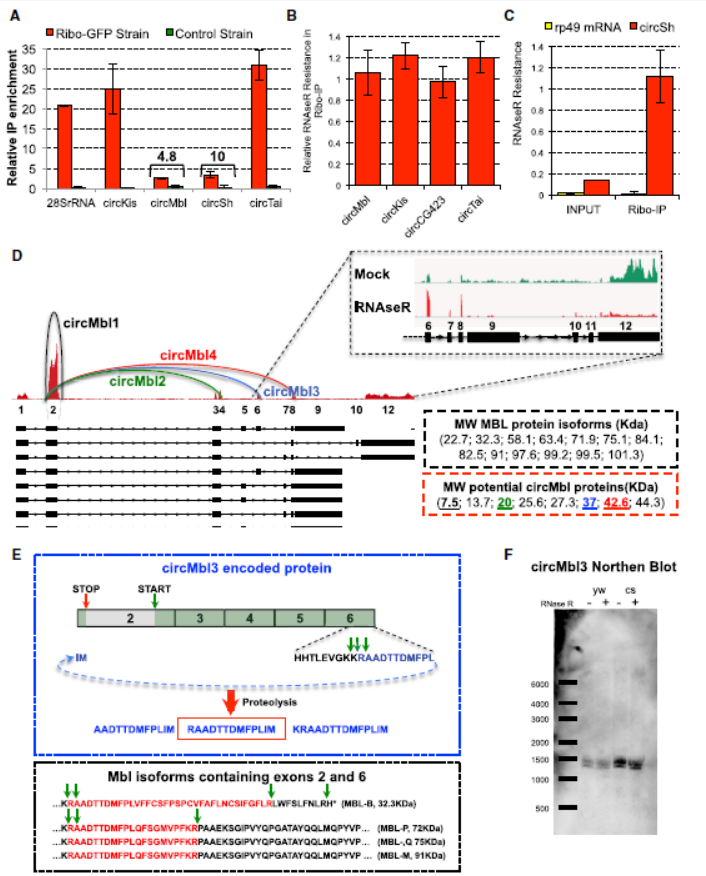
图3 果蝇CircMbl可翻译蛋白(来自[3])
Circ-ZNF609可直接翻译蛋白并参与肌肉发生过程:
3月16日,Molecular Cell杂志在线发表了意大利罗马萨皮安扎大学Irene Bozzoni教授,介绍发现了环状RNA Circ-ZNF609可直接翻译蛋白,该蛋白参与肌肉发生过程[4]。

文中作者首先筛选与肌肉发生有关的环状RNA,人和小鼠中都进行了筛选,还选择了杜氏肌营养不良疾病模型一并进行筛选。最终筛选到一批特征性的环状RNA,接着,作者又针对这些环状RNA进行了RNAi文库筛选,以找出对肌肉发生功能有关的环状RNA。基于这一系列的筛选,作者最终发现了Circ-ZNF609。接下来针对Circ-ZNF609的功能研究作者验证了其翻译多肽的功能,并针对该功能进行了一系列的生化验证实验[4]。
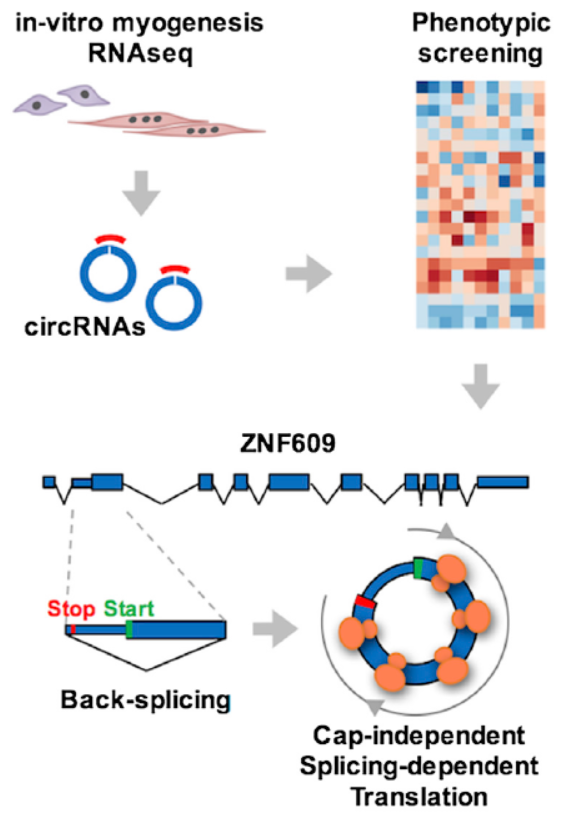
图4 Circ-ZNF609可直接翻译蛋白并参与肌肉发生过程(来自[4])
2. RNA解旋酶DHX9特异性结合反向Alu元件调控RNA加工和环状RNA形成
3月29日,Nature杂志在线发表了德国马普学会免疫生物学和表观遗传学研究所Asifa Akhtar教授为通讯作者的Letter文章,介绍发现反向Alu元件通过RNA解旋酶DHX9调控RNA转录后加工过程。DHX9缺失会导致环状RNA增多,为反向Alu元件调控RNA转录产物加工过程提供了全新的机制模型[5]。

人类基因组含有大量的转座性元件,其中包括以Alu元件为代表的短散在序列(SINE),约占到了人类基因组的10%左右的体量。在这篇文章中,作者发现RNA解旋酶DHX9特异性识别反向重复Alu元件。DHX9缺失会导致RNA转录后剪接异常,环状RNA增多[5]。
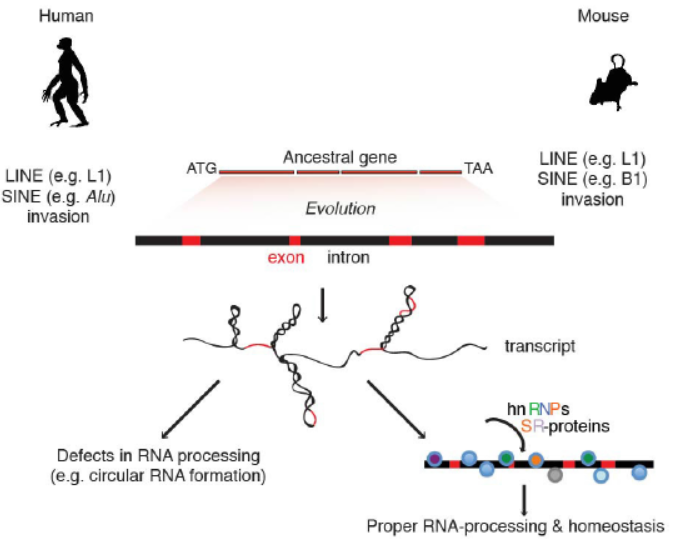
图5 DHX9特异性结合反向互补Alu元件调控RNA加工与环状RNA形成(来自[5])
3. 果蝇母系遗传内含子来源环状RNA调节发育进程
3月17日,Cell子刊Current Biology在线发表了新加坡国立大学Jun Wei Pek教授为通讯作者的文章,介绍发现母系遗传的内含子来源环状RNA sisR-4调控宿主基因的表达并影响发育过程[6]。

文中作者一开始希望找出果蝇发育过程中母系遗传的内含子来源的环状RNA,经过RNase R处理后测序,找到了5个环状RNA,再结合遗传特性,确定CG9775 和deadpan基因内含子来源的环状RNA符合要求。作者在本文中重点探讨了来源于deadpan基因内含子的环状RNA sisR-4在胚胎发育过程中的作用以及与宿主基因deadpan的关系。最终结果表明sisR-4可以母系遗传的方式通过提高宿主基因的表达在发育过程中发挥作用[6]。
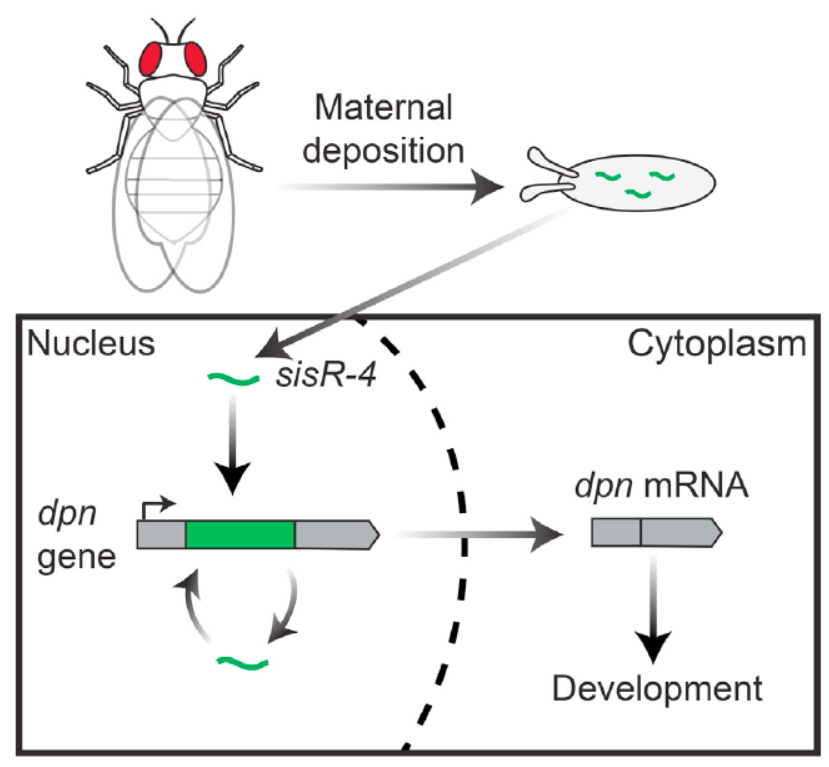
图7 内含子来源的环状RNA sisR-4调控宿主基因表达和发育过程(来自[6])
4. RNA结合蛋白FUS调控神经元环状RNA形成
3月30日,Nature Communication杂志在线发表了罗马萨皮安扎大学Mariangela Morlando教授和意大利技术研究所Irene Bozzoni教授为共同通讯作者的文章,介绍发现RNA结合蛋白FUS在神经元中介导RNA反向拼接活动,与环状RNA的形成有关[7]。

文中作者首先比较了FUS基因敲除前后自ES分化的运动神经元中环状RNA的表达状况,发现FUS敲除后有一批环状RNA的表达明显降低。接着基于RNA干扰和过表达ALS疾病相关的FUS基因后环状RNA的表达变化情况,最终明确了FUS蛋白在神经元中调控了RNA反向拼接的作用,与环状RNA的形成有关[7]。
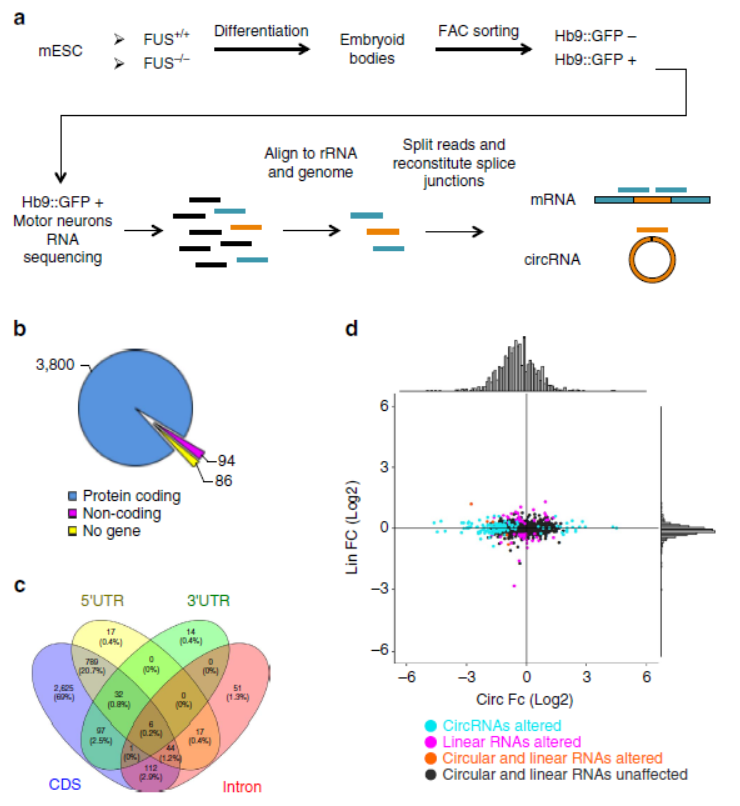
图6 FUS敲除对运动神经元环状RNA的影响(来自[7])
5. 环状RNA CCDC66促进结肠癌增殖和转移
3月1日,Cancer Research杂志在线发表了台湾国立成功大学Shaw-Jenq Tsai博士和H. Sunny Sun博士为共同通讯作者的文章,介绍发现环状RNA CCDC66促进结肠癌增殖和转移[8]。

文章作者首先通过测序筛选结肠癌相关的环状RNA,作者挑选了circCCDC66进行深入探索分析。发现circCCDC66能促进结肠癌细胞增殖和迁移。在结肠癌中circCCDC66过表达,并与结肠癌的临床症状表型有相关性[8]。
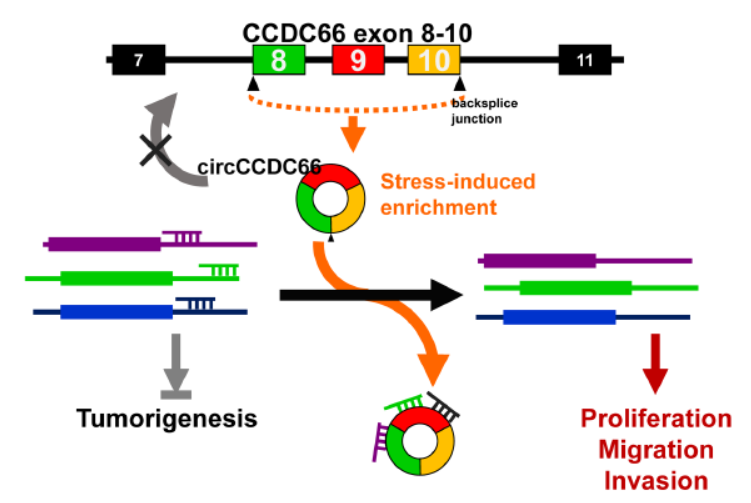
图8 circCCDC66促进结肠癌增殖和转移(来自[8])
6. 环状RNA信息学研究方法技术进展
3月11日, 著名预印本杂志BioRxiv在线发表了中山大学屈良鹄教授和杨建华教授为共同通讯作者的文章。介绍利用circScan分析环状RNA相互作用蛋白的技术[9]。

作者开发了一种全新的分析环状RNA相互作用蛋白的技术体系,称为circScan,主要是基于交联技术将互作蛋白钓取的RNA进行深度测序(CLIP-Seq),找出包含反向拼接的Reads。利用该技术流程,作者在人的样品中发现了12540种环状RNA-蛋白相互作用,小鼠中发现了1090种。有趣的是,作者发现了一些环状RNA与eIF3相互作用,这是一种5’-帽子不依赖的蛋白翻译起始因子,也发现了环状RNA具有m6A修饰的情况[9]。这与其他文献报道的发现吻合。
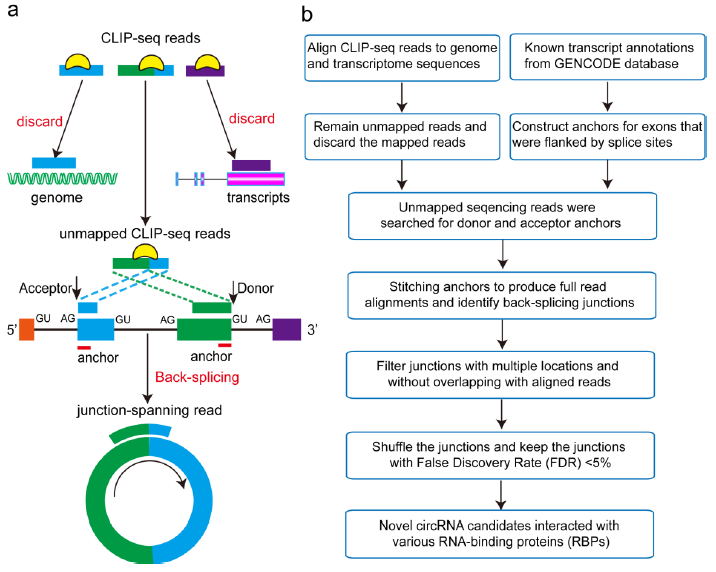
图9 circScan流程分析蛋白-环状RNA相互作用(来自[9])
7. 环状RNA信息学研究方法技术进展
3月份有多篇环状RNA数据和信息分析相关的研究文章发表,包括中科院基因组研究所赵方庆教授发表在Brief Bioinform的文章,提出了全新的多重种子匹配算法及最大似然估计模型[10]。3月8日,Bioinformatics在线发表了内华达州医学院Zhou Tong和 东南大学Gu Wanjun为共同通讯作者的文章,介绍开发了一种新的从RNA测序数据中分析环状RNA定量的工具[11]。3月17日,Methods杂志在线发表了韩国中安大学Hyeyoung Min为通讯作者的文章,介绍一种基于机器学习的miRNA Sponge分析方法[12]。
8. 其他环状RNA研究论文和综述
传统的环状RNA研究思路包括基于二代测序进行差异表达环状RNA的筛选鉴定,然后进行定量PCR验证。分析一下候选的特征性环状RNA的结构,基因组中对应序列特征信息,miRNA sponge预测分析和通路分析等等。3月份环状RNA研究的论文体量较大,以传统的研究思路进行的研究论文就不再详述了,列表如下:
| 通讯作者 | 文章题目 | 杂志 | 概要 |
| 浙江大学第二附属医院眼科Ke Yao | Transcriptome-wide Investigation ofmRNA/circRNA in miR-184 and Its r.57c > u Mutant Type Treatment of Human Lens Epithelial Cells | Molecular Therapy: Nucleic Acids | 人类晶状体上皮细胞中miR-184及其r.57c> u突变体中mRNA / circRNA的转录组全面研究 |
| 山东大学齐鲁医院Daoxin Ma | Characterization of hsa_circ_0004277 as a New Biomarker for Acute Myeloid Leukemia via Circular RNA Profile and Bioinformatics Analysis | International Journal of Molecular Sciences | hsa_circ_0004277在急性髓系白血病中的作用 |
| 巴黎 – 萨克雷大学Hannu Myllykallio | High-Throughput Sequencing Reveals Circular Substrates for an Archaeal RNA ligase | RNA Biology | 古细菌中环状RNA相关的RNA连接酶 |
| 郑州大学附属河南省人民医院李牧蔚 | Hsa_circ_0054633 in peripheral blood can be used as a diagnostic biomarker of pre-diabetes and type 2 diabetes mellitus | Acta Diabetol | 外周血Hsa_circ_0054633可作为II型糖尿病标志物 |
| 意大利马尼塔斯大学Giulia Soldà | The Characterization of GSDMB Splicing and Backsplicing Profiles Identifies Novel Isoforms and a Circular RNA That Are Dysregulated in Multiple Sclerosis | International Journal of Molecular Sciences | GSDMB基因对应环状RNA与MLS的关系 |
| 天津医科大学洪伟 | The circular RNA ciRS-7 promotes APP and BACE1 degradation in an NF-κB-dependent manner | The FEBS Journal | ciRS-7通过NF-κB通路促进APP 和 BACE1降解 |
| 广州军区总医院 Zhang Ying | CircRNA hsa_circ_0005105 up regulates NAMPT expression and promotes chondrocyte extracellular matrix degradation by sponging miR-26a | Cell Biology International | hsa_circ_0005105上调NAMPT |
| 南京医科大学Yun Chen和Feng Yan | Circular BANP, an upregulated circular RNA that modulates cell proliferation in colorectal cancer | Biomedicine & Pharmacotherapy | 环状RNA circBANP在结肠癌中过表达 |
| 中山三院Wei Lai | Circular RNA profiling reveals that circCOL3A1-859267 regulate type I collagen expression in photoaged human dermal fibroblasts | BBRC | circCOL3A1-859267在人真皮成纤维细胞表达光老化中I型胶原表达 |
| 南京医科大学附属医院Cheng Qing和Jia Ruizhe | Circular RNAs expressed in chorionic villi are probably involved in the occurrence of recurrent spontaneous abortion | Biomedicine & Pharmacotherapy | 环状RNA与复发性流产 |
| 四川大学Li Yizhou和Li Menglong | Computational identifying and characterizing circular RNAs and their associated genes in hepatocellular carcinoma | PLoS One | 基于数据信息分析找出肝细胞癌相关的环状RNA |
| 法国施维雅研究所Mark J. Millan | Linking deregulation of non-coding RNA to the core pathophysiology of Alzheimer’s disease: an integrative review | Progress in Neurobiology | 阿尔兹海默症非编码RNA综述,涉及到环状RNA在其中的作用 |
| 西安交通大学He Aili | Circles reshaping the RNA world: from waste to treasure | Molecular Cancer | 环状RNA综述 |
| 卢森堡卢森堡健康研究所心血管研究所Yvan Devaux | Circular RNAs in heart failure | European Journal of Heart Failure | 心力衰竭中环状RNA研究 |
| 浙江大学医学院He Jun和第三军医大学Li Yongsheng | Circular RNAs and Cancer | Cancer Letters | 肿瘤环状RNA进展综述 |
| 浙江大学Fan Longjiang | PlantcircBase: a database for plant circular RNAs | Molecular Plant | 植物环状RNA数据库 |
| 青岛大学Kun Wang和Peifeng Li | The Role and Molecular Mechanism of Non-Coding RNAs in Pathological Cardiac Remodeling | International Journal of Molecular Sciences | 非编码RNA在病理性心脏重塑中的作用和分子机制 |
和风送暖,沐浴着春天的阳光,拜读着环状RNA最新的研究进展,心情激动万分。环状RNA研究的春天来了。
参考文献:
1. Yang, Y., et al., Extensive translation of circular RNAs driven by N6-methyladenosine. Cell Res, 2017.
2. Chan Zhou, B.M., Kaveh Daneshvar, Joshua V. Pondick, Jinkai Wang, Nicholas O. Van Wittenberghe, Yi Xing, Cosmas C. Giallourakis, and Alan C. Mullen, Identification and characterization of m6A circular RNA epitranscriptomes. BioRxiv, 2017.
3. Pamudurti, N.R., et al., Translation of CircRNAs. Mol Cell, 2017.
4. Legnini, I., et al., Circ-ZNF609 Is a Circular RNA that Can Be Translated and Functions in Myogenesis. Mol Cell, 2017.
5. Aktas, T., et al., DHX9 suppresses RNA processing defects originating from the Alu invasion of the human genome. Nature, 2017.
6. Tay, M.L. and J.W. Pek, Maternally Inherited Stable Intronic Sequence RNA Triggers a Self-Reinforcing Feedback Loop during Development. Curr Biol, 2017.
7. Errichelli, L., et al., FUS affects circular RNA expression in murine embryonic stem cell-derived motor neurons. Nat Commun, 2017. 8: p. 14741.
8. Hsiao, K.Y., et al., Non-coding effects of circular RNA CCDC66 promote colon cancer growth and metastasis. Cancer Res, 2017.
9. Bin Li, X.-Q.Z., Shu-Rong Liu, Wen-Ju Sun, Qiao Lin, Yu-Xia Luo, Ke -Ren Zhou, Chen-Min Zhang, Ye-Ya Tan, Jian -Hua Yang, Liang -Hu Qu, Discovering The Interactions Between Circular RNAs and RNA-binding proteins from CLIP-Seq Data using circScan. BioRxiv, 2017.
10. Gao, Y., J. Zhang, and F. Zhao, Circular RNA identification based on multiple seed matching. Brief Bioinform, 2017.
11. Li, M., et al., Quantifying circular RNA expression from RNA-seq data using model-based framework. Bioinformatics, 2017.
12. Kang, S., et al., Machine learning-based identification of endogenous cellular microRNA sponges against viral microRNAs. Methods, 2017.
.png)