单革 博士/教授(中国科学技术大学生命科学学院)
报告主题:环RNA的种类与功能

嘉宾简介:
美国佐治亚州立大学博士,先后在美国艾默瑞大学和耶鲁大学进行RNA方向的博士后研究。2010年进入中国科学技术大学工作。长期从事非编码RNA功能及功能机理方面的研究。实验室成立以来,发现了新的非编码RNA类型、研究了新的非编码RNA功能、揭示了新的非编码RNA作用机理、探索了新的非编码RNA研究方法。在国际高水平期刊发表多篇研究论文。获科技部中青年科技创新领军人才,基金委国家杰出青年基金,国家万人计划科技创新领军人才。
代表性成果介绍:
BioRxiv:线粒体来源circRNA及其功能
2019年6月12日,生物学预印本杂志BioRxiv发表了中国科学技术大学单革教授的最新研究成果,首次报道人与小鼠线粒体来源的circRNA(mecciRNAs)。mecciRNAs可以通过与TOM40和PNPASE相互作用调控蛋白进入线粒体(参考文献[1])。
真核生物的遗传物质除了细胞核中的基因组DNA,还包括胞质中线粒体的DNA。线粒体最早是由内共生的远古生物经过亿万年的进化后保留在真核生物细胞内的,是真核生物最重要的能量代谢场所。哺乳动物的线粒体DNA也可以被转录和翻译出蛋白,经过亿万年的进化,哺乳动物中线粒体DNA编码的蛋白有13个,除了这13种蛋白,其他的线粒体蛋白都是由核基因组表达后转运至线粒体的。本文中作者发现线粒体DNA也能形成circRNA,作者称其为mecciRNAs。功能研究表明mecciRNAs能够通过与TOM40和PNPASE相互作用调控细胞核编码的蛋白进入线粒体。本文是首次报道哺乳动物线粒体基因组编码的circRNA,具有特殊意义。(参考文献[1])
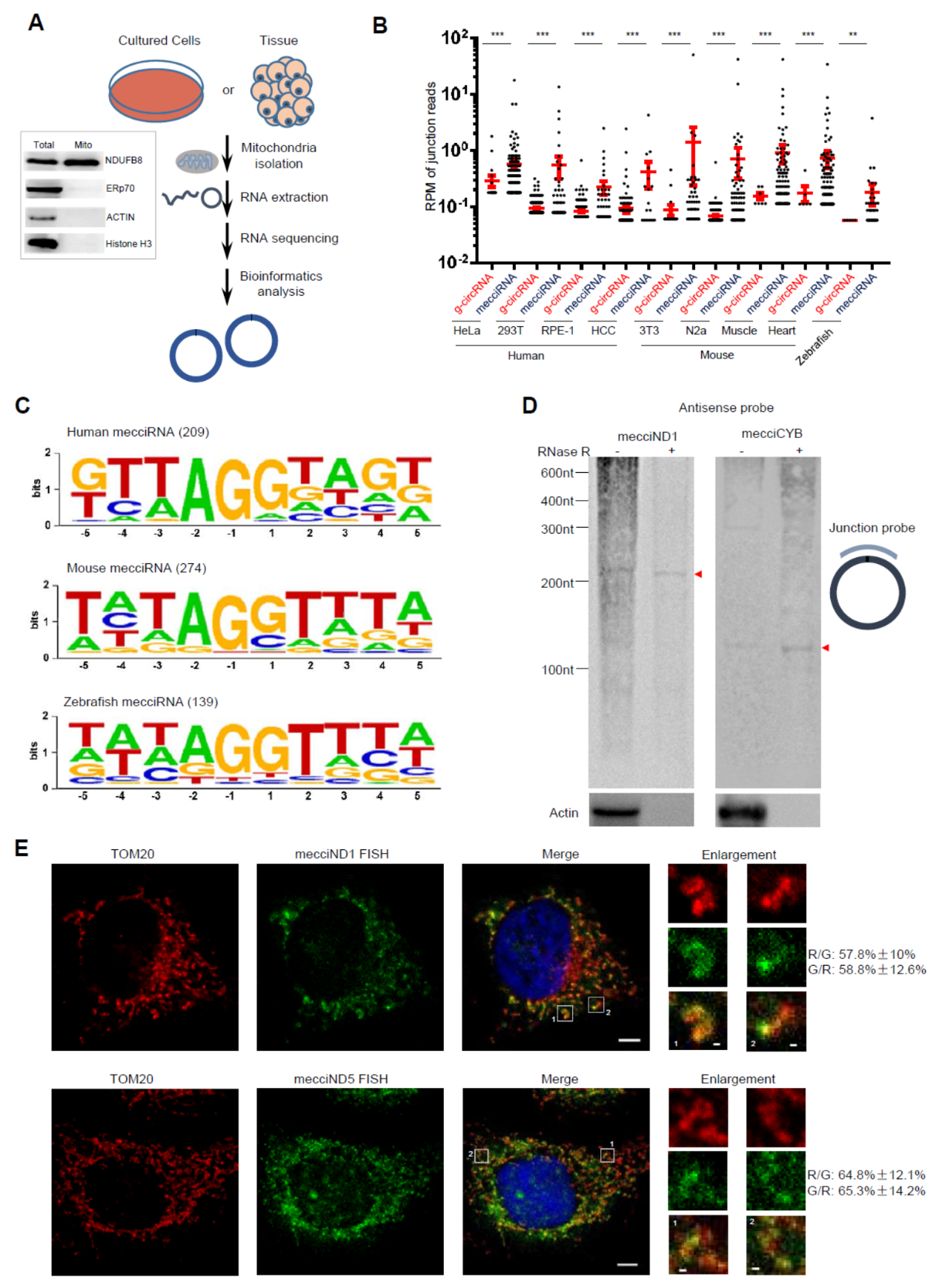
mecciRNAs鉴定与分析 (参考文献[1])
Nature Structural & Molecular Biology:EIciRNA调控基因转录
2015年2月9日,Nature Structural & Molecular Biology杂志发表了中国科学技术大学单革教授为通讯作者的文章,报道发现一类同时携带外显子和内含子的circRNA(EIciRNA)能定位于细胞核中,调控基因转录。
作者通过RNA聚合酶II的CLIP分析发现了这一类特殊的circRNA分子,它们既携带外线子序列,也有一段内含子序列,作者称其为Exon-Intron circular RNA (EIciRNA),功能分析表明EIciRNA倾向于定位于细胞核中,通过与U1 snRNP相互作用,促进这类circRNA的来源基因转录。这一项研究报道是早期circRNA功能研究的重要进展,为circRNA作为蛋白复合物支架的功能模型提供了有力证据。(参考文献[21])
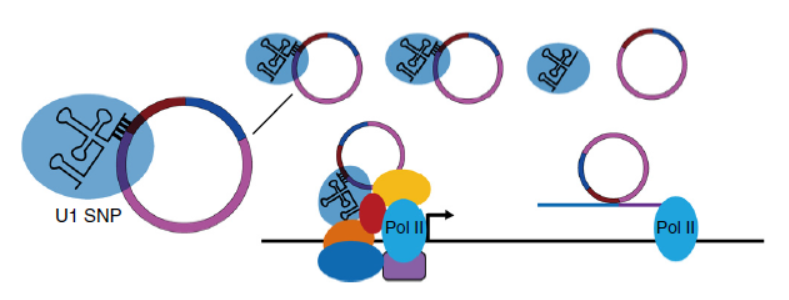
EIciRNA调控基因转录 (参考文献[21])
Frontiers in Genetics:肝癌中RNA表达网络分析
2019年5月8日,Frontiers in Genetics发表了中国科学技术大学生命科学学院单革教授,陈亮博士和中国科学技术大学附属第一医院(原安徽省立医院)许戈良主任为共同通讯作者的文章,报道了肝细胞癌临床标本中各种RNA的共表达网络分析。
本文主要基于高通量测序技术,分析miRNA,circRNA,lncRNA和mRNA的表达水平,然后进行共表达分析,找出HCC特异性的RNA表达变化状态,基于这些特定RNA分子做通路分析和临床标本验证。收集新鲜的HCC标本和癌旁组织对照的总RNA,小分子RNA建库测miRNA,去除核糖体RNA后建库做测序分析mRNA,lncRNA和circRNA,然后比较四对HCC与癌旁组织中相关分子的变化。(参考文献[2])
推荐阅读:Frontiers in Genetics:肝细胞癌中RNA 表达网络分析

Genome Biology:线虫Cas9敲除株系统分析lincRNA功能
2019年1月8日,中国科学技术大学单革教授为通讯作者在Genome Biology杂志发表了一项lincRNA的重要研究工作,他们通过Cas-9打靶技术在线虫中构建了155株lincRNA的敲除株,为系统研究lincRNA提供了重要工具资源。(参考文献[3])

Development Cell:线虫GABA能神经元中受UNC-30和UNC-55调控下游基因分析
2017年10月23日,Development Cell杂志发表一项线虫中基因表达调控机制的文章,报道利用Cas-9将GFP敲入UNC-30和UNC-55基因中,结合ChIP分析,系统研究了线虫GABA能神经元中受两种转录因子调控的下游基因。文章的通讯作者是中国科学技术大学的单革教授。(参考文献[8])

Nature Structural & Molecular Biology:保守的lncRNA中Alu序列增加可变剪切产物
2016年10月3日, Nature Structural & Molecular Biology杂志在线发表了中国科学技术大学单革教授为通讯作者的文章,报道发现一个从裂殖酵母到灵长类均保守的lncRNA,5S-OT。5S-OT可以在基因的原位发挥作用,调控下游的5S rRNA的转录。在灵长类中,该lncRNA存在多种可变剪切产物,基因组分析发现5S-OT中插入了一个反义Alu序列,该序列介道了5S-OT的可变剪切作用。(参考文献[13])

PNAS:特异性识别p53突变的核酸适配体
2015年8月11日,PNAS报道了中国科学技术大学单革教授为通讯作者的文章,报道开发一种可特异性识别p53突变的核酸适配体分子。(参考文献[20])

代表性研究成果列表
1. Xu Liu, Xiaolin Wang, Jingxin Li, Shanshan Hu, Yuqi Deng, Hao Yin, Xichen Bao, Qiangfeng Cliff Zhang, Geng Wang, Baolong Wang, Qinghua Shi1, Ge Shan. The identification of mecciRNAs and their roles in mitochondrial entry of proteins. bioRxiv, Jun. 12, 2019; doi: http://dx.doi.org/10.1101/668665.
2. Sheng Z, Wang X, Xu G*, Shan G*, Chen L*. Analyses of a Panel of Transcripts Identified from a Small Sample Size and Construction of RNA Networks in Hepatocellular Carcinoma. Front Genet. 2019 May 8;10:431. doi: 10.3389/fgene.2019.00431
3. Wei S, Chen H, Dzakah EE, Yu B, Wang X, Fu T, Li J, Liu L, Fang S, Liu W, Shan G*. Systematic evaluation of C. elegans lincRNAs with CRISPR knockout mutants. Genome Biol. 2019 Jan 8;20(1):7. doi: 10.1186/s13059-018-1619-6
4. Dzakah EE, Waqas A, Wei S, Yu B, Wang X, Fu T, Liu L, Shan G*. Loss of miR-83 extends lifespan and affects target gene expression in an age-dependent manner in Caenorhabditis elegans. J Genet Genomics. 2018 Dec 20;45(12):651-662. doi: 10.1016/j.jgg.2018.11.003
5. Chen L*, Dzakah EE, Shan G*. Targetable long non-coding RNAs in cancer treatments. Cancer Lett. 2018 Apr 1;418:119-124. doi: 10.1016/j.canlet.2018.01.042
6. Shah A, Rashid F, Awan HM, Hu S, Wang X, Chen L*, Shan G*. The DEAD-Box RNA Helicase DDX3 Interacts with m6A RNA Demethylase ALKBH5. Stem Cells Int. 2017:8596135. doi: 10.1155/2017/8596135
7. Wang X, Shan G. Nonradioactive Northern Blot of circRNAs. Methods Mol Biol. 2018; 1724:135-141. doi: 10.1007/978-1-4939-7562-4_11
8. Yu B, Wang X, Wei S, Fu T, Dzakah EE, Waqas A, Walthall WW, Shan G*. Convergent Transcriptional Programs Regulate cAMP Levels in C. elegans GABAergic Motor Neurons. Dev Cell. 2017 Oct 23;43(2):212-226.e7. doi: 10.1016/j.devcel.2017.09.013
9. Awan HM, Shah A, Rashid F, Wei S, Chen L*, Shan G*. Comparing two approaches of miR-34a target identification, biotinylated-miRNA pulldown vs miRNA overexpression. RNA Biol. 2018 Jan 2;15(1):55-61. doi: 10.1080/15476286.2017.1391441
10. Rashid F, Awan HM, Shah A, Chen L, Shan G*. Induction of miR-3648 Upon ER Stress and Its Regulatory Role in Cell Proliferation. Int J Mol Sci. 2017 Jun 29;18(7). pii: E1375. doi: 10.3390/ijms18071375.
11. Awan HM, Shah A, Rashid F, Shan G*. Primate-specific Long Non-coding RNAs and MicroRNAs. Genomics Proteomics Bioinformatics. 2017 Jun;15(3):187-195. doi: 10.1016/j.gpb.2017.04.002
12. Qian L, Xu F, Wang X, Jiang M, Wang J, Song W, Wu D, Shen Z, Feng D, Ling B, Cheng Y, Xiao W*, Shan G*, Zhou Y*. LncRNA expression profile of ΔNp63α in cervical squamous cancers and its suppressive effects on LIF expression. Cytokine. 2017 Aug;96:114-122. doi: 10.1016/j.cyto.2017.04.001
13. Hu S, Wang X, Shan G*. Insertion of an Alu element in a lncRNA leads to primate-specific modulation of alternative splicing. Nat Struct Mol Biol. 2016 Nov;23(11):1011-1019. doi: 10.1038/nsmb.3302.
14. Rashid F, Shah A, Shan G*. Long Non-coding RNAs in the Cytoplasm. Genomics Proteomics Bioinformatics. 2016 Apr;14(2):73-80. doi: 10.1016/j.gpb.2016.03.005
15. Yu B, Shan G*. Functions of long noncoding RNAs in the nucleus. Nucleus. 2016 Apr 25;7(2):155-66. doi: 10.1080/19491034.2016.1179408
16. Chuan Huang, Xiaolin Wang, Xu Liu, Shuhuan Cao, Ge Shan*. RNAi pathway participates into chromosome segregation in mammalian cells. Cell Discovery 2015, 1(1): 15029. doi:10.1038/celldisc.2015.29
17. Chen, Liang; Huang, Chuan; Wang, Xiaolin; Shan, Ge*. Circular RNAs in Eukaryotic Cells. Current Genomics 2015, 16(5): 312-318.
18. Liang Chen, Ge Shan*. Circular RNAs remain peculiarly unclear in biogenesis and function. Science China Life Sciences 2015, 58(6): 616-618.
19. Chuan Huang, Ge Shan*. What happens at or after transcription: Insights into circRNA biogenesis and function.Transcription 2015, 6(4): 61-64.
20. Liang Chen, Farooq Rashid, Abdullah Shah, Hassaan M. Awan, Mingming Wu, An Liu, Jun Wang, Tao Zhu, Zhaofeng Luo, Ge Shan*. The isolation of an RNA aptamer targeting to p53 protein with single amino acid mutation. P.N.A.S. 2015, 112(32):10002-10007.
21. Zhaoyong Li, Chuan Huang, Chun Bao, Liang Chen, Mei Lin, Xiaolin Wang, Guolin Zhong, Bin Yu, Wanchen Hu, Limin Dai, Pengfei Zhu, Zhaoxia Chang, Qingfa Wu, Yi Zhao, Ya Jia, Ping Xu, Huijie Liu, Ge Shan*. Exon-Intron circular RNAs regulate transcription in the nucleus. Nat. Struct. Mol. Bio.2015, 22(3):256-264.
22.LF Meng, L Chen, ZY Li, ZX Wu, G Shan*. Environmental RNA interference in animals. Chinese Science Bulletin, 2013; 58(35):4418-4425.
23. Meng L, Chen L, Li Z, Wu ZX, Shan G*. Roles of MicroRNAs in the Caenorhabditis elegans Nervous System. J Genet Genomics. 2013; 40(9):445-452.
24. Yang L, Meng Y, Bao C, Liu W, Ma C, Li A, Xuan Z, Shan G*, Jia Y*. Robustness and backbone motif of a cancer network regulated by miR-17-92 cluster during the G1/S transition. PLoS One. 2013;8(3):e57009.
25.Liu H, Wang X, Wang HD, Wu J, Ren J, Meng L, Wu Q, Dong H, Wu J, Kao TY, Ge Q, Wu ZX, Yuh CH, Shan G*. Escherichia coli noncoding RNAs can affect gene expression and physiology of Caenorhabditis elegans. Nature Communications, 2012, 25 (3): 1073. doi: 10.1038/ncomms2071.
26.Hu S, Wu J, Chen L, Shan G*. Signals from noncoding RNAs: Unconventional roles for conventional pol III transcripts. The International Journal of Biochemistry & Cell Biology, 2012; 44(11): 1847-51. doi: 10.1016/j.biocel.2012.07.013.
27. Lin Mei, Wu Jing, Shan Ge*. Noncoding RNAs: different roles in tumorigenesis. Chinese Science Bulletin, 2012; 57(9): 959-965.
.png)