早些时候对circRNA-disease相关的数据库以及方法进行过盘点,虽然预测工具众多(>67个),然而对用户非常不友好。
详情请访问:circRNA-disease数据库与计算工具盘点
2023年8月16日,西安交通大学付来义团队发表在 Briefings in Bioinformatics (IF=9.5)的文章【KGETCDA: an efficient representation learning framework based on knowledge graph encoder from transformer for predicting circRNA-disease associations 】打破了这个尴尬的局面。

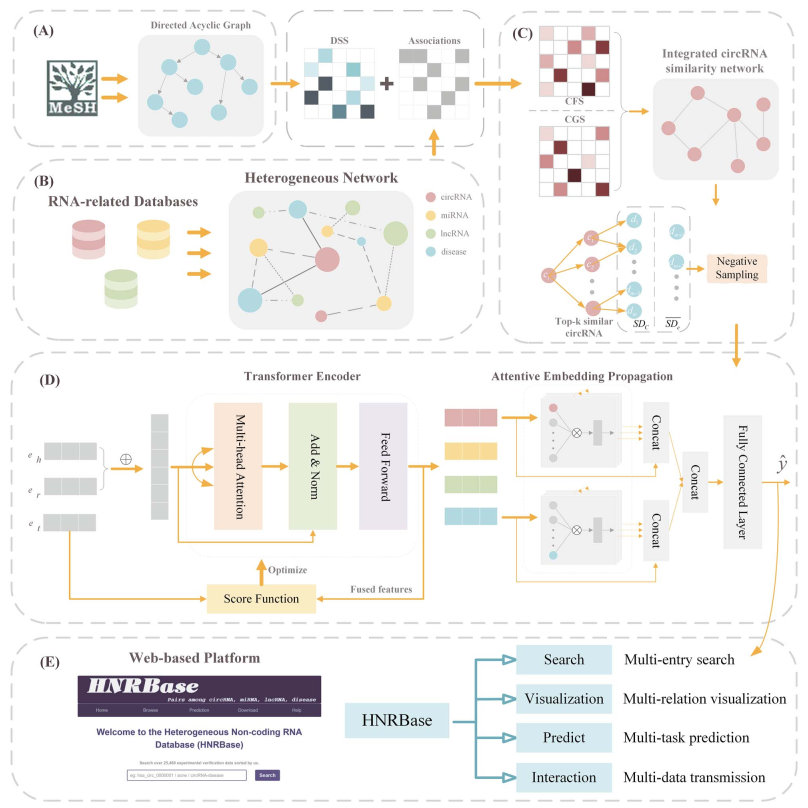
研究团队开发了一个用于预测 circRNA-disease关系的深度学习方法 KGETCDA(Knowledge Graph Encoder from Transformer for predicting CircRNA-Disease Associations)。整合了10多个非编码疾病关系数据库,构建了circRNA、miRNA、lncRNA与疾病的关系数据集,根据该数据集构建了生物学知识图;然后构建了Transformer(继CNN和RNN之后又一个高效的特征提取器)知识提取图层,精确地捕获高阶的交互信息;最后多层感知器被用来预测circRNA-disease associations(CDAs)。
 经过评估,KGETCDA比其他最新的方法性能都要优越。
经过评估,KGETCDA比其他最新的方法性能都要优越。
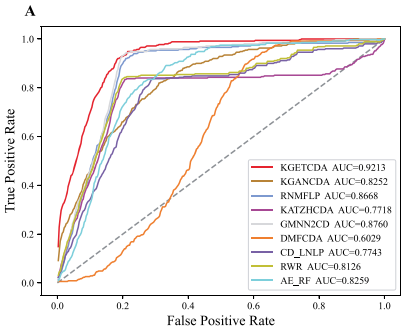
图A.AUC比较
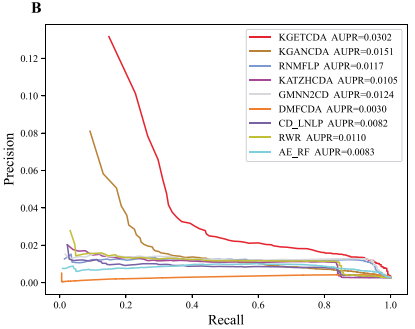 图B.AUPR比较
图B.AUPR比较
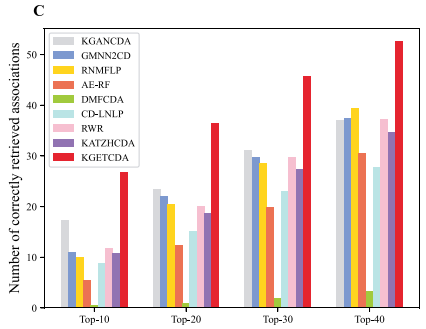 图C.准确评估数量比较
图C.准确评估数量比较
最后,为了让大家享受到自己的研究成果,作者还开发了一个用户友好的网络平台HNRBase(网址:http://lab-fly.site/KGETCDA)。下图展示了网络平台的一些功能。

.png)