环状RNA(circRNA)由下游剪接供体和上游剪接受体共价连接而成。其最重要的功能之一是通过结合RNA结合蛋白(RBP)发挥作用。然而,目前还没有有效的算法来识别全基因组的circRNA和RBP相互作用。
为此,华中农业大学生物医学与健康学院团队开发了一种独特的算法——circRIP,用于从RNA免疫沉淀测序(RIP-Seq)数据中识别circRNA与RBP互作。circRIP可以敏感、特异且系统地从RIP-Seq和eCLIP-seq数据中识别RBP和circRNA的相互作用,这可以造福于研究学者对circRNA结合RBP的功能探索。该成果发表在Briefings in Bioinformatics期刊上。
虽然RIP-Seq是一种检测RBP和RNA相互作用的有效技术,但目前基于RIP-Seq的pipeline和算法是专门针对线性RNA设计的,不适用于circRNA。为了克服这一限制,作者开发了一种独特的方法来检测RBP和circRNA的结合,流程如图1所示。首先,根据之前广泛使用的算法,对IP和RIP-Seq输入样本中的circRNAs进行识别,并提取circRNA的计数[1]。使用每百万计数(CPM)来标准化circRNA的计数。接下来,将IP和Input的所有读数(reads)映射到基因组中,以计数宿主基因的正向剪接点(forward spliced reads)。
然后计算在IP和Input中的circRNA表达倍数变化。为了评估抗体对circRNA的显著富集程度,采用条件法检测两个泊松率(Poisson rates)之比来比较IP和Input样本中检测到circRNA的概率,并生成p值。
最后,结合circRNA表达的倍数变化和p值,估算IP中circRNA的富集程度。倍数变化≥2且P值<0.05的circRNA被认为是IP富集的circRNA,也就是RIP结合的circRNA。否则,认为是一个非富集的circRNA。综上,作者研发了一个名为circRIP 的工具,可以通过github访问(https://github.com/bioinfolabwhu/circRIP)。
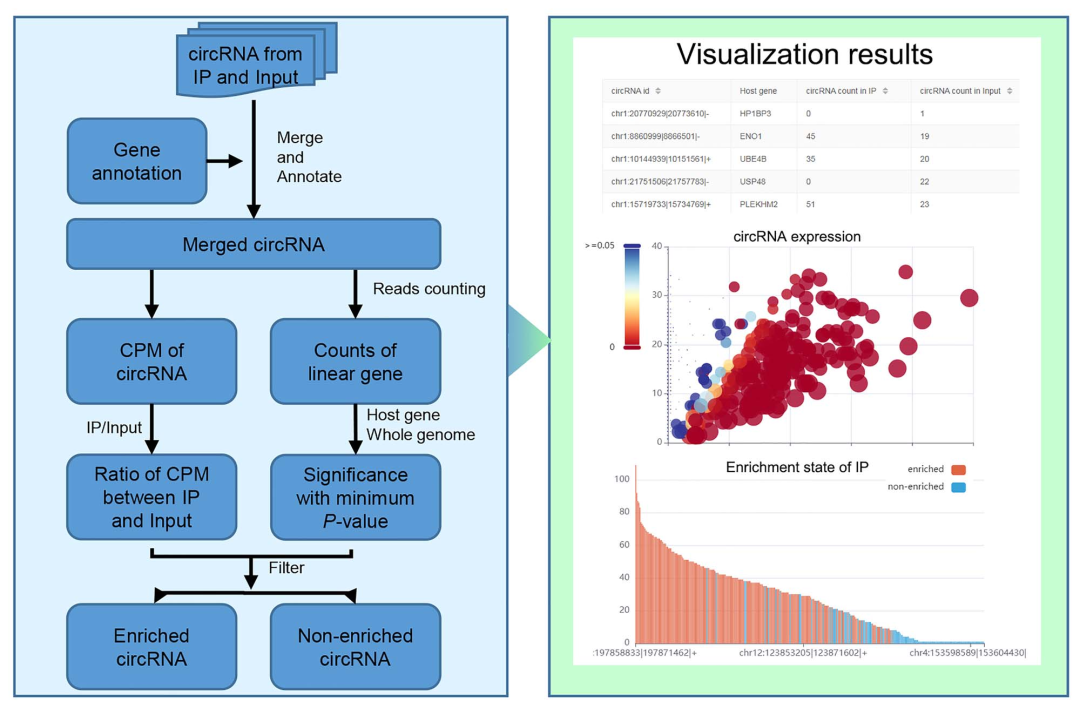
图1. circRIP的原理图和工作流程。左图:circRIP的设计;右图:circRIP结果的可视化。
作者构建模拟了RIP-Seq数据,并测试了circRIP的敏感性和特异性。首先,从GENCODE和circBase中分别获得人类基因组中所有线性转录本和circRNA的全长序列。然后,随机选取9000个线性转录本和1000个具有潜在表达的circRNA构建模拟Input样本。最后,随机加入IP/Input和背景的倍数变化来模拟IP样本。作者将倍数变化随机设为2-10,结果显示>88%的富集circRNA被召回(AUC=0.96)。然后,通过分别设置2到10的倍数变化生成9个模拟数据集。通过应用circRIP,观察九个数据集的可靠性能:9个数据集的平均AUC为0.92。在数据集中观察到最低的AUC为0.9,变化为2倍,表明circRIP性能稳健。作者评估模拟数据集的假发现率(FDR),结果表明,9个模拟数据集的FDR中位数为0.005,表明circRIP能够以高灵敏度和特异性识别IP富集(RBP结合)的circRNA。并且circRIP比Clirc (AUC=0.76)具有显著优势(AUC=0.94)。
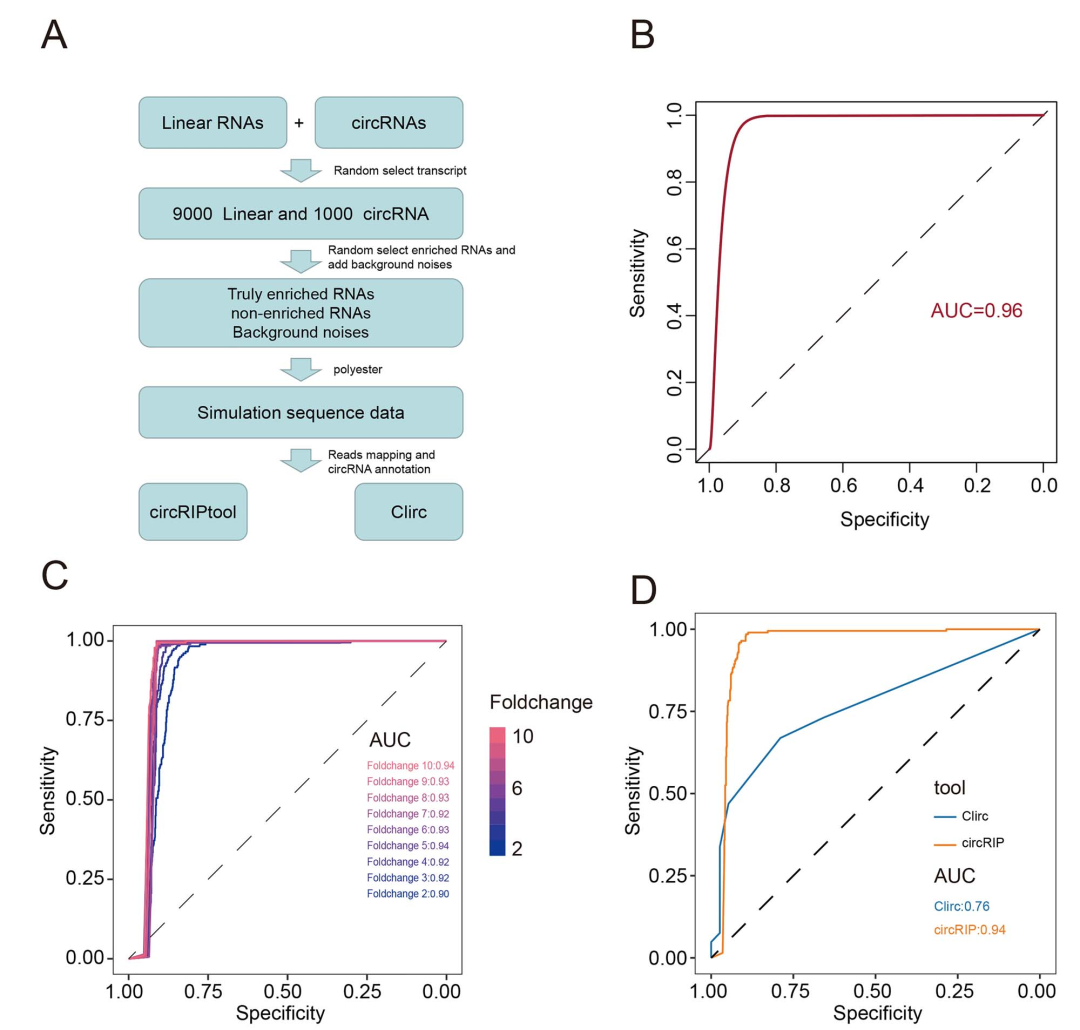 图2. 模拟测试circRIP的性能
基于RIP-Seq数据鉴定IGF2BP3结合的circRNA
IGF2BP3是调控mRNA稳定性和翻译的m6A readers之一。为了研究IGF2BP3和circRNA之间的相互作用,作者使用circRIP分析了WM278细胞系中的IGF2BP3 RIP-Seq数据集。总共鉴定出1285个circRNA,其中95个circRNA在IP样本中显著富集,表明它们与IGF2BP3存在潜在的相互作用。并且CDR1as是与IGF2BP3结合最多的富集的circRNA,这与之前RIP-PCR结果一致,表明了circRIP的可靠性。作者还比较了IP样本中IGF2BP3富集和非富集circRNA的reads计数(reads count),发现IP样本中也检测到大量来自非富集circRNA的reads,这表明在识别RBP结合circRNA时,消除这些circRNA是必要的,而circRIP也是这样做的。并且circRIP可以识别出,富含IGF2BP3的circRNA的基因组分布主要来自外显子,而不是内含子和基因间区。
图2. 模拟测试circRIP的性能
基于RIP-Seq数据鉴定IGF2BP3结合的circRNA
IGF2BP3是调控mRNA稳定性和翻译的m6A readers之一。为了研究IGF2BP3和circRNA之间的相互作用,作者使用circRIP分析了WM278细胞系中的IGF2BP3 RIP-Seq数据集。总共鉴定出1285个circRNA,其中95个circRNA在IP样本中显著富集,表明它们与IGF2BP3存在潜在的相互作用。并且CDR1as是与IGF2BP3结合最多的富集的circRNA,这与之前RIP-PCR结果一致,表明了circRIP的可靠性。作者还比较了IP样本中IGF2BP3富集和非富集circRNA的reads计数(reads count),发现IP样本中也检测到大量来自非富集circRNA的reads,这表明在识别RBP结合circRNA时,消除这些circRNA是必要的,而circRIP也是这样做的。并且circRIP可以识别出,富含IGF2BP3的circRNA的基因组分布主要来自外显子,而不是内含子和基因间区。
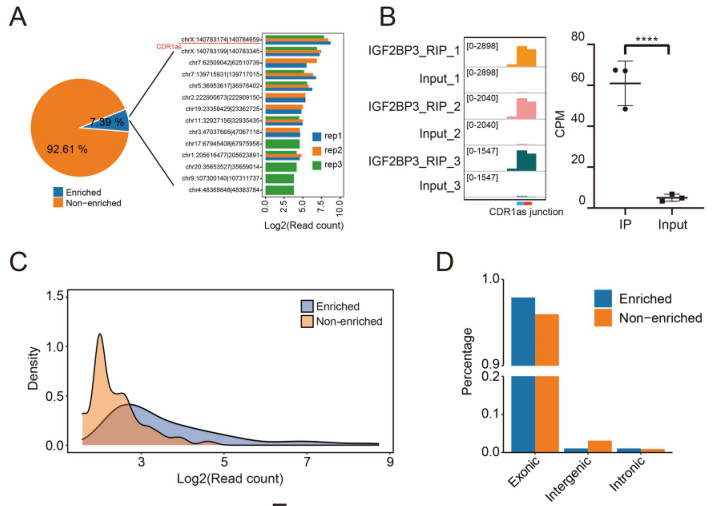
图3. 基于RIP-Seq数据的IGF2BP3富集circRNA的鉴定
基于eCLIP数据识别数千个circRNA-RBP相互作用
作者从ENCODE项目(https://www.encodeproject.org/)中收集了119个K562细胞系RBPs和103个HepG2细胞系RBPs的eCLIP数据集,测试circRIP在eCLIP数据中的性能。在鉴定出的10361个和5433个circRNA中,分别有2823和1333个circRNA在K562和HepG2细胞中被RBP显著富集,这表明circRNA和RBP存在广泛的相互作用(http://bib.oxfordjournals.org/)。作者比较了RBP在circRNA和线性RNA上的结合motif,发现大多数motif在circRNA和线性RNA中显著相似。随机选取的验证结果与上述一致。
进一步研究这些RBP结合circRNA的基因组分布发现,RBP AQR与大部分来自内含子区的circRNA结合,而非外显子区和基因间区。这显示AQR可能在内含子circRNA的生成中发挥重要作用。此外,此外,另一种RBP SF3B4也与更多内含子circRNA结合,而其他RBP与外显子circRNA结合更多一些。
为了揭示这些circRNA的潜在功能,作者进行了通路分析验证这些RBP结合的circRNA在多个生物学过程中富集。例如,AQR-结合的circRNA在有丝分裂细胞周期、蛋白质分解代谢、DNA损伤、mRNA代谢和组蛋白修饰中富集。DROSHA结合的circRNA参与蛋白质分解代谢、细胞内转运和mRNA代谢过程。这些结果表明,circRNA RBP相互作用在细胞系中普遍存在,并可能在病理或生理过程中发挥重要作用。
综上两个案例表明:circRIP运用到研究circRNA的RBP功能上,具有灵敏、特异和适用性广泛的优点。
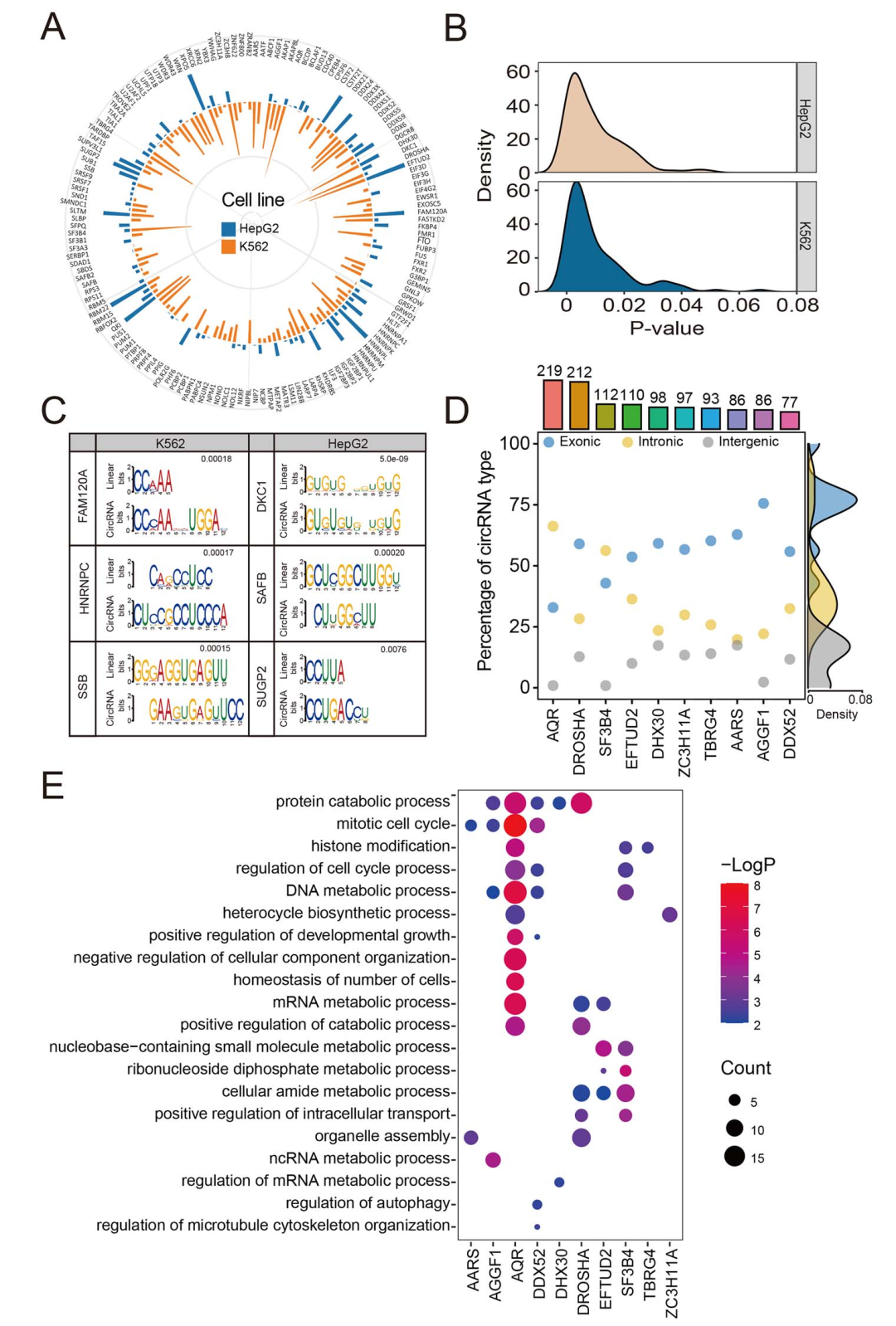 图4. 基于eCLIP数据的circRNA-RBP
作者基于RIP-Seq和eCLIP数据,研发了一种独特的算法——circRIP,用于识别circRNA和RBP相互作用,并运用了模拟实验和真实案例验证了circRIP的敏感性和特异性,弥补了目前尚无有效算法来识别circRNA 和RBP互作的缺陷。为研究circRNA的RBP功能增添了浓墨重彩的一笔。
原文链接:https://doi.org/10.1093/bib/bbac186
图4. 基于eCLIP数据的circRNA-RBP
作者基于RIP-Seq和eCLIP数据,研发了一种独特的算法——circRIP,用于识别circRNA和RBP相互作用,并运用了模拟实验和真实案例验证了circRIP的敏感性和特异性,弥补了目前尚无有效算法来识别circRNA 和RBP互作的缺陷。为研究circRNA的RBP功能增添了浓墨重彩的一笔。
原文链接:https://doi.org/10.1093/bib/bbac186
参考文献:
[1] Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature 2013;495:333–8.
转载请联系邮箱授权:circRNA@163.com

 图2. 模拟测试circRIP的性能
图2. 模拟测试circRIP的性能

 图4. 基于eCLIP数据的circRNA-RBP
图4. 基于eCLIP数据的circRNA-RBP
.png)