哺乳动物肠上皮的快速周转和再生由定位于肠隐窝的肠道干细胞(ISCs)支持,这些细胞向上迁移并分化为肠细胞、簇毛细胞、杯状细胞或肠内分泌细胞[1]。在再生过程中,组织模式和内稳态受到多种信号通路调控,包括Wnt,Notch和BMP等。虽然维持干细胞功能和隐窝稳态的信号通路需求已经得到了很好的研究,但ISCs自我更新能力的潜在机制仍有待进一步探索。环状RNA (circRNAs)由亲代基因的外显子或内含子通过反向剪接而成。由于circRNA的高稳定性,它们可以在特定的细胞类型中以一种临时调节的方式积累[2]。而针对ISCs,有报道circPan3可通过小位ILC2s产生的IL-13促进ISCs的自我更新,而更多的机制研究有待进一步发现。
2023年1月,中国科学院生物物理研究所范祖森教授在The EMBO Journal发表文章:Noncoding RNA circBtnl1 suppresses self-renewal of intestinal stem cells via disruption of Atf4 mRNA stability。作者发现了一种在ISCs中高度表达的circRNA—circBtnl1。circBtnl1的缺失增强了小鼠ISCs的自我更新能力和上皮再生,而其亲本基因Btnl1的mRNA和蛋白质水平没有变化。ATF4可通过独特的基序与Sox9启动子结合,激活了Sox9的转录,增强了ISCs的自我更新能力和上皮再生。在机制上,circBtnl1可与Atf4 mRNA竞争性结合ATP依赖的RNA解旋酶Ddx3y,损害Atf4 mRNA在野生型ISCs中的稳定性,抑制ISCs的活性。这表明circBtnl1介导的Atf4 mRNA衰变抑制了Sox9转录,从而负向调节了ISCs的自我更新机制。 为了探索circRNA在ISCs中的作用,作者从C57BL/6小鼠中分离出小肠隐窝,并进行高通量circRNA-seq。然后,在小鼠ISCs中选择了前8个高表达的circRNA (circOgdh, circRasa2a, circBtnl1, circMed13I, circVapa, circSlc43a2, circCnot6I, circAlg12),均做了背靠背引物设计验证成环,RNase R和放线菌素D验证它们的稳定性。接下来,作者在ISCs中使用shRNA分别敲低这8种circRNA,并建立了类器官分析来确定它们的损失对ISCs活性的影响。结果发现敲低circBtnl1最显著地诱导了类器官的形成。许多circRNA表现出组织特异性的表达模式,这意味着它们在各种组织中具有特定的功能。
为了探索circRNA在ISCs中的作用,作者从C57BL/6小鼠中分离出小肠隐窝,并进行高通量circRNA-seq。然后,在小鼠ISCs中选择了前8个高表达的circRNA (circOgdh, circRasa2a, circBtnl1, circMed13I, circVapa, circSlc43a2, circCnot6I, circAlg12),均做了背靠背引物设计验证成环,RNase R和放线菌素D验证它们的稳定性。接下来,作者在ISCs中使用shRNA分别敲低这8种circRNA,并建立了类器官分析来确定它们的损失对ISCs活性的影响。结果发现敲低circBtnl1最显著地诱导了类器官的形成。许多circRNA表现出组织特异性的表达模式,这意味着它们在各种组织中具有特定的功能。
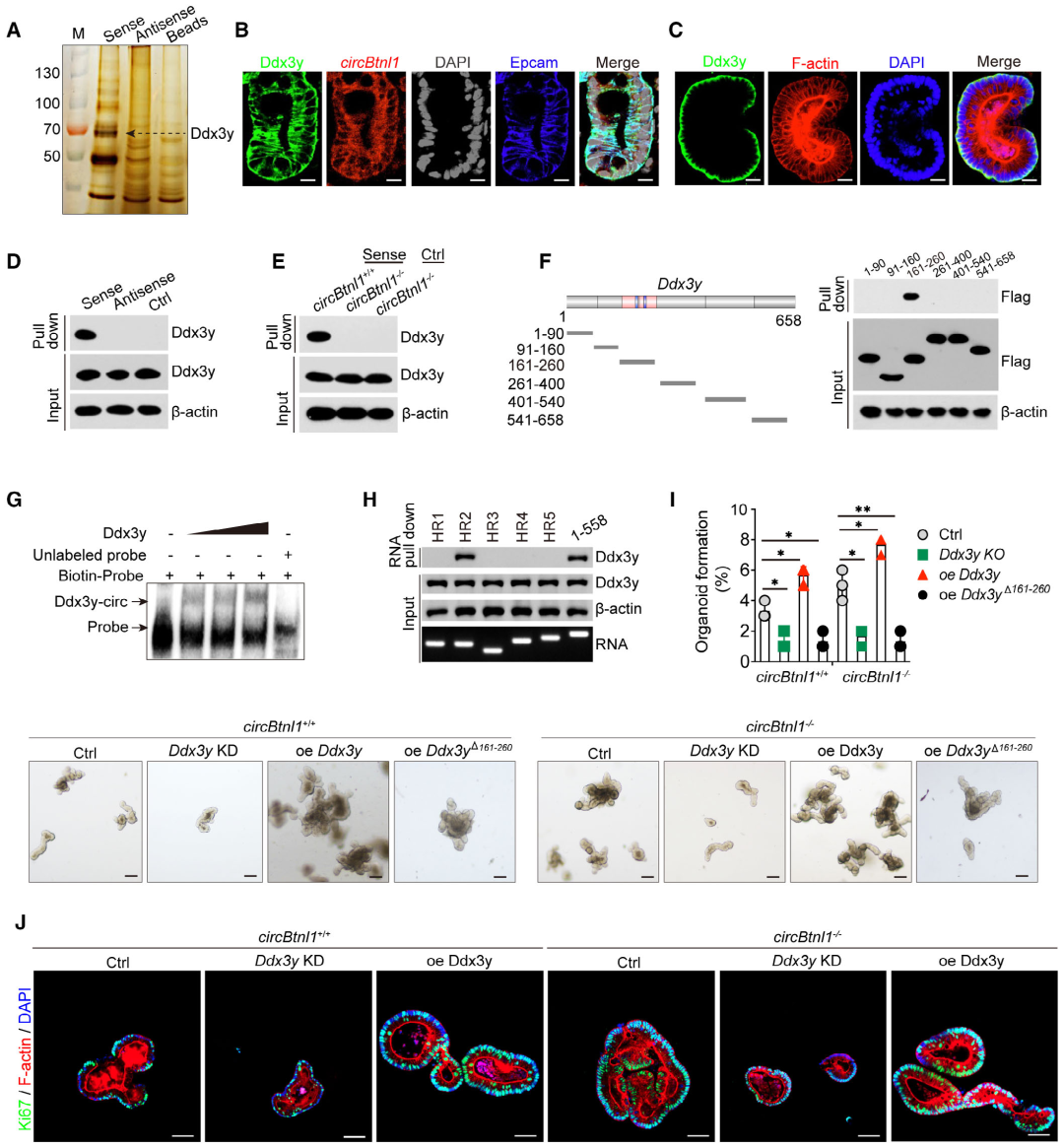
接下来,作者评估了circBtnl1在ISCs中的功能。敲低circBtnl1显著破坏了小肠和结肠的类器官形成能力。FISH检测了circBtnl1在小鼠各组织中的表达,作者发现circBtnl1在各组织中广泛表达,其中小肠、结肠和胃是高表达的前三个组织;并且circBtnl1仅定位于肠隐窝和小肠类器官的细胞质中,核-细胞质分离实验也验证了这一结果。这些结果表明circBtnl1在小鼠ISCs中高表达。
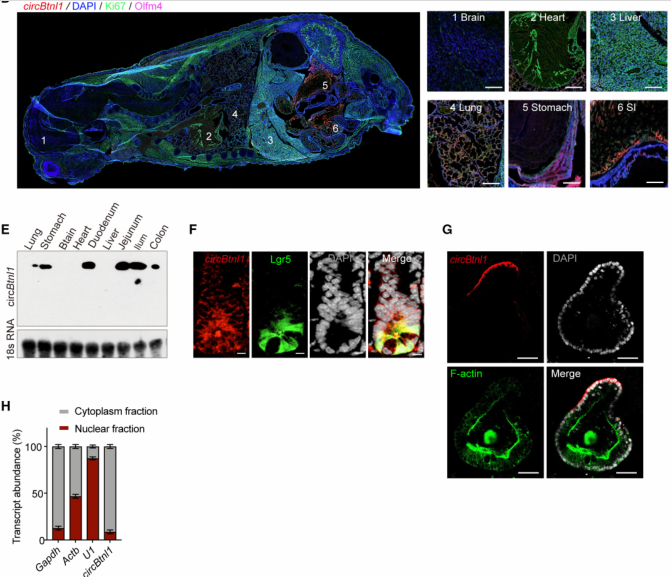
图1. circBtnl1在ISCs中高表达
CircBtnl1基因敲除促进了ISCs的自我更新
为了确定circBtnl1在调节ISCs中的生理作用,作者用CRISPR/Cas9技术构建circBtnl1敲除小鼠模型。作者发现circBtnl1敲除小鼠相比对照组小鼠肠道长度、隐窝数量及细胞数量增加;Ki67和EdU染色检测circBtnl1敲除促进小鼠ISCs的增殖。提示circBtnl1敲除促进ISCs的增殖。
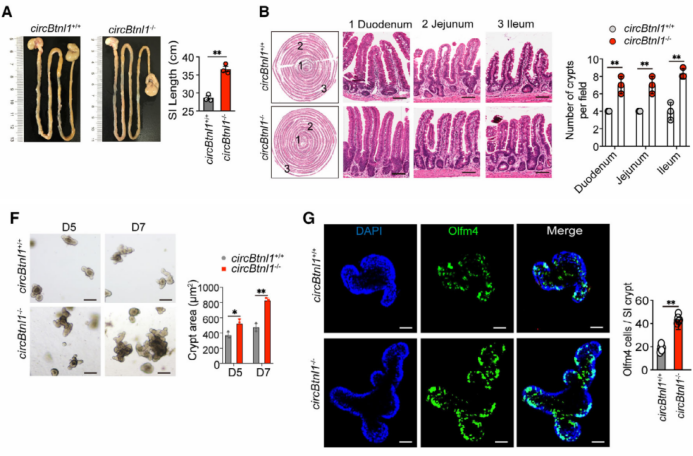
图2. circBtnl1基因敲除促进了ISCs的自我更新
鉴于circBtnl1的缺失不会改变其亲本基因的mRNA和蛋白水平,circBtnl1可以通过其他途径调节ISCs的自我更新,比如发挥RBP功能。为了鉴定circBtnl1的结合蛋白候选位点,作者在小鼠ISCs裂解液中进行了RNA pulldown和质谱测定。Ddx3y被确定为相关的候选蛋白。Ddx3y是一个依赖于ATP的RNA解旋酶。此外,RNA-FISH和免疫荧光实验证实circBtnl1与Ddx3y在小鼠小肠隐窝和ISCs类器官的细胞质中共定位。通过RNA pulldown和WB分析进一步验证了circBtnl1与Ddx3y的相互作用。通过Uniprot网站分析,Ddx3y中有两个RNA结合位点,表明Ddx3y可以作为RNA结合点。体外测图发现,Flag标记的Ddx3y蛋白的RNA结合域对其与circBtnl1的相互作用至关重要。片段映射分析亦发现,circBtnl1转录本(HR2)的外显子4与Ddx3y蛋白结合是必要的。这些结果提示circBtnl1在小鼠ISCs中主要定位于细胞质,并与Ddx3y蛋白相互作用。
作者发现,过表达Ddx3y显著促进了组织腔形成能力。而circbtnl1则逆转Ddx3y过表达在类器官中的作用。Ki67染色检测小肠类器官的结果与上述结果一致。此外,circBtnl1在类器官形成和增殖中的作用可以受Ddx3y反向调控,这表明circBtnl1在ISCs中通过Ddx3y发挥作用。
图3. CircBtnl1在ISCs中与Ddx3y结合
CircBtnl1和Atf4 mRNA竞争性结合Ddx3y,调节Atf4 mRNA的稳定性
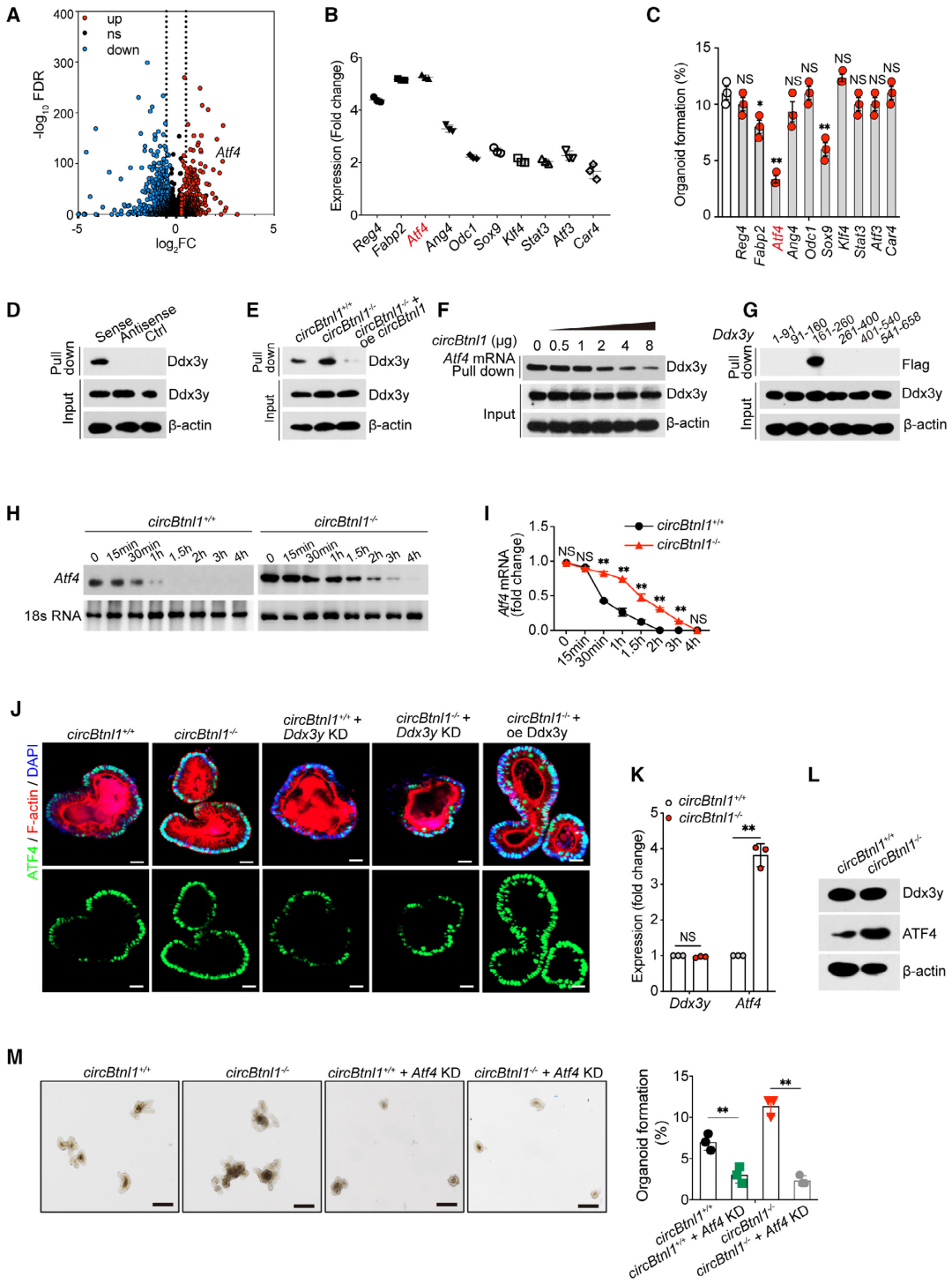
为了进一步鉴定circBtnl1的靶基因,作者对模型小鼠的Lgr5+ ISCs进行了转录组微阵列分析。经过重重筛选,作者观察到Atf4下调后,类器官形成效果最差,提示Atf4对ISC活性的正向调控作用。而巧合的是,Atf4 mRNA能够与Ddx3y相互作用。而过表达circBtnl1则破坏Atf4 mRNA与Ddx3y的相互作用。接下来,作者确定Ddx3y和circBtnl1是否结合在Atf4 mRNA的同一位点上。正如预期的那样,WB证实Atf4 mRNA与Ddx3y的相互作用呈剂量依赖性;截断作图显示Atf4 mRNA与Ddx3y的相互作用位点与circBtnl1相同,提示CircBtnl1和Atf4可竞争性结合Ddx3y。敲除circBtnl1后,ISCs Atf4 mRNA的稳定性增强,表明circBtnl1负向调节Atf4 mRNA的稳定性。为了确定Ddx3y在ISCs自我更新中的作用,作者发现在circBtnl1敲除的小鼠类器官中,Ddx3y敲除显著降低ATF4的表达,而过表达Ddx3y的结果相反,提示Ddx3y正向调控ATF4的表达。但值得注意的是,无论是在哪种模型小鼠上,Atf4敲除显著减少了ISCs形成类器官的能力。综上所述,circBtnl1缺失使得Ddx3y释放,从而促进Atf4 表达。
图4. CircBtnl1和Atf4 mRNA竞争性结合Ddx3y,调节Atf4 mRNA的稳定性
CircBtnl1敲除可促进ATF4进入Sox9启动子,触发其转录
作者进一步确定ATF4在下游靶基因调控中的生物学功能。通过Homer实验预测了小鼠(向上)和人类(向下)Sox9启动子中预测的ATF4-结合基序。发现ATF4在Sox9启动子的100 ~ 200 bp区域富集。双荧光素酶报告实验也发现了Sox9启动子缺失显著降低ATF4介导的Sox9荧光素酶活性。Sox9可促进ISCs自我更新。circBtnl1缺陷始终导致Sox9 mRNA转录升高。在circBtnl1敲除类器官中,敲低Atf4显著降低Sox9表达;相比之下,过表达circBtnl1可抑制Sox9的表达。这些数据表明,Sox9在ISC自我更新维持调控中作为ATF4的下游靶点。综上所述,circBtnl1的缺失促进了ATF4的表达,从而增强了ISCs中Sox9的转录。
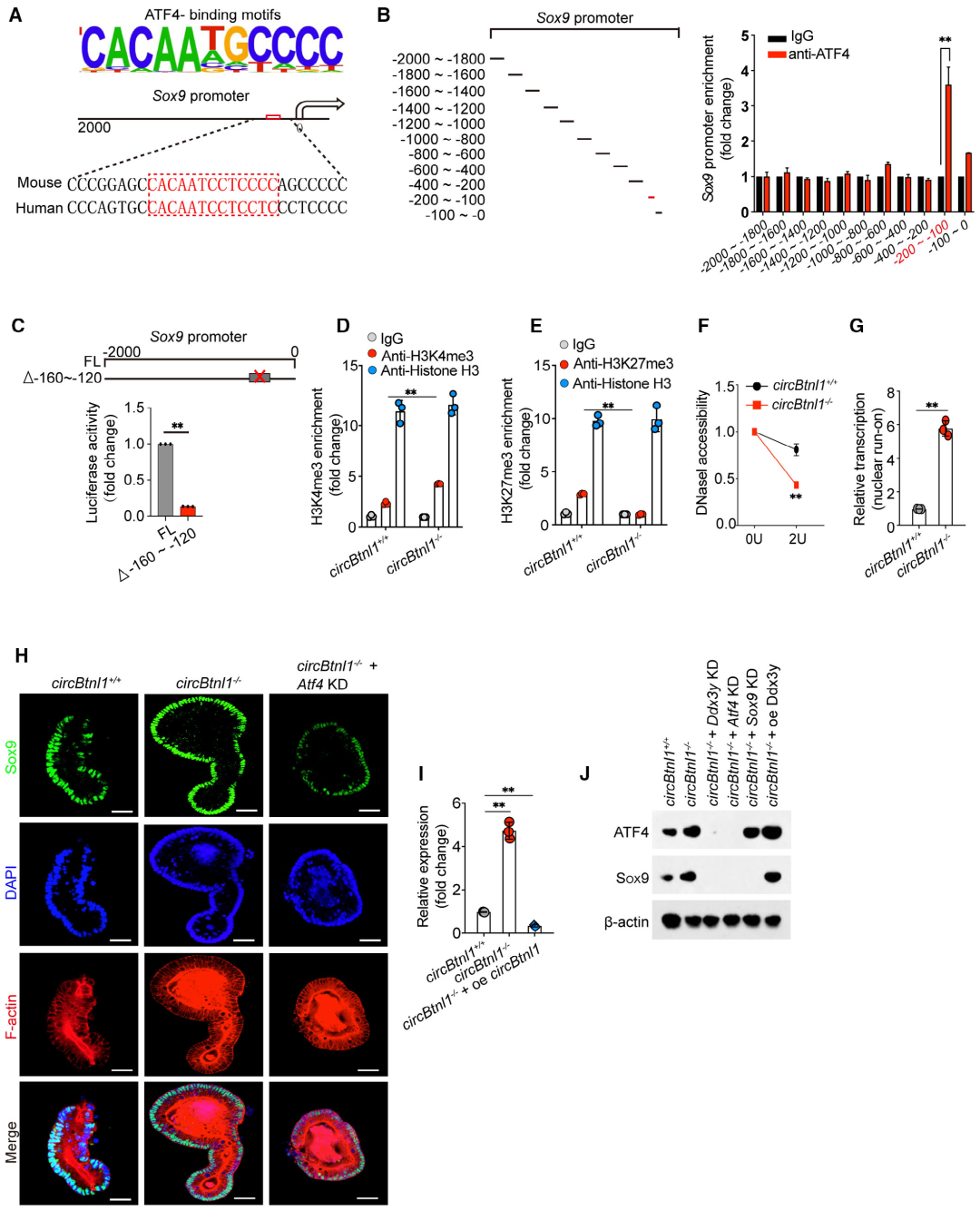
图5. CircBtnl1敲除可促进ATF4进入Sox9启动子,触发其转录
为了进一步验证Sox9在ISCs自我更新维持中的生理作用,作者通过CRISPR/Cas9技术构建了Sox9缺陷小鼠(circBtnl1+/+;sgSox9和Circbtnl1-/-;sgSox9)。在circBtnl1+/+中,sgSox9显著降低,同时ISC标记物Olfm4也随之降低。如预期的那样,Sox9缺失导致circBtnl1+/+小鼠小肠和结肠中Ki67+ISC亚群数量显著减少。此外,在circBtnl1+/+和Circbtnl1-/-小鼠中,Sox9缺失导致小肠组织中隐窝数量减少。作者随后检测了Sox9缺失小鼠的肠道再生情况,发现在辐射诱导肠上皮损伤后,circBtnl1+/+和Circbtnl1-/-小鼠中,Sox9缺失导致肠再生受损。与此一致,作者发现circBtnl1+/+和circBtnl1-/-类器官中Sox9的缺失显著抑制类器官的形成。这些数据表明Sox9参与circBtnl1介导的ISCs自我更新。
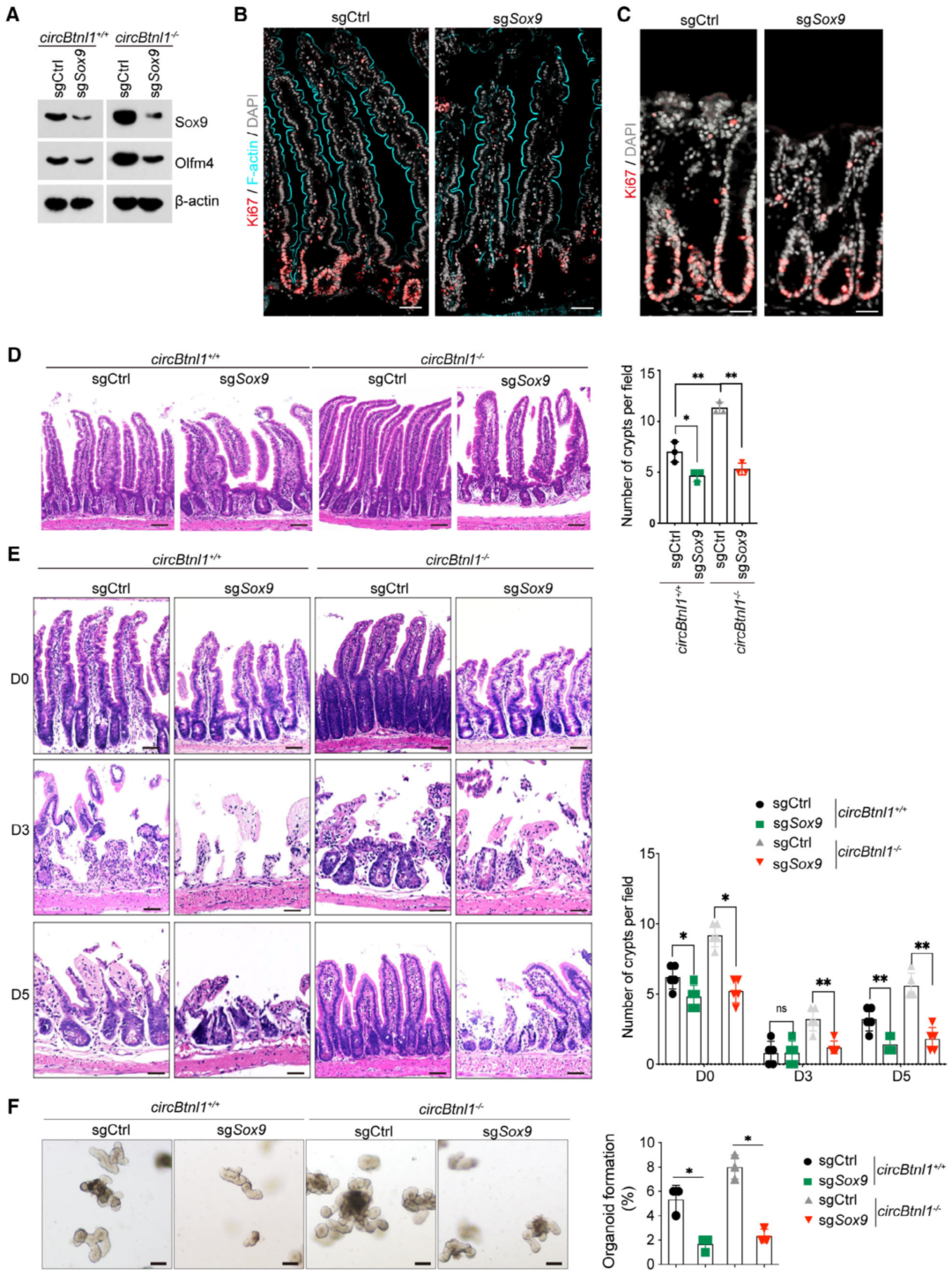
图6. Sox9基因敲除会损害ISCs的自我更新
原文链接:10.15252/embj.2022112039
参考文献:
[1] Yan KS, Janda CY, Chang J, Zheng GXY, Larkin KA, Luca VC, Chia LA, Mah AT, Han A, Terry JM et al (2017) Non-equivalence of Wnt and R-spondin ligands during Lgr5(+) intestinal stem-cell self-renewal. Nature 545:238–242
[2] Vo JN, Cieslik M, Zhang Y, Shukla S, Xiao L, Zhang Y, Wu YM, Dhanasekaran SM, Engelke CG, Cao X et al (2019) The landscape of circular RNA in cancer. Cell 176: 869–881.e13
转载请联系邮箱授权:circRNA@163.com





.png)