陈玲玲教授简介
2009年毕业于美国康涅狄格大学医学院获生物医学博士学位,同时获商学院工商管理学硕士学位。同年5月作为独立PI获得Connecticut Stem Cell Seed Award研究经费资助,受聘于康涅狄格大学干细胞学院从事博士后研究,并于2010年5月任助理教授。2011年1月任上海生化与细胞所研究员。目前担任国际期刊Trends in genetics、Genome biology、Journal of Biological Chemistry编委。2017年入选为霍华·德休斯医学院研究所(HHMI)国际研究员、入选国家杰出青年科学基金,获谈家桢生命科学创新奖;2016年入选全球华人生物学家协会CBIS杰出青年研究员奖、中国青年女科学家奖等荣誉。

刘楚霄 博士(中国科学院上海生命科学研究院) 陈玲玲教授课题组
报告主题:环形RNA调控天然免疫炎症反应

嘉宾简介:
刘楚霄博士在中国科学院上海生物化学与细胞生物学研究所攻读博士学位期间,发现了环形RNA在病毒感染时的表达变化以及在抗病毒免疫方面的功能,首次揭示环形RNA可能参与天然免疫,工作以共同第一作者身份发表于国际著名学术期刊Molecular Cell上。其后续工作发现靶向底物RNA上的m6A修饰可以抑制A-to-I RNA编辑的发生,工作以共同第一作者身份发表于国际著名学术期刊Molecular Cell上。在陈玲玲教授课题组开展博士后工作期间,解析了几十个环形RNA的二级结构,揭示环形RNAs在天然免疫过程中抑制PKR的激活并与SLE密切关联,为今后研究深入环形RNA的功能和自身免疫性疾病提供了崭新方向,相关工作以第一作者身份发表于国际著名学术期刊Cell。
在“Structure and degradation of circular RNAs regulate PKR activation in innate immunity”的研究中,科研人员首次发现环形RNA在细胞受病毒感染时被核糖核酸酶RNase L降解的过程,并解析了环形RNA形成16-26 bp的双链RNA茎环结构,并以此为基础结合天然免疫因子PKR的特性。深入研究发现,在正常细胞状态下,环形RNA通过结合PKR并抑制其活性,避免了PKR过度激活引起免疫反应;而当细胞被病毒感染时,环形RNA被RNase L快速降解进而释放PKR参与细胞的参与天然免疫炎症反应。进一步通过对系统性红斑狼疮病人来源的外周血单核细胞分析表明,在病人体内环形RNA普遍低表达且PKR异常激活;而增加环形RNA则可以显著抑制病人来源外周血单核细胞和T细胞中的PKR及其下游免疫信号通路的过度激活。这些发现首次揭示了环形RNA的降解途径及其特殊二级结构特征,并发现环形RNA发挥天然免疫炎症反应调控的全新功能。相关研究进展为环形RNA代谢和功能研究奠定了重要基础,也为炎症性自身免疫病系统性红斑狼疮的发病机制提出了环形RNA参与的新型机制。
代表性成果介绍:
Cell: circRNA调节先天免疫中PKR激活
2019年4月25日,Cell杂志发表了一篇circRNA的重磅文章,介绍发现circRNA参与细胞抗病毒免疫效应机制,该机制在系统性红斑狼疮等自身免疫疾病中发挥特殊作用。文章的通讯作者是陈玲玲教授,杨力教授和上海交通大学医学院附属仁济医院沈南教授。刘楚霄博士是本文的共同第一作者。
文中作者发现许多circRNA倾向于形成16-26bp不等的小颈环结构,该结构可结合并抑制双链RNA依赖性蛋白激酶R(double-stranded RNA-activated protein kinase,PKR),当细胞受到poly(I:C)刺激处理或病毒感染时,核酸内切酶RNase L激活并降解circRNA分子,PKR被释放并激活下游抗病毒效应机制。系统性红斑狼疮(Systematic lupus erythematosus,SLE)患者外周血单个核细胞(peripheral blood mononuclear cells,PBMC)中circRNA总体降低,PKR活性紊乱,但人为过表达circRNA有助于降低SLE患者PBMC中PKR的活性,将来或许有助于SLE等自身免疫疾病的治疗。(参考文献[1])
推荐阅读:重磅! Cell杂志再发circRNA研究论文

circRNA参与天然免疫调控 (参考文献[1])
Molecular Cell:RNA的m6A修饰负调控A-to-I修饰作用
2018年1月4日,Molecular Cell杂志在线发表了陈玲玲教授和杨力教授为共同通讯作者的文章,介绍发现RNA的m6A修饰会负调控A-to-I修饰作用。刘楚霄博士是本文的共同第一作者。(参考文献[5])
本文作者在探索m6A和A-to-I两种常见的RNA修饰方式的过程中意外发现m6A修饰较高的RNA往往对应于较少的A-to-I修饰,因此针对该发现展开了相关机制的探索研究。测序结果表明m6A和A-to-I富集的RNA区域存在明显差异,实验证明了m6A修饰与A-to-I修饰存在负相关的关系,m6A修饰越高的RNA越不容易进行A-to-I修饰。相反,针对m6A修饰酶进行抑制后能促进A-to-I修饰,可能的机制是促进相关RNA分子与ADAR1酶的结合。(参考文献[5])
推荐阅读:Molecular Cell杂志发表陈玲玲教授和杨力教授新作

(参考文献[5])
Molecular Cell:病毒感染相关因子调控环形RNA形成
2017年6月15日,Cell子刊Molecular Cell杂志在线发表了一篇环形RNA的研究论文,报道发现病毒感染相关的因子NF90/NF110参与环形RNA的形成过程。陈玲玲教授和杨力教授作为共同通讯作者的文章。刘楚霄博士是本文的共同第一作者。(参考文献[7])
本文中,作者利用全基因的RNA干扰文库(ON-TARGETplus)筛选体系在所构建的RNA环化报告系统中筛选调控circRNA生成有关的基因。作者构建了Dox诱导表达的含IRES的反向拼接报告系统,如果实现了正确的RNA拼接过程,中间部分会因为反向互补的内含子序列而被环化,形成完整的mCherry荧光蛋白,可检测到红光,其余的RNA正确拼接后会形成GFP蛋白。而如果仅仅是常规的拼接活动,没有中间的环化过程,则会因为GFP中间插入了核酸序列而无法形成正确的荧光蛋白。该系统可基于荧光检测实现高通量快速筛选目标蛋白。结果发现NF90/NF110通过稳定内含子互补序列结构促进环形RNA的形成。病毒感染的条件下,诱导NF90/NF110出核并抑制病毒复制,环形RNA生成量也随之降低。(参考文献[7])
推荐阅读:陈玲玲教授和杨力教授的最新力作:病毒感染相关因子调控环形RNA形成
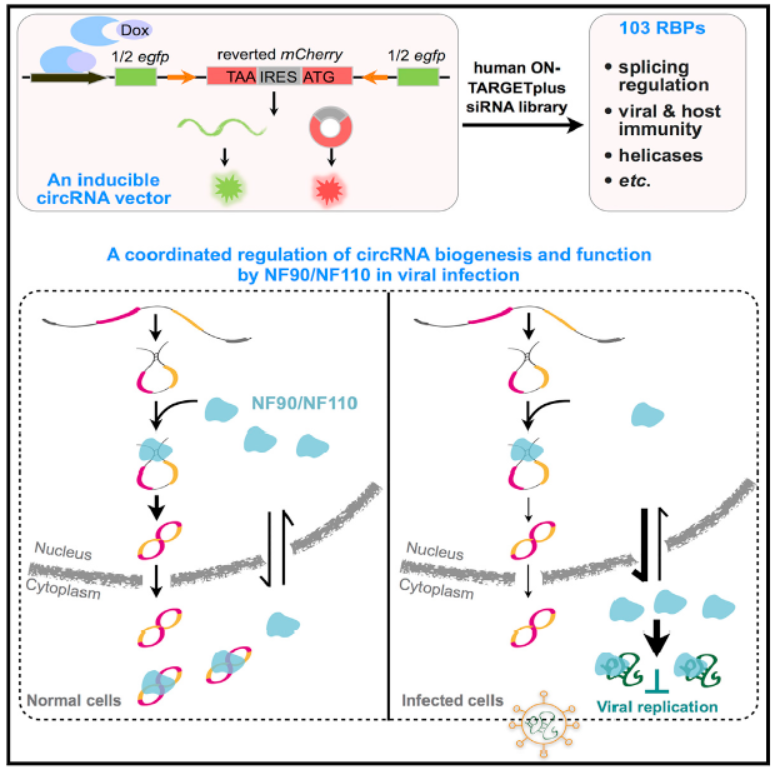
NF90/NF110参与环形RNA的形成过程(参考文献[7])
Cell:SLERT 通过影响DDX21环状结构调控RNA聚合酶I的转录活动
2017年5月4日,Cell 杂志在线发表了中科院上海生化细胞所陈玲玲教授团队最新的研究论文,介绍一类非编码RNA SLERT 通过影响DDX21环状结构调控RNA聚合酶I的转录活动。(参考文献[9])
SLERT长694nt,两端分别为box H/ACA 家族的snoRNA:SNORA5A 和SNORA5C,SLERT生成和核仁定位需要这两个snoRNA。干扰SLERT可降低pre-rRNA转录和rRNA生成,降低肿瘤发生。SLERT通过143-nt非snoRNA序列与DEAD-box RNA解旋酶DDX21相互作用。超分辨率图像显示DDX21形成环绕多个Pol I复合物的环状结构并抑制pre-rRNA转录。SLERT通过结合DDX21影响后者的分子内构象,降低与RNA聚合酶I的结合能力,释放RNA聚合酶I,促进rRNA的转录。
推荐阅读:Cell杂志在线发表陈玲玲教授最新lncRNA研究文章

SLERT 通过影响DDX21环状结构调控RNA聚合酶I的转录活动(参考文献[9])
Molecular Cell:circRNA生成机制与功能研究综述
2018年8月2日,Molecular Cell杂志在线发表了陈玲玲教授和杨力教授为通讯作者的重要circRNA综述,系统汇总了circRNA生成机制,功能模型等方面的研究进展,也剖析了目前circRNA研究所面临的问题。(参考文献[4])
推荐阅读:杨力、陈玲玲教授在Molecular Cell杂志发表长篇circRNA综述

Genome Research:circRNA存在可变剪切
2016年9月,Genome Research杂志发表了陈玲玲教授与杨力教授为共同通讯作者的文章,报道发现circRNA也存在可变剪切。与本文几乎同步,赵方庆教授在Nature Communications杂志的文章也报道发现了circRNA的可变剪接作用。这两篇文章是最早发现和探索circRNA可变剪接的研究论文。
本文中,作者发现四种理论上的可变剪切方式均存在。5’或3’选择近端或远端拼接位点会因细胞类型不同而有巨大差异,这说明环状RNA的形成与可变剪切方式与细胞类型关系非常密切。作者也通过信息分析结合体外表达验证的思路针对环状RNA环化位点周围的反向互补序列对可变剪切的影响进行了分析,结果表明反向互补序列的分布的确能影响环状RNA的可变剪切形式。作者在分析环状RNA可变剪切序列所对应的外显子序列中,发现了数千种环状RNA独有的外显子!它们无法直接与已注释的线性RNA中存在的外显子序列匹配,也不是完整的内含子序列,而是新形式的外显子!这类外显子的保守性明显低于已注释的外显子,在不同细胞中呈现高度多样性,个别的能在多种细胞中同时检测到。这说明RNA可变剪切方面还有很多未知的机制需要进一步的研究。(参考文献[16])
推荐阅读:环状RNA的可变剪接研究又一新作

可变的Back-splice剪切与经典RNA可变剪切类型模型(参考文献[16])
Cell Reports:环状RNA形成的动态过程
2016年4月19日,陈玲玲教授和杨力教授作为共同通讯作者在Cell Reports杂志发表了题为“The Biogenesis of Nascent Circular RNAs”的重要文章,介绍了利用4sUDRB脉冲标记后测序的技术检测环状RNA形成的动态过程,探索了环状RNA形成早期的基本规律。(参考文献[17])
在该文中,作者用了一种基于标记的测序技术,专门富集新转录形成的RNA进行分析。该技术称为4sUDRB sequencing (4sUDRB-seq),概括而言,就是首先用DRB(5,6-dichloro-1–d-ribofuranosylbenzimidazole,5,6-二氯-1–D-呋喃核糖苯并咪唑)处理细胞,抑制RNA聚合酶II(PA1细胞中处理3个小时即可),去掉DRB的同时加上4sU,这样新生成的RNA中即掺入4sU,然后通过HPDP-biotin(N-[6-(Biotinamido)hexyl]-3’-(2’- pyridyldithio)propionamide)纯化。为防止HPDP-biotin干扰或影响4sU标记环状RNA形成过程和纯化得率,作者通过体外过表达体系进行了验证分析,表明该套系统不会影响环状RNA的形成,也不会降低对4sU标记环状RNA回收效率。
利用该体系,作者发现环状RNA的形成速率其实是非常低的,形成过程与聚合酶II的快速延伸有关。以神经元为例,环状RNA的富集依赖于其中的快速转录过程和长时间积累效应。本文首次对环状RNA形成过程进行探索研究,表明环状RNA的形成是快速转录活动,其中的顺式元件(RNA内部的反向互补序列)以及细胞内长时间积累效应共同决定的。(参考文献[17])

(参考文献[17])
Cell:内含子中反向互补序列促进环状RNA形成
2014年9月25日,Cell杂志发表了陈玲玲教授和杨力教授为共同通讯作者的文章,报道发现内含子中反向互补序列介导circRNA的形成。(参考文献[27])
本文在分析人胚胎干细胞系H9的poly(A)–/Ribo–及RNase R消化的poly(A)–/Ribo– 的RNA样品RNA-Seq数据得到了一大批反向拼接的RNA产物,最终得到了1662种环状RNA,其中有12%的环状RNA为H9细胞中独有的。首次提出并通过实验验证了内含子区存在的反向互补RNA序列是驱动形成环状RNA的重要原因。反向Alu元件(IRAlu)已成为预测和分析环状RNA形成机制的重要依据(参考文献[27])。该文献是环状RNA研究领域中的重量级成果之一,所提出的环状RNA形成机制模型获得了学术界的高度认可。
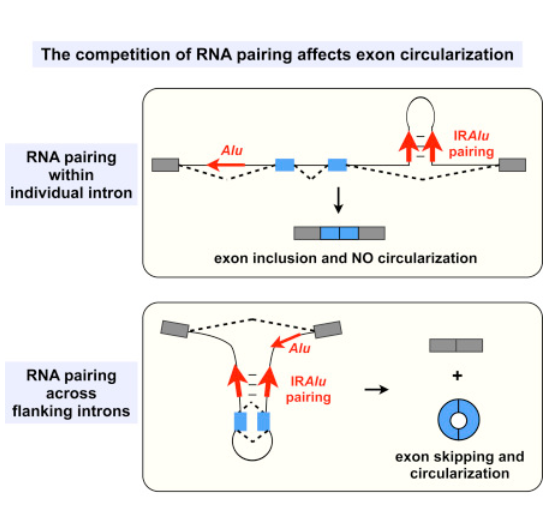
内含子反向互补序列介导circRNA形成(参考文献[27])
Molecular Cell:内含子来源ciRNA调控基因转录
2013年9月26日,Molecular Cell杂志发表了陈玲玲教授和杨力教授为共同通讯作者的文章,发现了一类由内含子来源的环状RNA(ciRNA)具有调控基因转录的功能。(参考文献[33])
该研究发现了环状RNA的一种重要功能:参与调控基因转录作用过程。研究者探索了内含子形成的环状RNA(ciRNA)的功能,发现大部分ciRNA定位于细胞核内,几乎没有miRNA结合的功能,但有趣的是将ciRNA敲低,却会阻碍对应的基因转录的过程。研究选取ci-ankrd52为研究对象,发现该ciRNA会大量富集于该基因的转录起始区,促进了RNA聚合酶II的功能。初步证明了带有内含子序列的环状RNA的功能是促进所对应的基因转录的活动。本文是环状RNA功能研究的重要成果,在早期探索circRNA功能中是除了miRNA Sponge之外的环状RNA最重要的功能模型。(参考文献[33])
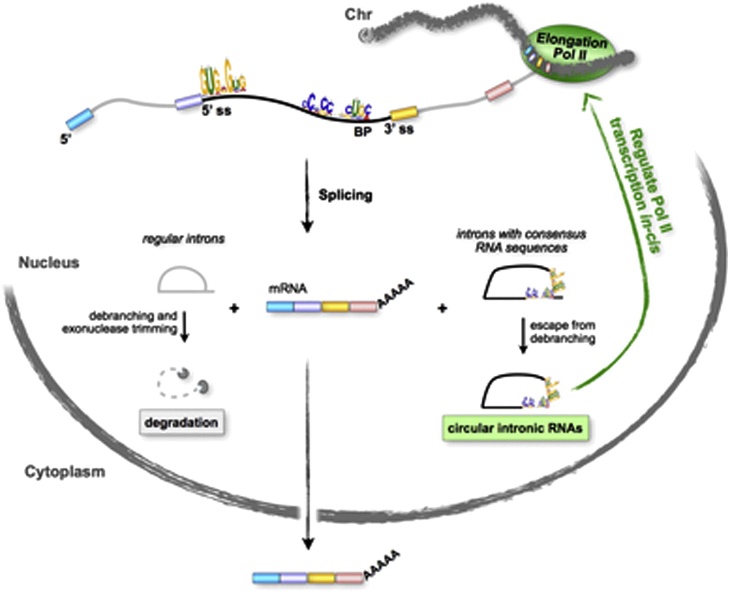
内含子来源ciRNA调控基因转录(参考文献[33])
陈玲玲教授和杨力教授是国内最早开展circRNA研究的科研团队之一,有一系列重要的circRNA原创性研究工作,除了上述列举的研究,还有一些非常有价值的研究工作,包括发现circRNA来源的假基因,发现circRNA随着进化而逐渐增多(参考文献[10])等等。感兴趣的可以去他们实验室主页查找相关文献。
陈玲玲教授实验室主页:http://www.sibcb.ac.cn/PI.asp?id=138
已发表文章:
1. Liu CX#, Li X#, Nan F#, Jiang S#, Gao X, Guo SK, Xue W, Cui YG, Dong KG, Ding HH, Qu B, Shen N*, Yang L*, Chen LL*. 2019. Structure and degradation of circular RNAs regulate PKR activation in innate immunity. Cell.
2. Yao RW, Wang Y and Chen LL*. 2019. Cellular functions of long noncoding RNAs. Nat Cell Biol, 21:542–551.
3. Wang Y#, Hu SB#, Wang MR#, Yao RW, Wu D, Yang L and Chen LL*. 2018. Genome-wide screening of NEAT1 regulators reveals cross-regulation between paraspeckles and mitochondria. Nat Cell Biol, 20:1145-1158.
4. Li X, Yang L* and Chen LL*. 2018. The biogenesis, functions and challenges of circular RNAs. Mol Cell, 71:428-442.
5. Xiang JF#, Yang Q#, Liu CX#, Wu M, Chen LL* and Yang L*. 2018. N6-methyladenosines modulate A-to-I RNA editing. Mol Cell, 69:126-135.
6. Yang L* and Chen LL*. 2017. Enhancing the RNA engineering tool kit. Science, 358: 996-997.
7. Li X#, Liu CX#, Xue W#, Zhang Y, Jiang S, Yin QF, Wei J, Yao RW, Yang L* and Chen LL*. 2017. Coordinated circRNA biogenesis and function with NF90/NF110 in viral infection. Mol Cell, 67: 214-222.
8. Wu H, Yang L*, Chen LL*. 2017. The diversity of long noncoding RNAs and their generation. Trends Genet, 33:540-552.
9. Xing YH#, Yao RW#, Zhang Y#, Guo CJ, Jiang S, Xu G, Dong R, Yang L, Chen LL*. 2017. SLERT regulates DDX21-rings associated with Pol I transcription. Cell, 169: 664-678.
10. Dong R#, Ma XK#, Chen LL* and Yang L*. 2017. Increased complexity of circRNA expression during species evolution. RNA Biol, 14:1064-1074.
11. Chen LL* and Yang L*. 2017. AlUternative regulation for gene expression. Trends Cell Biol, 27:480-490.
12. Wu H#, Yin QF#, Luo Z#, Yao RW, Zheng CC, Zhang J, Xiang JF, Yang L and Chen LL*. 2016. Unusual processing generates SPA lncRNAs that sequester multiple RNA binding proteins. Mol Cell, 64:435-437.
13. Xing YH#, Bai ZQ#, Liu CX, Hu SB, Ruan MH and Chen LL*. 2016. Research progress of long noncoding RNA in China. IUBMB Life, 68:887-893.
14. Hu SB, Yao RW and Chen LL*. 2016. Shedding light on paraspeckle structure by super-resolution microscopy. J Cell Biol, 214:789-791.
15. Chen LL*. 2016. Linking long noncoding RNA localization and function. Trends Biochem Sci, 41:761-772.
16. Zhang XO#, Dong R#, Zhang Y#, Zhang JL, Luo Z, Zhang J, Chen LL*and Yang L*. 2016. Diverse alternative back-splicing and alternative splicing landscape of circular RNAs. Genome Res, 26: 1277-1287.
17. Zhang Y#, Xue W#, Li X, Zhang J, Zhang JL, Yang L* and Chen LL*. 2016. The biogenesis of nascent circular RNAs. Cell Rep, 15:611-624.
18. Chen LL*. 2016. The biogenesis and emerging roles of circular RNAs. Nat Rev Mol Cell Biol, 17:205-211.
19. Zhang Y, Yang L and Chen LL*. 2016. Characterization of circular RNAs. Methods Mol Biol, 1402:215-27.
20. Xiang JF, Yang L and Chen LL*. 2015. The long noncoding RNA regulation at the MYC locus. Curr Opin Genet Dev, 33:41-48.
21. Chen LL* and Yang L*. 2015. Gear up in circles. Mol Cell, 58:715-717.
22. Chen LL* and Yang L*. 2015. Regulation of circRNA biogenesis. RNA Biol, 12:381-388.
23. Hu SB, Xiang JF, Li X, Xu YF, Xue W, Huang M, Wong CC, Sagum CA, Bedford MT, Yang L, Cheng D* and Chen LL*. 2015. Protein arginine methyltransferase CARM1 attenuates the nuclear retention of mRNAs containing IRAlus at paraspeckles. Genes Dev, 29: 630-645.
24. Chen T#, Xiang JF#, Zhu S#, Chen S, Yin QF, Zhang XO, Zhang J, Feng H, Dong R, Li XJ, Yang L* and Chen LL*. 2015. ADAR1 is required for differentiation and neural induction by regulating microRNA processing in a catalytically independent manner. Cell Res, 25:459-476.
25. Yin QF#, Hu SB#, Xu YF, Yang L, Carmichael GG and Chen LL*. 2015. SnoVectors for nuclear expression of RNA. Nucleic Acids Res, 43(1):e5.
26. Yang L* and Chen LL*. 2014. Microexons go big. Cell, 159:1488-1489.
27. Zhang XO#, Wang HB#, Zhang Y, Lu X, Chen LL* and Yang L*. 2014. Complementary sequence- mediated exon circularization. Cell, 159:134-147.
28. Zhang Y, Yang L and Chen LL*. 2014. Life without A tail: new formats of long noncoding RNAs. Int J Biochem Cell Biol, 54:338-349.
29. Zhang XO #, Yin QF #, Wang HB, Zhang Y, Chen T, Zheng P, Lu X, Chen LL* and Yang L*. 2014. Species-specific alternative splicing leads to unique expression of sno-lncRNAs. BMC Genomics, 15:287.
30. Xiang JF, Yin QF, Chen T, Zhang Y, Zhang XO, Wang HB, Ge JH, Lu XH, Yang L and Chen LL*. 2014. Human colorectal cancer-specific CCAT1-L lncRNA regulates long-range chromatin interactions in the MYC locus. Cell Res, 24:513-531.
31. Chen LL* and Zhao JC*. 2014. Functional study of long noncoding RNAs in development and diseases. Adv Exp Med Biol, 825:129-158.
32. Zhu S#, Xiang JF#, Chen T, Chen LL* and Yang L*. 2013. Prediction of constitutive A-to-I editing sites from human transcriptomes in the absence of genomic sequences. BMC Genomics, 14:206.
33. Zhang Y#, Zhang XO#, Chen T, Xiang JF, Yin QF, Xing YH, Zhu S, Yang L* and Chen LL*. 2013. Circular intronic long noncoding RNAs, Mol Cell, 51: 792-806.
34. Yin QF#, Yang L#, Zhang Y, Xiang JF, Wu YW, Carmichael GG* and Chen LL*. 2012. Long noncoding RNAs with snoRNA ends. Mol Cell, 48: 219-230.
35. Yang L, Duff MO, Graveley BR, Carmichael GG and Chen LL*. 2011. Genomewide characterization of non- polyadenylated RNAs. Genome Biol, 12: R16.
36. Chen LL* and Carmichael GG. 2010. Long non-coding RNAs in mammalian cells: What, Where and Why? WIREs RNA, 1: 2-21.
37. Chen LL* and Carmichael GG*. 2010. Decoding the function of nuclear long non-coding RNAs. Curr Opin Cell Biol, 22: 357-364.
38. Chen LL*, Yang L and Carmichael GG*. 2010. Molecular basis for an attenuated cytoplasmic dsRNA response in human embryonic stem cells. Cell Cycle, 9:3552-3564.
39. Chen LL and Carmichael GG. 2009. Altered nuclear retention of mRNAs containing inverted repeats in human embryonic stem cells: functional role of a nuclear noncoding RNA. Mol Cell, 35: 467-478.
40. Chen LL and Carmichael GG. 2008. Gene regulation by SINES and inosines: biological consequences of A-to-I editing of Alu element inverted repeats. Cell Cycle, 7: 3297-3301.
41. Chen LL, Decerbo JN and Carmichael GG. 2008. Alu element-mediated gene silencing. EMBO J, 27: 1694-1705.
.png)