概述
 OncoRNALab由比利时根特大学的Jo Vandesompele和Pieter Mestdagh教授负责监管,属于根特医学遗传学中心和癌症研究所的一部分。该实验室的研究旨在利用RNA进行诊断和治疗,目前大量研究集中于非编码RNA在癌症中的作用以及细胞外RNA在液体活检中的应用。该实验室试图通过高通量测序技术与生物信息学工具的结合来回答各种基础和转化研究的问题。
OncoRNALab由比利时根特大学的Jo Vandesompele和Pieter Mestdagh教授负责监管,属于根特医学遗传学中心和癌症研究所的一部分。该实验室的研究旨在利用RNA进行诊断和治疗,目前大量研究集中于非编码RNA在癌症中的作用以及细胞外RNA在液体活检中的应用。该实验室试图通过高通量测序技术与生物信息学工具的结合来回答各种基础和转化研究的问题。

这篇文章很短,但是故事很长。
文章很短,文章只做了一件环状RNA研究者都会做的事:对高通量测序检测到的环状RNA进行低通量验证。
故事很长,本文联合了环状RNA领域的诸多大咖一起,通过3种低通量检测方法对16个高通量circRNA检测方法的精确性和敏感性等指标进行了评估。而这16个工具,来自不同的时间、不同的研究小组,见证了环状RNA领域的发展史;每一个工具的背后应该都有一段故事——叙说了开发者对环状RNA的认知和期许。
文章对3种人类癌症细胞系的total RNA进行了深度测序,通过16种知名的检测工具识别到了315312个环状RNA,随后通过三种正交的低通量验证方法:
qPCR
RNase R
amplicon sequencing
评估过程与部分结果
一些环状RNA引物存在脱靶风险,将不被考虑用于验证,剩余1560个分子。
同时由于偶然原因,不同的工具,不同的细胞系,相同的circRNA被选中,最后1516个circRNAs被用于最后的验证。
 每个工具具体的环状RNA精确性(验证率)如下图
每个工具具体的环状RNA精确性(验证率)如下图
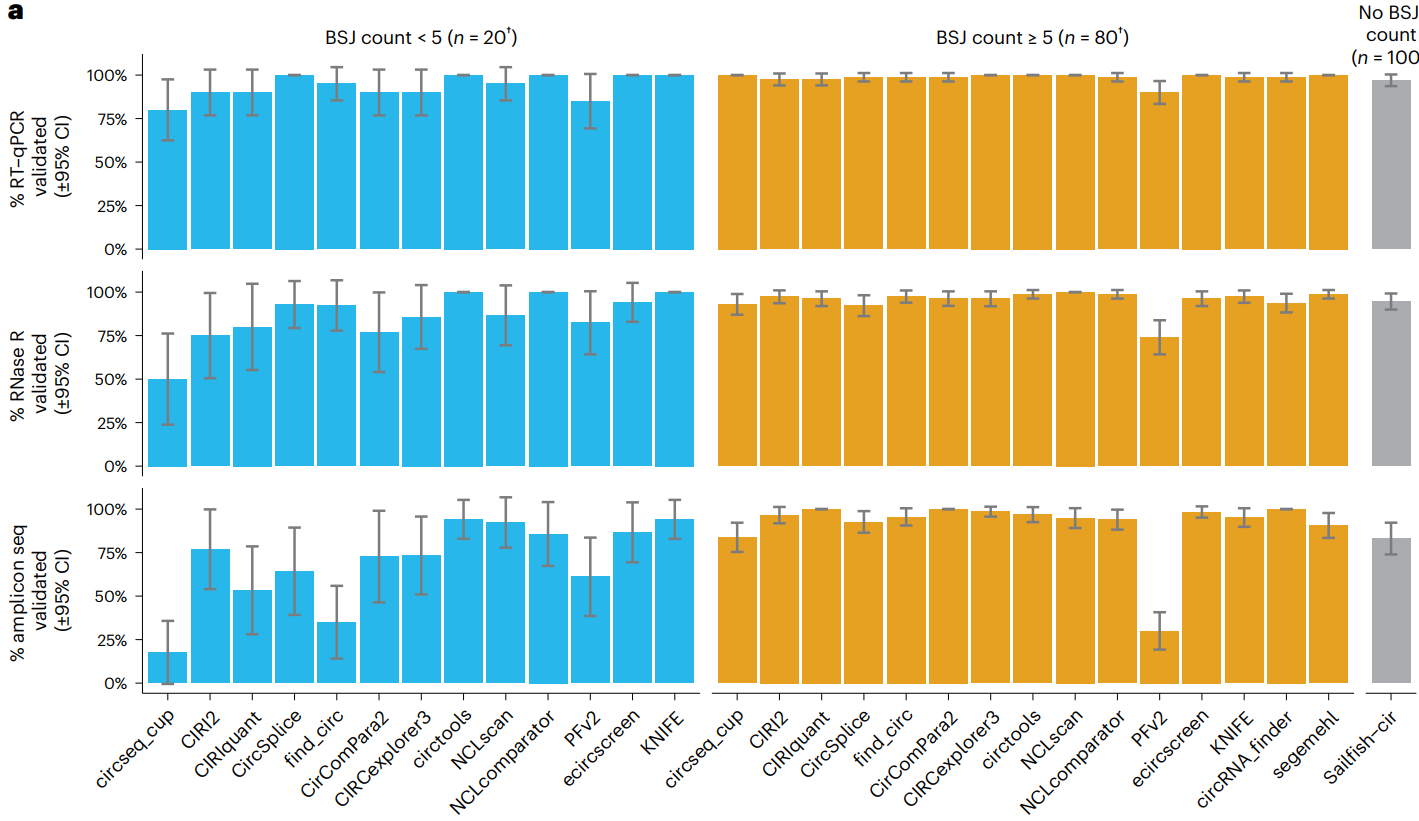
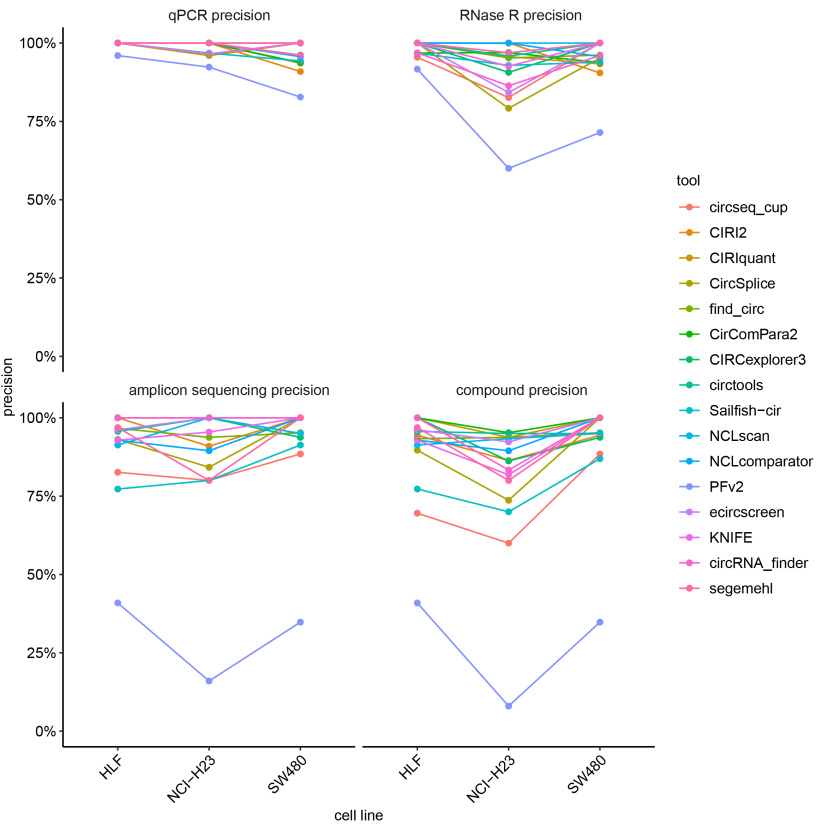
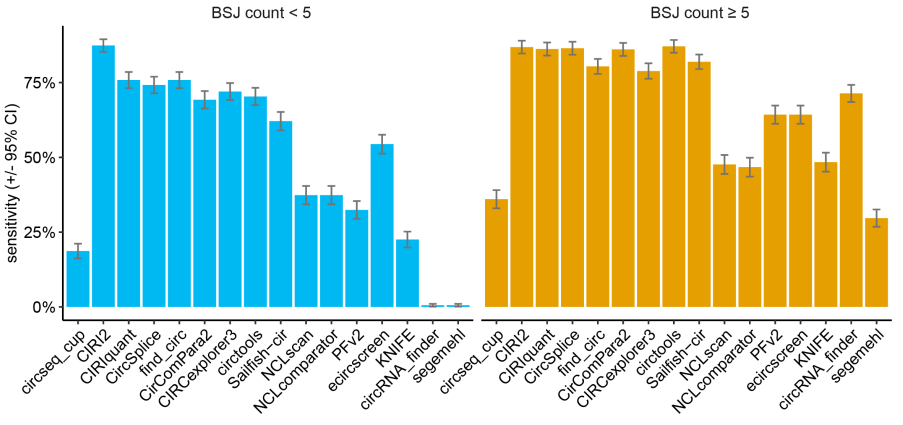
敏感性评估如下图

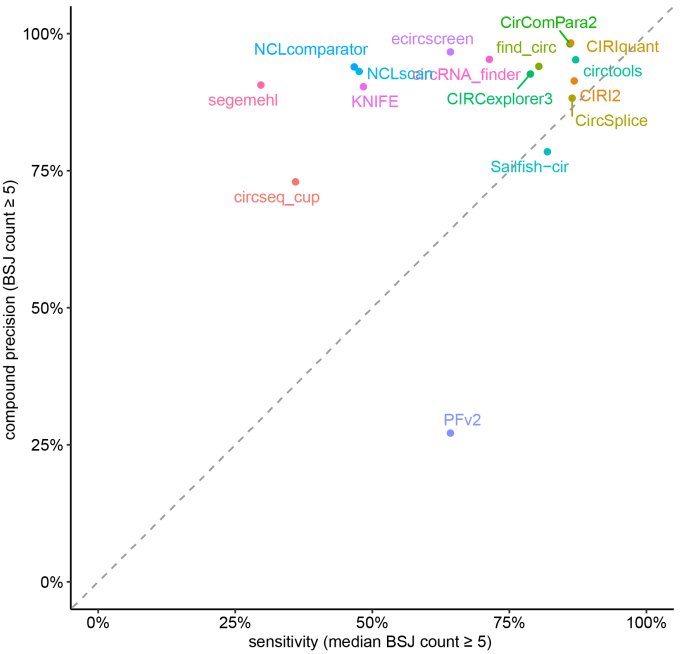
敏感性和精确性的综合评估
 更详细的结果请访问原文:
更详细的结果请访问原文:小知识
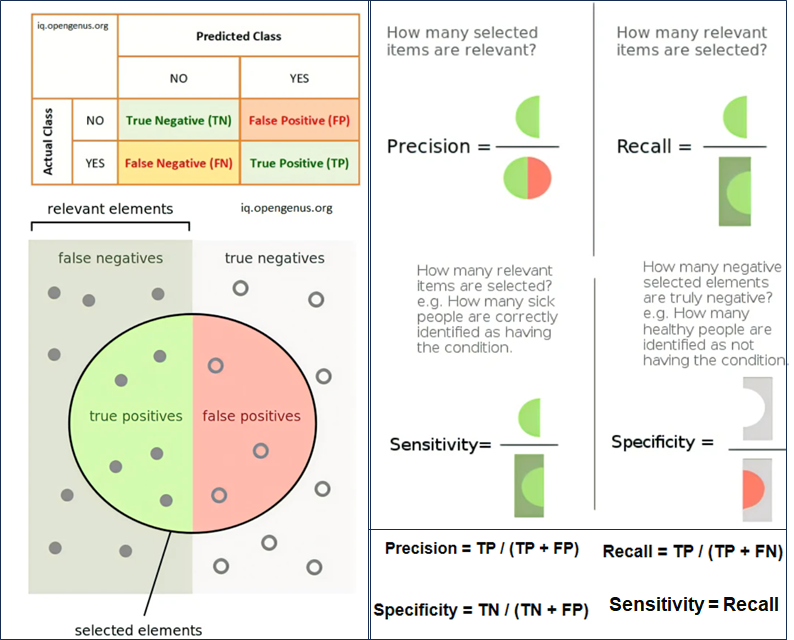 中肯的建议
中肯的建议
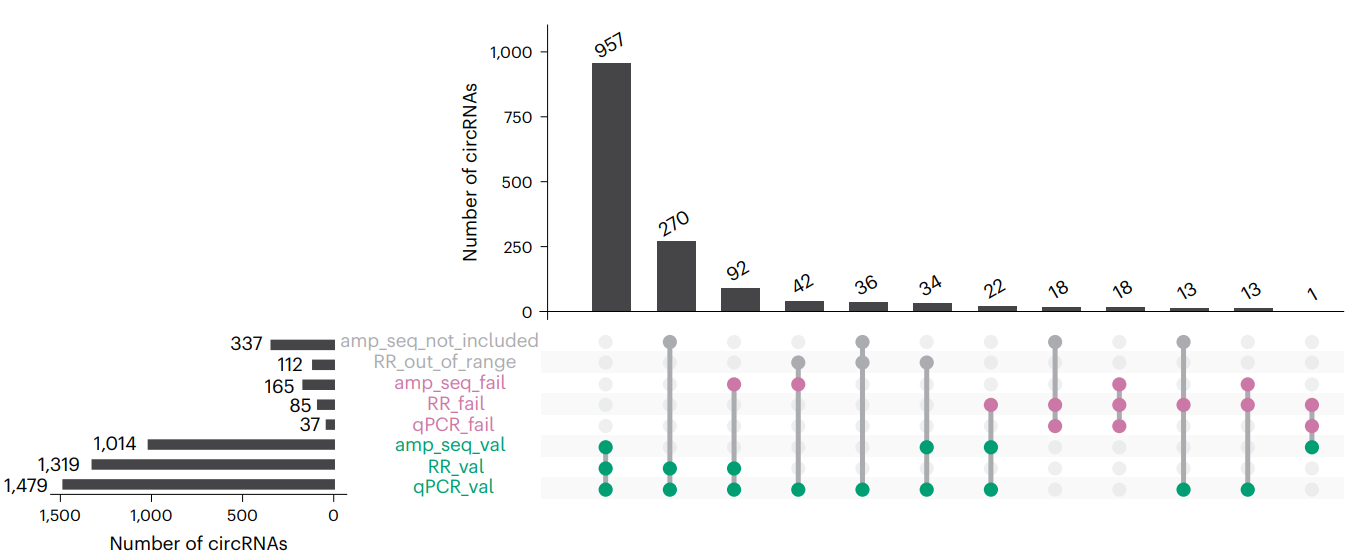 因此,作者表明
因此,作者表明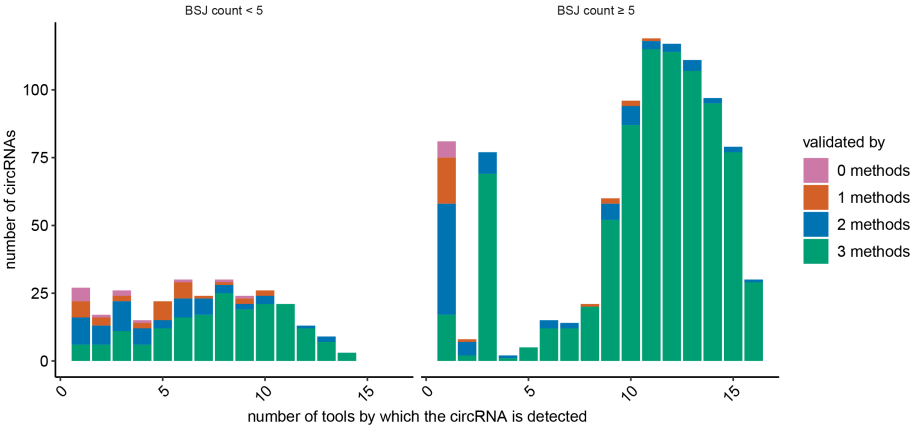 表格链接:
表格链接:
 同时,文章中检测以及验证的环状RNA都可以在github上获取到,地址为:
同时,文章中检测以及验证的环状RNA都可以在github上获取到,地址为:
• 东南大学,温国霞
• 浙江大学,叶楚玉
• 复旦大学,杨力
• 上海中科院,袁国华
• 北京中科院,赵方庆
• 台湾中央研究院,莊樹諄

.png)